Escriba el proceso de transcripción y traducción procariótica
Los procariotas, que incluyen bacterias y arqueas, son en su mayoría organismos unicelulares que, por definición, carecen de núcleos unidos a membranas y otros orgánulos. Un cromosoma bacteriano es un círculo cerrado covalentemente que, a diferencia de los cromosomas eucariotas, no está organizado en torno a proteínas histónicas. La región central de la célula en la que reside el ADN procariota se denomina nucleoide. Además, los procariotas suelen tener abundantes plásmidos, que son moléculas de ADN circular más cortas que pueden contener sólo uno o unos pocos genes. Los plásmidos pueden transferirse independientemente del cromosoma bacteriano durante la división celular y a menudo portan rasgos como la resistencia a los antibióticos. Debido a estas características únicas, la transcripción y la regulación de los genes es algo diferente entre las células procariotas y las eucariotas.
Objetivos de aprendizaje
- Comprender los pasos básicos de la transcripción del ADN en ARN en las células procariotas
- Comprender los fundamentos de la traducción procariótica y en qué se diferencia de la traducción eucariota
Transcripción procariota
Inicio de la transcripción en procariotas
Los procariotas no tienen núcleosmembranas. Por lo tanto, los procesos de transcripción, traducción y degradación del ARNm pueden ocurrir simultáneamente. El nivel intracelular de una proteína bacteriana puede ampliarse rápidamente mediante múltiples eventos de transcripción y traducción que ocurren simultáneamente en la misma plantilla de ADN. La transcripción procariótica a menudo abarca más de un gen y produce ARNm policistrónicos que especifican más de una proteína.
Nuestra discusión aquí ejemplificará la transcripción describiendo este proceso en Escherichia coli, una especie bacteriana bien estudiada. Aunque existen algunas diferencias entre la transcripción en E. coli y la transcripción en las arqueas, la comprensión de la transcripción en E. coli puede aplicarse prácticamente a todas las especies bacterianas.
La ARN polimerasa procariótica
Los procariotas utilizan la misma ARN polimerasa para transcribir todos sus genes. En E. coli, la polimerasa está compuesta por cinco subunidades polipeptídicas, dos de las cuales son idénticas. Cuatro de estas subunidades, denominadas α, α, β y β′, constituyen el núcleo de la polimerasa. Estas subunidades se ensamblan cada vez que se transcribe un gen y se desensamblan una vez completada la transcripción. Cada subunidad tiene una función única; las dos subunidades α son necesarias para ensamblar la polimerasa en el ADN; la subunidad β se une al ribonucleósido trifosfato que formará parte de la molécula naciente de ARNm «recién nacida»; y la β′ se une a la cadena molde del ADN. La quinta subunidad, la σ, participa únicamente en el inicio de la transcripción. Confiere especificidad transcripcional de manera que la polimerasa comienza a sintetizar ARNm a partir de un sitio de iniciación apropiado. Sin σ, la enzima central transcribiría desde sitios aleatorios y produciría moléculas de ARNm que especificarían un galimatías de proteínas. La polimerasa compuesta por las cinco subunidades se denomina holoenzima (una holoenzima es un compuesto bioquímicamente activo formado por una enzima y su coenzima).
Promotores procarióticos

Figura 1. La subunidad σ de la ARN polimerasa procariota reconoce secuencias consenso que se encuentran en la región promotora aguas arriba de la vista de inicio de la transcripción. La subunidad σ se disocia de la polimerasa una vez iniciada la transcripción.
Un promotor es una secuencia de ADN a la que la maquinaria de transcripción se une e inicia la transcripción. En la mayoría de los casos, los promotores existen aguas arriba de los genes que regulan. La secuencia específica de un promotor es muy importante porque determina si el gen correspondiente se transcribe todo el tiempo, parte del tiempo o con poca frecuencia. Aunque los promotores varían entre los genomas procariotas, algunos elementos se conservan. En las regiones -10 y -35 aguas arriba del sitio de iniciación, hay dos secuencias consenso del promotor, o regiones que son similares en todos los promotores y en varias especies bacterianas (Figura 1).
La secuencia consenso -10, llamada región -10, es TATAAT. La secuencia -35, TTGACA, es reconocida y unida por σ. Una vez realizada esta interacción, las subunidades del núcleo de la enzima se unen al sitio. La región -10, rica en A-T, facilita el desenrollado de la plantilla de ADN y se producen varios enlaces fosfodiéster. La fase de iniciación de la transcripción termina con la producción de transcritos abortivos, que son polímeros de aproximadamente 10 nucleótidos que se fabrican y se liberan.
Elongación y terminación en procariotas
La fase de elongación de la transcripción comienza con la liberación de la subunidad σ de la polimerasa. La disociación de σ permite a la enzima central proceder a lo largo del molde de ADN, sintetizando ARNm en la dirección 5′ a 3′ a una velocidad de aproximadamente 40 nucleótidos por segundo. A medida que avanza la elongación, el ADN se desenrolla continuamente por delante de la enzima central y se rebobina detrás de ella (Figura 2). El emparejamiento de bases entre el ADN y el ARN no es lo suficientemente estable como para mantener la estabilidad de los componentes de la síntesis del ARNm. En su lugar, la ARN polimerasa actúa como un enlazador estable entre el molde de ADN y las hebras de ARN nacientes para garantizar que la elongación no se interrumpa prematuramente.

Figura 2. Haga clic para ampliar la imagen. Durante la elongación, la ARN polimerasa procariota se desplaza a lo largo de la plantilla de ADN, sintetiza ARNm en la dirección 5′ a 3′, y desenrolla y rebobina el ADN a medida que lo lee.
Señales de terminación procariota
Una vez que se transcribe un gen, la polimerasa procariota necesita recibir instrucciones para disociarse de la plantilla de ADN y liberar el ARNm recién hecho. Dependiendo del gen que se transcribe, hay dos tipos de señales de terminación. Una se basa en las proteínas y la otra en el ARN. La terminación dependiente de Rho está controlada por la proteína rho, que sigue a la polimerasa en la cadena de ARNm en crecimiento. Cerca del final del gen, la polimerasa encuentra una serie de nucleótidos G en la plantilla de ADN y se detiene. Como resultado, la proteína rho choca con la polimerasa. La interacción con rho libera el ARNm de la burbuja de transcripción.
La terminación independiente de rho está controlada por secuencias específicas en la cadena molde de ADN. Cuando la polimerasa se acerca al final del gen que se está transcribiendo, se encuentra con una región rica en nucleótidos C-G. El ARNm se pliega sobre sí mismo y los nucleótidos C-G complementarios se unen. El resultado es una horquilla estable que hace que la polimerasa se detenga en cuanto comienza a transcribir una región rica en nucleótidos A-T. La región complementaria U-A del transcrito del ARNm sólo forma una débil interacción con el ADN molde. Esto, junto con el estancamiento de la polimerasa, induce la suficiente inestabilidad para que la enzima central se desprenda y libere el nuevo transcrito de ARNm.
Al terminar, el proceso de transcripción está completo. En el momento en que se produce la terminación, el transcrito procariota ya habría sido utilizado para iniciar la síntesis de numerosas copias de la proteína codificada, ya que estos procesos pueden ocurrir simultáneamente. La unificación de la transcripción, la traducción e incluso la degradación del ARNm es posible porque todos estos procesos ocurren en la misma dirección 5′ a 3′, y porque no hay compartimentación membranosa en la célula procariota (Figura 3). Por el contrario, la presencia de un núcleo en las células eucariotas impide la transcripción y traducción simultáneas.

Figura 3. Múltiples polimerasas pueden transcribir un único gen bacteriano mientras numerosos ribosomas traducen simultáneamente los transcritos de ARNm en polipéptidos. De este modo, una proteína específica puede alcanzar rápidamente una alta concentración en la célula bacteriana.
Visite esta animación de BioStudio para ver el proceso de transcripción procariota.
Preguntas de práctica
¿Qué subunidad de la E. coli confiere especificidad a la transcripción?
- α
- β
- β′
- σ
Las regiones -10 y -35 de los promotores procariotas se denominan secuencias consenso porque ________.
- son idénticas en todas las especies bacterianas
- son similares en todas las especies bacterianas
- existen en todos los organismos
- tienen la misma función en todos los organismos
Traducción procariótica
La traducción es similar en procariotas y eucariotas. Aquí exploraremos cómo se produce la traducción en E. coli, un procariota representativo, y especificaremos cualquier diferencia entre la traducción bacteriana y la eucariótica.
Iniciación
La iniciación de la síntesis de proteínas comienza con la formación de un complejo de iniciación. En E. coli, este complejo incluye el pequeño ribosoma 30S, el molde de ARNm, tres factores de iniciación que ayudan al ribosoma a ensamblarse correctamente, el trifosfato de guanosina (GTP) que actúa como fuente de energía, y un ARNt iniciador especial que lleva N-formilmetionina (fMet-ARNtfMet) (Figura 4). El ARNt iniciador interactúa con el codón de inicio AUG del ARNm y lleva una metionina formilada (fMet). Debido a su implicación en la iniciación, fMet se inserta al principio (extremo N) de cada cadena polipeptídica sintetizada por E. coli. En el ARNm de E. coli, una secuencia líder aguas arriba del primer codón AUG, denominada secuencia Shine-Dalgarno (también conocida como sitio de unión ribosomal AGGAGG), interactúa mediante el emparejamiento de bases complementarias con las moléculas de ARNr que componen el ribosoma. Esta interacción ancla la subunidad ribosomal 30S en el lugar correcto de la plantilla de ARNm. En este punto, la subunidad ribosomal 50S se une entonces al complejo de iniciación, formando un ribosoma intacto.
En los eucariotas, la formación del complejo de iniciación es similar, con las siguientes diferencias:
- El ARNt iniciador es un ARNt especializado diferente que lleva metionina, llamado Met-ARNi
- En lugar de unirse al ARNm en la secuencia Shine-Dalgarno, el complejo de iniciación eucariota reconoce la tapa 5′ del ARNm eucariota, y luego sigue a lo largo del ARNm en la dirección 5′ a 3′ hasta que se reconoce el codón de inicio AUG. En este punto, la subunidad 60S se une al complejo de Met-tRNAi, ARNm y la subunidad 40S.

Figura 4. La traducción en las bacterias comienza con la formación del complejo de iniciación, que incluye la subunidad ribosomal pequeña, el ARNm, el ARNt iniciador que lleva N-formilmetionina y los factores de iniciación. Luego se une la subunidad 50S, formando un ribosoma intacto.
Elongación
En procariotas y eucariotas, los fundamentos de la elongación de la traducción son los mismos. En E. coli, la unión de la subunidad ribosomal 50S para producir el ribosoma intacto forma tres sitios ribosomales funcionalmente importantes: El sitio A (aminoacil) se une a los tRNAs aminoacil cargados entrantes. El sitio P (peptidil) se une a los ARNt cargados que llevan aminoácidos que han formado enlaces peptídicos con la cadena polipeptídica en crecimiento, pero que aún no se han disociado de su correspondiente ARNt. El sitio E (de salida) libera los ARNt disociados para que puedan recargarse con aminoácidos libres. Hay una notable excepción a esta línea de ensamblaje de los ARNt: Durante la formación del complejo de iniciación, el fMet-tRNAfMet bacteriano o el Met-tRNAi eucariótico entran directamente en el sitio P sin entrar primero en el sitio A, proporcionando un sitio A libre listo para aceptar el tRNA correspondiente al primer codón después del AUG.
La prolongación procede con movimientos de un solo código del ribosoma, cada uno de los cuales se denomina evento de translocación. Durante cada evento de translocación, los ARNt cargados entran en el sitio A, luego se desplazan al sitio P y finalmente al sitio E para su eliminación. Los movimientos ribosomales, o pasos, son inducidos por cambios conformacionales que hacen avanzar al ribosoma tres bases en la dirección 3′. Los enlaces peptídicos se forman entre el grupo amino del aminoácido unido al ARNt del sitio A y el grupo carboxilo del aminoácido unido al ARNt del sitio P. La formación de cada enlace peptídico es catalizada por la peptidil transferasa, una ribozima basada en el ARN que está integrada en la subunidad ribosomal 50S. El aminoácido unido al ARNt del sitio P también se une a la cadena polipeptídica en crecimiento. Cuando el ribosoma atraviesa el ARNm, el antiguo ARNt del sitio P entra en el sitio E, se separa del aminoácido y es expulsado. Varios de los pasos durante la elongación, incluyendo la unión de un aminoacil ARNt cargado al sitio A y la translocación, requieren energía derivada de la hidrólisis de GTP, que es catalizada por factores de elongación específicos. Sorprendentemente, el aparato de traducción de E. coli tarda sólo 0,05 segundos en añadir cada aminoácido, lo que significa que una proteína de 200 aminoácidos puede traducirse en sólo 10 segundos.
Terminación
La terminación de la traducción se produce cuando se encuentra un codón sin sentido (UAA, UAG o UGA) para el que no hay ARNt complementario. Al alinearse con el sitio A, estos codones sin sentido son reconocidos por factores de liberación en procariotas y eucariotas que hacen que el aminoácido del sitio P se separe de su ARNt, liberando el nuevo polipéptido. Las subunidades ribosómicas pequeñas y grandes se disocian del ARNm y entre sí; se reclutan casi inmediatamente en otro complejo de iniciación de la traducción.
En resumen, hay varias características clave que distinguen la expresión génica procariota de la observada en eucariotas. Éstas se ilustran en la Figura 5 y se enumeran en la Tabla 1.
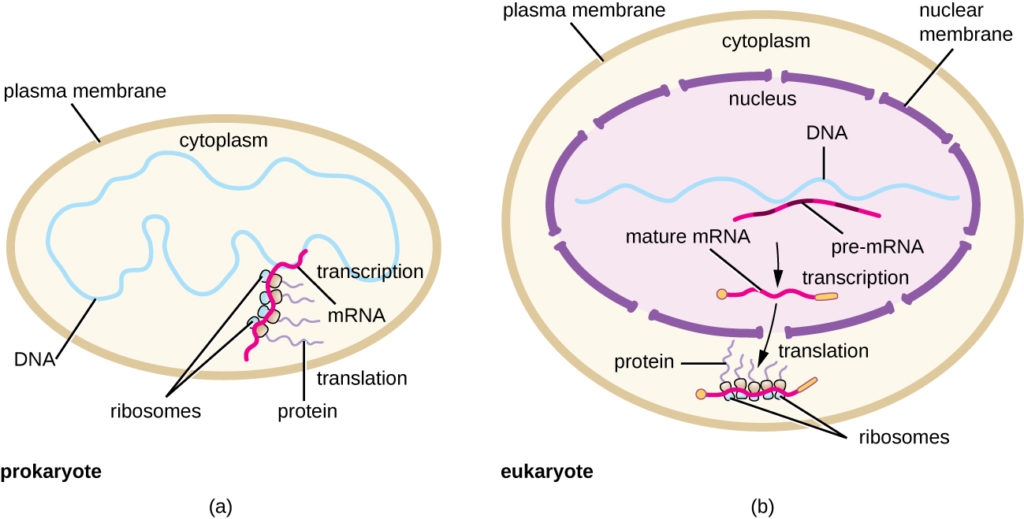
Figura 5. (a) En los procariotas, los procesos de transcripción y traducción ocurren simultáneamente en el citoplasma, lo que permite una rápida respuesta celular a una señal ambiental. (b) En los eucariotas, la transcripción se localiza en el núcleo y la traducción en el citoplasma, separando estos procesos y necesitando el procesamiento del ARN para su estabilidad.
| Tabla 1. Comparación de la traducción en bacterias frente a eucariotas | ||
|---|---|---|
| Propiedad | Bacterias | Eucariotas |
| Ribosomas | 70S
|
80S
|
| Aminoácido transportado por el tRNA iniciador | fMet | Met |
| Shine-Secuencia Dalgarno en ARNm | Presente | Absente |
| Transcripción y traducción simultáneas | Sí | No |
Compruebe su comprensión
Responda a la(s) siguiente(s) pregunta(s) para comprobar su comprensión de los temas tratados en la sección anterior. Este breve cuestionario no cuenta para su calificación en la clase, y puede repetirlo un número ilimitado de veces.
Use este cuestionario para comprobar su comprensión y decidir si (1) estudiar más la sección anterior o (2) pasar a la sección siguiente.