Beschreiben Sie den Prozess der prokaryotischen Transkription und Translation
Die Prokaryoten, zu denen Bakterien und Archaeen gehören, sind meist einzellige Organismen, denen per Definition membrangebundene Kerne und andere Organellen fehlen. Ein bakterielles Chromosom ist ein kovalent geschlossener Kreis, der im Gegensatz zu eukaryotischen Chromosomen nicht durch Histonproteine organisiert ist. Der zentrale Bereich der Zelle, in dem sich die prokaryotische DNA befindet, wird als Nukleoid bezeichnet. Darüber hinaus verfügen Prokaryonten oft über reichlich Plasmide, kürzere zirkuläre DNA-Moleküle, die nur ein oder wenige Gene enthalten können. Plasmide können während der Zellteilung unabhängig vom bakteriellen Chromosom übertragen werden und tragen oft Eigenschaften wie Antibiotikaresistenz in sich. Aufgrund dieser einzigartigen Merkmale unterscheiden sich die Transkription und die Genregulierung zwischen prokaryotischen und eukaryotischen Zellen etwas.
Lernziele
- Verstehen Sie die grundlegenden Schritte der Transkription von DNA in RNA in prokaryotischen Zellen
- Verstehen Sie die Grundlagen der prokaryotischen Translation und wie sie sich von der eukaryotischen Translation unterscheidet
Prokaryotische Transkription
Initiation der Transkription in Prokaryoten
Prokaryoten haben keine membranumschlosseneneingeschlossene Zellkerne. Daher können die Prozesse der Transkription, der Translation und des mRNA-Abbaus alle gleichzeitig stattfinden. Der intrazelluläre Gehalt eines bakteriellen Proteins kann durch mehrere gleichzeitig stattfindende Transkriptions- und Translationsvorgänge an derselben DNA-Vorlage schnell erhöht werden. Die prokaryotische Transkription umfasst oft mehr als ein Gen und produziert polycistronische mRNAs, die mehr als ein Protein spezifizieren.
In unserer Diskussion wird die Transkription anhand der Beschreibung dieses Prozesses in Escherichia coli, einer gut untersuchten Bakterienart, dargestellt. Obwohl es einige Unterschiede zwischen der Transkription in E. coli und der Transkription in Archaeen gibt, kann das Verständnis der E. coli-Transkription auf praktisch alle Bakterienarten angewandt werden.
Prokaryotische RNA-Polymerase
Prokaryoten verwenden dieselbe RNA-Polymerase zur Transkription aller ihrer Gene. In E. coli besteht die Polymerase aus fünf Polypeptid-Untereinheiten, von denen zwei identisch sind. Vier dieser Untereinheiten, die als α, α, β und β′ bezeichnet werden, bilden das Polymerase-Kernenzym. Diese Untereinheiten setzen sich jedes Mal zusammen, wenn ein Gen transkribiert wird, und bauen sich wieder ab, sobald die Transkription abgeschlossen ist. Jede Untereinheit hat eine einzigartige Aufgabe: Die beiden α-Untereinheiten sind notwendig, um die Polymerase an der DNA zu montieren; die β-Untereinheit bindet an das Ribonukleosidtriphosphat, das Teil des neu entstandenen mRNA-Moleküls wird; und die β′-Untereinheit bindet an den DNA-Musterstrang. Die fünfte Untereinheit, σ, ist nur an der Initiation der Transkription beteiligt. Sie verleiht der Transkription Spezifität, so dass die Polymerase mit der Synthese der mRNA an einer geeigneten Initiationsstelle beginnt. Ohne σ würde das Kernenzym an zufälligen Stellen transkribieren und mRNA-Moleküle produzieren, die Protein-Kauderwelsch spezifizieren. Die Polymerase, die aus allen fünf Untereinheiten besteht, wird als Holoenzym bezeichnet (ein Holoenzym ist eine biochemisch aktive Verbindung, die aus einem Enzym und seinem Coenzym besteht).
Prokaryotische Promotoren

Abbildung 1. Die σ-Untereinheit der prokaryotischen RNA-Polymerase erkennt Konsensussequenzen, die sich in der Promotorregion stromaufwärts der Transkriptionsstartstelle befinden. Die σ-Untereinheit dissoziiert von der Polymerase, nachdem die Transkription eingeleitet wurde.
Ein Promotor ist eine DNA-Sequenz, an die die Transkriptionsmaschine bindet und die Transkription einleitet. In den meisten Fällen befinden sich Promotoren stromaufwärts von den Genen, die sie regulieren. Die spezifische Sequenz eines Promotors ist sehr wichtig, da sie bestimmt, ob das entsprechende Gen ständig, manchmal oder selten transkribiert wird. Obwohl die Promotoren in den verschiedenen prokaryotischen Genomen unterschiedlich sind, gibt es einige konservierte Elemente. In den -10- und -35-Regionen stromaufwärts der Initiationsstelle gibt es zwei Promotor-Konsensus-Sequenzen oder Regionen, die in allen Promotoren und in verschiedenen Bakterienarten ähnlich sind (Abbildung 1).
Die -10-Konsensus-Sequenz, die so genannte -10-Region, ist TATAAT. Die -35-Sequenz, TTGACA, wird von σ erkannt und gebunden. Sobald diese Wechselwirkung hergestellt ist, binden die Untereinheiten des Kernenzyms an die Stelle. Die A-T-reiche -10-Region erleichtert das Abwickeln der DNA-Vorlage, und es werden mehrere Phosphodiesterbindungen hergestellt. Die Initiationsphase der Transkription endet mit der Produktion abortiver Transkripte, d.h. Polymere von etwa 10 Nukleotiden, die gebildet und wieder freigesetzt werden.
Verlängerung und Terminierung in Prokaryoten
Die Elongationsphase der Transkription beginnt mit der Freisetzung der σ-Untereinheit von der Polymerase. Die Dissoziation von σ ermöglicht es dem Kernenzym, sich entlang der DNA-Vorlage fortzubewegen und mRNA in der 5′- zu 3′-Richtung mit einer Geschwindigkeit von etwa 40 Nukleotiden pro Sekunde zu synthetisieren. Während der Elongation wird die DNA kontinuierlich vor dem Kernenzym abgewickelt und hinter ihm wieder aufgewickelt (Abbildung 2). Die Basenpaarung zwischen DNA und RNA ist nicht stabil genug, um die Stabilität der Komponenten der mRNA-Synthese zu gewährleisten. Stattdessen fungiert die RNA-Polymerase als stabiles Bindeglied zwischen der DNA-Vorlage und den entstehenden RNA-Strängen, um sicherzustellen, dass die Elongation nicht vorzeitig unterbrochen wird.

Abbildung 2. Klicken Sie für eine größere Darstellung. Während der Elongation folgt die prokaryotische RNA-Polymerase der DNA-Vorlage, synthetisiert mRNA in 5′- bis 3′-Richtung und wickelt die DNA beim Ablesen ab und wieder auf.
Prokaryotische Abbruchsignale
Nach der Transkription eines Gens muss die prokaryotische Polymerase angewiesen werden, sich von der DNA-Vorlage zu lösen und die neu gebildete mRNA freizusetzen. Je nach dem zu transkribierenden Gen gibt es zwei Arten von Terminierungssignalen. Das eine ist proteinbasiert, das andere basiert auf RNA. Die Rho-abhängige Terminierung wird durch das Rho-Protein gesteuert, das hinter der Polymerase auf der wachsenden mRNA-Kette mitläuft. In der Nähe des Endes des Gens stößt die Polymerase auf eine Reihe von G-Nukleotiden auf der DNA-Vorlage und kommt zum Stillstand. Infolgedessen stößt das Rho-Protein mit der Polymerase zusammen. Durch die Interaktion mit rho wird die mRNA aus der Transkriptionsblase freigesetzt.
Die rho-unabhängige Terminierung wird durch spezifische Sequenzen im DNA-Musterstrang gesteuert. Wenn sich die Polymerase dem Ende des zu transkribierenden Gens nähert, stößt sie auf eine Region, die reich an C-G-Nukleotiden ist. Die mRNA faltet sich auf sich selbst zurück, und die komplementären C-G-Nukleotide binden sich aneinander. Das Ergebnis ist eine stabile Haarnadel, die die Polymerase zum Stillstand bringt, sobald sie beginnt, einen Bereich mit vielen A-T-Nukleotiden zu transkribieren. Die komplementäre U-A-Region des mRNA-Transkripts bildet nur eine schwache Wechselwirkung mit der Template-DNA. In Verbindung mit der blockierten Polymerase führt dies zu einer ausreichenden Instabilität, so dass das Kernenzym abbricht und das neue mRNA-Transkript freisetzt.
Mit der Terminierung ist der Transkriptionsprozess abgeschlossen. Zum Zeitpunkt der Terminierung wäre das prokaryotische Transkript bereits verwendet worden, um die Synthese zahlreicher Kopien des kodierten Proteins zu beginnen, da diese Prozesse gleichzeitig ablaufen können. Die Vereinheitlichung von Transkription, Translation und sogar mRNA-Abbau ist möglich, weil alle diese Prozesse in der gleichen 5′-zu-3′-Richtung ablaufen und weil es in der prokaryotischen Zelle keine membranöse Kompartimentierung gibt (Abbildung 3). Im Gegensatz dazu schließt das Vorhandensein eines Zellkerns in eukaryotischen Zellen die gleichzeitige Transkription und Translation aus.

Abbildung 3. Mehrere Polymerasen können ein einziges bakterielles Gen transkribieren, während zahlreiche Ribosomen gleichzeitig die mRNA-Transkripte in Polypeptide übersetzen. Auf diese Weise kann ein spezifisches Protein schnell eine hohe Konzentration in der Bakterienzelle erreichen.
Besuchen Sie diese BioStudio-Animation, um den Prozess der prokaryotischen Transkription zu sehen.
Praxisfragen
Welche Untereinheit der E. coli-Polymerase verleiht der Transkription Spezifität?
- α
- β
- β′
- σ
Die -10- und -35-Regionen prokaryotischer Promotoren werden als Konsensussequenzen bezeichnet, weil ________.
- sie sind in allen Bakterienarten identisch
- sie sind in allen Bakterienarten ähnlich
- sie existieren in allen Organismen
Prokaryotische Translation
Die Translation ist bei Prokaryoten und Eukaryoten ähnlich. Im Folgenden wird untersucht, wie die Translation in E. coli, einem repräsentativen Prokaryonten, abläuft, und es werden etwaige Unterschiede zwischen bakterieller und eukaryotischer Translation aufgezeigt.
Initiation
Die Initiierung der Proteinsynthese beginnt mit der Bildung eines Initiationskomplexes. In E. coli umfasst dieser Komplex das kleine 30S-Ribosom, die mRNA-Vorlage, drei Initiationsfaktoren, die den korrekten Zusammenbau des Ribosoms unterstützen, Guanosintriphosphat (GTP), das als Energiequelle dient, und eine spezielle Initiator-tRNA, die N-Formyl-Methionin (fMet-tRNAfMet) trägt (Abbildung 4). Die Initiator-tRNA interagiert mit dem Startcodon AUG der mRNA und trägt ein formyliertes Methionin (fMet). Aufgrund seiner Beteiligung an der Initiation wird fMet am Anfang (N-Terminus) jeder von E. coli synthetisierten Polypeptidkette eingefügt. In der mRNA von E. coli interagiert eine Leadersequenz stromaufwärts des ersten AUG-Codons, die so genannte Shine-Dalgarno-Sequenz (auch bekannt als ribosomale Bindungsstelle AGGAGG), durch komplementäre Basenpaarung mit den rRNA-Molekülen, aus denen das Ribosom besteht. Durch diese Wechselwirkung wird die 30S ribosomale Untereinheit an der richtigen Stelle der mRNA-Vorlage verankert. An dieser Stelle bindet sich dann die 50S ribosomale Untereinheit an den Initiationskomplex und bildet ein intaktes Ribosom.
In Eukaryoten verläuft die Bildung des Initiationskomplexes ähnlich, mit den folgenden Unterschieden:
- Die Initiator-tRNA ist eine andere spezialisierte tRNA, die Methionin trägt, genannt Met-tRNAi
- Anstatt an der Shine-Dalgarno-Sequenz an die mRNA zu binden, erkennt der eukaryotische Initiationskomplex die 5′-Kappe der eukaryotischen mRNA und folgt dann der mRNA in Richtung 5′ zu 3′, bis das AUG-Startcodon erkannt wird. An diesem Punkt bindet die 60S-Untereinheit an den Komplex aus Met-tRNAi, mRNA und der 40S-Untereinheit.

Abbildung 4. Die Translation in Bakterien beginnt mit der Bildung des Initiationskomplexes, der die kleine ribosomale Untereinheit, die mRNA, die Initiator-tRNA, die N-Formyl-Methionin trägt, und Initiationsfaktoren umfasst. Dann bindet die 50S-Untereinheit und bildet ein intaktes Ribosom.
Elongation
In Prokaryonten und Eukaryonten sind die Grundlagen der Elongation der Translation gleich. In E. coli werden bei der Bindung der 50S ribosomalen Untereinheit zur Herstellung des intakten Ribosoms drei funktionell wichtige ribosomale Stellen gebildet: Die A (Aminoacyl)-Stelle bindet eingehende geladene Aminoacyl-tRNAs. Die P (Peptidyl)-Stelle bindet geladene tRNAs, die Aminosäuren tragen, die Peptidbindungen mit der wachsenden Polypeptidkette gebildet haben, aber noch nicht von ihrer entsprechenden tRNA dissoziiert sind. Die E (Exit)-Stelle setzt dissoziierte tRNAs frei, so dass sie mit freien Aminosäuren wieder aufgeladen werden können. Es gibt eine bemerkenswerte Ausnahme von diesem Fließband der tRNAs: Während der Bildung des Initiationskomplexes tritt die bakterielle fMet-tRNAfMet oder die eukaryotische Met-tRNAi direkt in die P-Stelle ein, ohne vorher in die A-Stelle einzutreten, wodurch eine freie A-Stelle entsteht, die bereit ist, die tRNA aufzunehmen, die dem ersten Codon nach dem AUG entspricht.
Die Verlängerung erfolgt durch Einzelcodon-Bewegungen des Ribosoms, die jeweils als Translokationsereignis bezeichnet werden. Während jedes Translokationsereignisses treten die geladenen tRNAs an der A-Stelle ein, verschieben sich dann zur P-Stelle und schließlich zur E-Stelle, um dort entfernt zu werden. Ribosomale Bewegungen oder Schritte werden durch Konformationsänderungen ausgelöst, die das Ribosom um drei Basen in 3′-Richtung voranbringen. Zwischen der Aminogruppe der Aminosäure, die an die A-site tRNA gebunden ist, und der Carboxylgruppe der Aminosäure, die an die P-site tRNA gebunden ist, bilden sich Peptidbindungen. Die Bildung jeder Peptidbindung wird durch Peptidyltransferase katalysiert, ein Ribozym auf RNA-Basis, das in die 50S ribosomale Untereinheit integriert ist. Die an die tRNA der P-Stelle gebundene Aminosäure wird ebenfalls an die wachsende Polypeptidkette gebunden. Wenn das Ribosom die mRNA durchläuft, gelangt die ehemalige tRNA der P-Stelle in die E-Stelle, löst sich von der Aminosäure und wird ausgestoßen. Mehrere der Schritte während der Elongation, einschließlich der Bindung einer geladenen Aminoacyl-tRNA an die A-Stelle und der Translokation, erfordern Energie, die aus der GTP-Hydrolyse stammt, die von spezifischen Elongationsfaktoren katalysiert wird. Erstaunlicherweise benötigt der E. coli-Translationsapparat nur 0,05 Sekunden, um jede Aminosäure hinzuzufügen, was bedeutet, dass ein Protein mit 200 Aminosäuren in nur 10 Sekunden übersetzt werden kann.
Terminierung
Die Terminierung der Translation erfolgt, wenn ein Nonsense-Codon (UAA, UAG oder UGA) auftritt, für das es keine komplementäre tRNA gibt. Beim Alignment mit der A-Stelle werden diese Nonsense-Codons von Release-Faktoren in Prokaryonten und Eukaryonten erkannt, die dazu führen, dass sich die P-Stellen-Aminosäure von ihrer tRNA löst und das neu gebildete Polypeptid freisetzt. Die kleinen und großen ribosomalen Untereinheiten dissoziieren von der mRNA und voneinander; sie werden fast sofort in einen anderen Translationsinitiationskomplex rekrutiert.
Zusammenfassend gibt es mehrere Schlüsselmerkmale, die die prokaryotische Genexpression von der in Eukaryonten unterscheiden. Diese sind in Abbildung 5 dargestellt und in Tabelle 1 aufgeführt.
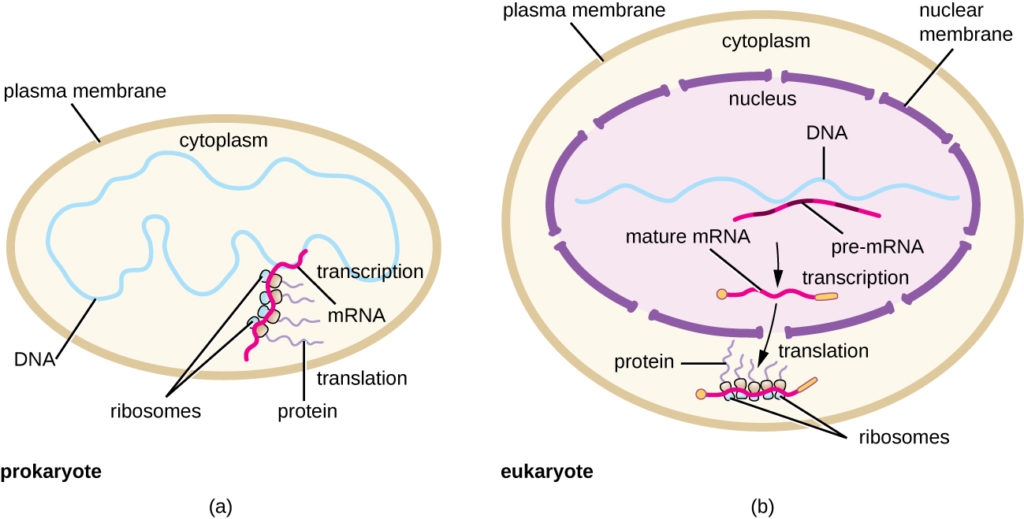
Abbildung 5. (a) Bei Prokaryonten finden die Prozesse der Transkription und Translation gleichzeitig im Zytoplasma statt, was eine schnelle zelluläre Reaktion auf einen Umweltreiz ermöglicht. (b) Bei Eukaryonten ist die Transkription im Zellkern und die Translation im Zytoplasma lokalisiert, wodurch diese Prozesse voneinander getrennt sind und eine RNA-Verarbeitung für die Stabilität erforderlich ist.
| Tabelle 1. Vergleich der Translation in Bakterien versus Eukaryoten | ||
|---|---|---|
| Eigenschaft | Bakterien | Eukaryoten |
| Ribosomen | 70S
|
80S
|
| Aminosäure getragen von Initiator-tRNA | fMet | Met |
| Glanz-Dalgarno-Sequenz in mRNA | Vorhanden | Abwesend |
| Simultane Transkription und Translation | Ja | Nein |
Prüfen Sie Ihr Verständnis
Beantworten Sie die folgende(n) Frage(n), um zu sehen, wie gut Sie die im vorherigen Abschnitt behandelten Themen verstehen. Dieses kurze Quiz zählt nicht zu Ihrer Note in der Klasse, und Sie können es beliebig oft wiederholen.
Nutzen Sie dieses Quiz, um Ihr Verständnis zu überprüfen und zu entscheiden, ob Sie (1) den vorherigen Abschnitt weiter studieren oder (2) zum nächsten Abschnitt übergehen wollen.