Definire il processo di trascrizione e traduzione procariotica
I procarioti, che includono batteri e archaea, sono per lo più organismi unicellulari che, per definizione, mancano di nuclei legati a membrana e altri organelli. Un cromosoma batterico è un cerchio chiuso covalentemente che, a differenza dei cromosomi eucarioti, non è organizzato intorno alle proteine istone. La regione centrale della cellula in cui risiede il DNA procariotico è chiamata nucleoide. Inoltre, i procarioti hanno spesso abbondanti plasmidi, che sono molecole di DNA circolare più corte che possono contenere solo uno o pochi geni. I plasmidi possono essere trasferiti indipendentemente dal cromosoma batterico durante la divisione cellulare e spesso portano tratti come la resistenza agli antibiotici. A causa di queste caratteristiche uniche, la trascrizione e la regolazione dei geni è un po’ diversa tra le cellule procariotiche e quelle eucariotiche.
Obiettivi di apprendimento
- Capire i passi fondamentali della trascrizione del DNA in RNA nelle cellule procariotiche
- Capire le basi della traduzione procariotica e come differisce dalla traduzione eucariotica
Trascrizione procariotica
Inizio della trascrizione nei procarioti
I procarioti non hanno nucleinuclei chiusi da una membrana. Pertanto, i processi di trascrizione, traduzione e degradazione dell’mRNA possono avvenire tutti simultaneamente. Il livello intracellulare di una proteina batterica può essere rapidamente amplificato da più eventi di trascrizione e traduzione che avvengono contemporaneamente sullo stesso modello di DNA. La trascrizione procariotica spesso copre più di un gene e produce mRNA policistronici che specificano più di una proteina.
La nostra discussione qui esemplifica la trascrizione descrivendo questo processo in Escherichia coli, una specie batterica ben studiata. Sebbene esistano alcune differenze tra la trascrizione in E. coli e la trascrizione negli archaea, la comprensione della trascrizione in E. coli può essere applicata praticamente a tutte le specie batteriche.
Polimerasi RNA procariotiche
I procarioti usano la stessa RNA polimerasi per trascrivere tutti i loro geni. In E. coli, la polimerasi è composta da cinque subunità polipeptidiche, due delle quali sono identiche. Quattro di queste subunità, denominate α, α, β, e β′ comprendono l’enzima centrale della polimerasi. Queste subunità si assemblano ogni volta che un gene viene trascritto, e si smontano una volta che la trascrizione è completa. Ogni subunità ha un ruolo unico; le due subunità α sono necessarie per assemblare la polimerasi sul DNA; la subunità β si lega al trifosfato ribonucleosidico che diventerà parte della molecola di mRNA nascente “appena nata”; e la β′ si lega al filamento di DNA modello. La quinta subunità, σ, è coinvolta solo nell’inizio della trascrizione. Essa conferisce una specificità trascrizionale tale che la polimerasi inizia a sintetizzare l’mRNA da un sito di iniziazione appropriato. Senza σ, l’enzima centrale trascriverebbe da siti casuali e produrrebbe molecole di mRNA che specificano proteine senza senso. La polimerasi composta da tutte e cinque le subunità è chiamata oloenzima (un oloenzima è un composto biochimicamente attivo composto da un enzima e dal suo coenzima).
Promotori procarioti

Figura 1. La subunità σ della RNA polimerasi procariotica riconosce le sequenze di consenso che si trovano nella regione del promotore a monte del punto di inizio della trascrizione. La subunità σ si dissocia dalla polimerasi dopo che la trascrizione è stata iniziata.
Un promotore è una sequenza di DNA su cui il macchinario di trascrizione si lega e inizia la trascrizione. Nella maggior parte dei casi, i promotori esistono a monte dei geni che regolano. La sequenza specifica di un promotore è molto importante perché determina se il gene corrispondente viene trascritto sempre, alcune volte o raramente. Anche se i promotori variano tra i genomi procarioti, alcuni elementi sono conservati. Nelle regioni -10 e -35 a monte del sito di iniziazione, ci sono due sequenze di consenso del promotore, o regioni che sono simili in tutti i promotori e nelle varie specie batteriche (Figura 1).
La sequenza di consenso -10, chiamata regione -10, è TATAAT. La sequenza -35, TTGACA, è riconosciuta e legata da σ. Una volta realizzata questa interazione, le subunità dell’enzima centrale si legano al sito. La regione -10, ricca di A-T, facilita lo srotolamento del modello di DNA e vengono creati diversi legami fosfodiesteri. La fase di iniziazione della trascrizione termina con la produzione di trascrizioni abortive, che sono polimeri di circa 10 nucleotidi che vengono realizzati e rilasciati.
Allungamento e terminazione nei procarioti
La fase di allungamento della trascrizione inizia con il rilascio della subunità σ dalla polimerasi. La dissociazione di σ permette all’enzima centrale di procedere lungo il modello di DNA, sintetizzando mRNA in direzione 5′-3′ ad una velocità di circa 40 nucleotidi al secondo. Mentre l’elongazione procede, il DNA viene continuamente srotolato davanti al nucleo dell’enzima e riavvolto dietro di esso (Figura 2). L’accoppiamento delle basi tra DNA e RNA non è abbastanza stabile da mantenere la stabilità dei componenti della sintesi dell’mRNA. Invece, l’RNA polimerasi agisce come un collegamento stabile tra il modello di DNA e i filamenti nascenti di RNA per assicurare che l’elongazione non sia interrotta prematuramente.

Figura 2. Clicca per ingrandire l’immagine. Durante l’elongazione, la RNA polimerasi procariotica segue le tracce lungo il modello di DNA, sintetizza l’mRNA nella direzione da 5′ a 3′, e svolge e riavvolge il DNA mentre viene letto.
Segnali di terminazione procariotica
Una volta che un gene viene trascritto, la polimerasi procariotica deve essere istruita a dissociarsi dal modello di DNA e liberare l’mRNA appena prodotto. A seconda del gene che viene trascritto, ci sono due tipi di segnali di terminazione. Uno è basato sulle proteine e l’altro sull’RNA. La terminazione Rho-dipendente è controllata dalla proteina rho, che segue la polimerasi sulla catena di mRNA in crescita. Verso la fine del gene, la polimerasi incontra una serie di nucleotidi G sul modello di DNA e si blocca. Come risultato, la proteina rho si scontra con la polimerasi. L’interazione con rho libera l’mRNA dalla bolla di trascrizione.
La terminazione indipendente da rho è controllata da sequenze specifiche nel filamento del DNA template. Quando la polimerasi si avvicina alla fine del gene da trascrivere, incontra una regione ricca di nucleotidi C-G. L’mRNA si ripiega su se stesso, e i nucleotidi C-G complementari si legano insieme. Il risultato è una forcina stabile che causa lo stallo della polimerasi non appena inizia a trascrivere una regione ricca di nucleotidi A-T. La regione complementare U-A della trascrizione dell’mRNA forma solo una debole interazione con il DNA modello. Questo, insieme alla polimerasi in stallo, induce un’instabilità sufficiente perché l’enzima centrale si stacchi e liberi il nuovo trascritto dell’mRNA.
Con la terminazione, il processo di trascrizione è completo. Nel momento in cui avviene la terminazione, il trascritto procariotico sarebbe già stato utilizzato per iniziare la sintesi di numerose copie della proteina codificata, perché questi processi possono avvenire contemporaneamente. L’unificazione della trascrizione, della traduzione e anche della degradazione dell’mRNA è possibile perché tutti questi processi avvengono nella stessa direzione da 5′ a 3′ e perché non c’è compartimentazione membranosa nella cellula procariotica (Figura 3). Al contrario, la presenza di un nucleo nelle cellule eucariotiche preclude la trascrizione e la traduzione simultanee.

Figura 3. Più polimerasi possono trascrivere un singolo gene batterico mentre numerosi ribosomi traducono simultaneamente i trascritti dell’mRNA in polipeptidi. In questo modo, una proteina specifica può raggiungere rapidamente un’alta concentrazione nella cellula batterica.
Visita questa animazione BioStudio per vedere il processo di trascrizione procariotica.
Domande pratiche
Quale subunità della polimerasi E. coli conferisce specificità alla trascrizione?
- α
- β
- β′
- σ
Le regioni -10 e -35 dei promotori procarioti sono chiamate sequenze di consenso perché ________.
- sono identiche in tutte le specie batteriche
- sono simili in tutte le specie batteriche
- esistono in tutti gli organismi
- hanno la stessa funzione in tutti gli organismi
Traduzione procariotica
La traduzione è simile nei procarioti e negli eucarioti. Qui esploreremo come avviene la traduzione in E. coli, un procariote rappresentativo, e specificheremo ogni differenza tra la traduzione batterica e quella eucariotica.
Iniziazione
L’inizio della sintesi proteica inizia con la formazione di un complesso di iniziazione. In E. coli, questo complesso coinvolge il piccolo ribosoma 30S, il modello di mRNA, tre fattori di iniziazione che aiutano il ribosoma ad assemblarsi correttamente, guanosina trifosfato (GTP) che agisce come fonte di energia, e uno speciale tRNA iniziatore che trasporta N-formil-metionina (fMet-tRNAfMet) (Figura 4). Il tRNA iniziatore interagisce con il codone di inizio AUG dell’mRNA e porta una metionina formilata (fMet). A causa del suo coinvolgimento nell’iniziazione, fMet è inserito all’inizio (terminale N) di ogni catena polipeptidica sintetizzata da E. coli. Nell’mRNA dell’E. coli, una sequenza leader a monte del primo codone AUG, chiamata sequenza Shine-Dalgarno (nota anche come sito di legame ribosomiale AGGAGG), interagisce attraverso un accoppiamento di basi complementari con le molecole di rRNA che compongono il ribosoma. Questa interazione ancorerà la subunità ribosomiale 30S alla posizione corretta sul template dell’mRNA. A questo punto, la subunità ribosomiale 50S si lega al complesso di iniziazione, formando un ribosoma intatto.
Negli eucarioti, la formazione del complesso di iniziazione è simile, con le seguenti differenze:
- Il tRNA iniziatore è un diverso tRNA specializzato che trasporta metionina, chiamato Met-tRNAi
- Invece di legarsi all’mRNA alla sequenza Shine-Dalgarno, il complesso di iniziazione eucariotico riconosce il tappo 5′ dell’mRNA eucariotico, poi segue lungo l’mRNA in direzione 5′-3′ fino a riconoscere il codone di inizio AUG. A questo punto, la subunità 60S si lega al complesso di Met-tRNAi, mRNA e la subunità 40S.

Figura 4. La traduzione nei batteri inizia con la formazione del complesso di iniziazione, che include la piccola subunità ribosomiale, l’mRNA, il tRNA iniziatore che trasporta N-formil-metionina e i fattori di iniziazione. Poi la subunità 50S si lega, formando un ribosoma intatto.
Allungamento
Nei procarioti e negli eucarioti, le basi dell’allungamento della traduzione sono le stesse. In E. coli, il legame della subunità ribosomiale 50S per produrre il ribosoma intatto forma tre siti ribosomiali funzionalmente importanti: Il sito A (aminoacile) lega i tRNA aminoacili carichi in entrata. Il sito P (peptidile) lega i tRNA carichi che trasportano aminoacidi che hanno formato legami peptidici con la catena polipeptidica in crescita ma non si sono ancora dissociati dal loro tRNA corrispondente. Il sito E (uscita) rilascia i tRNA dissociati in modo che possano essere ricaricati con aminoacidi liberi. C’è una notevole eccezione a questa linea di assemblaggio dei tRNA: Durante la formazione del complesso di iniziazione, l’fMet-tRNAfMet batterico o il Met-tRNAi eucariotico entra direttamente nel sito P senza entrare prima nel sito A, fornendo un sito A libero pronto ad accettare il tRNA corrispondente al primo codone dopo l’AUG.
L’allungamento procede con movimenti a singolo codone del ribosoma, ciascuno chiamato evento di traslocazione. Durante ogni evento di traslocazione, i tRNA carichi entrano nel sito A, poi si spostano nel sito P e infine nel sito E per la rimozione. I movimenti ribosomiali, o passi, sono indotti da cambiamenti conformazionali che fanno avanzare il ribosoma di tre basi nella direzione 3′. I legami peptidici si formano tra il gruppo amminico dell’aminoacido attaccato al tRNA del sito A e il gruppo carbossilico dell’aminoacido attaccato al tRNA del sito P. La formazione di ogni legame peptidico è catalizzata dalla peptidil transferasi, un ribozima basato sull’RNA che è integrato nella subunità ribosomiale 50S. L’aminoacido legato al tRNA del sito P è anche legato alla catena polipeptidica in crescita. Quando il ribosoma attraversa l’mRNA, l’ex tRNA del sito P entra nel sito E, si stacca dall’aminoacido e viene espulso. Molti dei passi durante l’elongazione, compreso il legame di un tRNA aminoacilico carico al sito A e la traslocazione, richiedono energia derivata dall’idrolisi del GTP, che è catalizzata da specifici fattori di elongazione. Sorprendentemente, l’apparato di traduzione dell’E. coli impiega solo 0,05 secondi per aggiungere ogni aminoacido, il che significa che una proteina di 200 aminoacidi può essere tradotta in soli 10 secondi.
Terminazione
La terminazione della traduzione avviene quando si incontra un codone non senso (UAA, UAG o UGA) per il quale non esiste un tRNA complementare. Allineandosi con il sito A, questi codoni senza senso sono riconosciuti da fattori di rilascio nei procarioti e negli eucarioti che hanno come risultato il distacco dell’aminoacido del sito P dal suo tRNA, rilasciando il polipeptide appena prodotto. Le piccole e grandi subunità ribosomiali si dissociano dall’mRNA e l’una dall’altra; sono reclutate quasi immediatamente in un altro complesso di iniziazione della traduzione.
In sintesi, ci sono diverse caratteristiche chiave che distinguono l’espressione genica procariotica da quella vista negli eucarioti. Queste sono illustrate nella Figura 5 ed elencate nella Tabella 1.
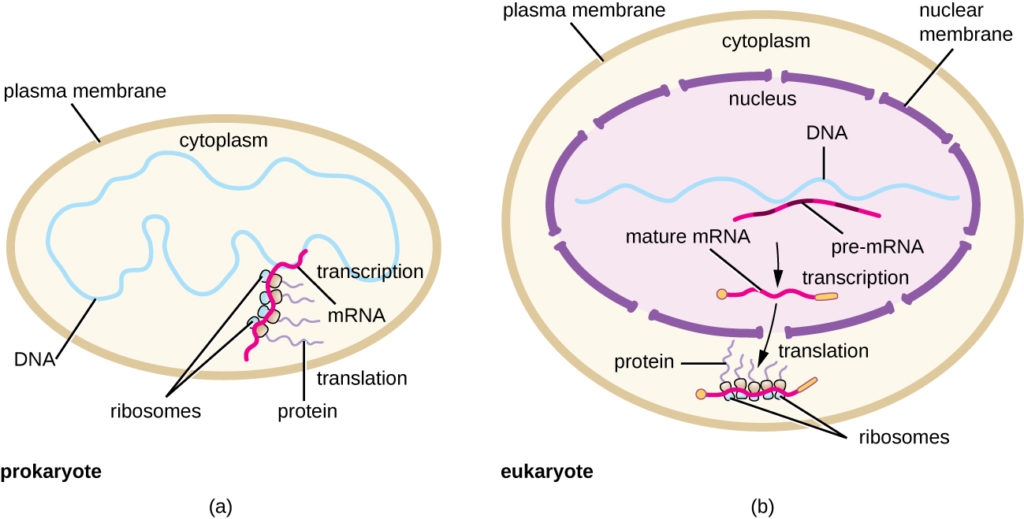
Figura 5. (a) Nei procarioti, i processi di trascrizione e traduzione avvengono simultaneamente nel citoplasma, permettendo una rapida risposta cellulare a un segnale ambientale. (b) Negli eucarioti, la trascrizione è localizzata nel nucleo e la traduzione è localizzata nel citoplasma, separando questi processi e richiedendo l’elaborazione dell’RNA per la stabilità.
| Tabella 1. Confronto della traduzione nei batteri rispetto agli eucarioti | ||
|---|---|---|
| Proprietà | Batteri | Eucarioti |
| Ribosomi | 70S
|
80S
|
| Aminoacido trasportato dal tRNA iniziatore | fMet | Met |
| Luce-Sequenza Dalgarno in mRNA | Presente | Assente |
| Trascrizione e traduzione simultanea | Sì | No |
Verifica la tua comprensione
Rispondi alla/e domanda/e qui sotto per vedere quanto hai capito gli argomenti trattati nella sezione precedente. Questo breve quiz non conta per il tuo voto in classe, e puoi ripeterlo un numero illimitato di volte.
Utilizza questo quiz per verificare la tua comprensione e decidere se (1) studiare ulteriormente la sezione precedente o (2) passare alla sezione successiva.