原核生物の転写と翻訳の過程を概説する
原核生物は、細菌や古細菌を含み、そのほとんどが単細胞生物であるため、膜で囲まれた核やその他の器官がないことが定義されています。 バクテリアの染色体は共有結合で閉じた円形で、真核生物の染色体とは異なり、ヒストン蛋白を中心に組織化されていない。 原核生物のDNAが存在する細胞の中心部をヌクレオイドと呼びます。 さらに、原核生物はプラスミドを豊富に持つことが多い。プラスミドはより短い円形のDNA分子で、1つまたは数個の遺伝子しか持っていないこともある。 プラスミドは、細胞分裂の際に細菌の染色体とは独立して移動することができ、抗生物質耐性などの形質を持っていることが多い。 これらのユニークな特徴のため、転写や遺伝子調節は原核細胞と真核細胞では多少異なっている。
学習目標
- 原核細胞におけるDNAのRNAへの転写の基本ステップを理解する
- 原核細胞の翻訳の基本を理解する と真核生物の翻訳との違い
原核生物の転写
原核生物における転写の開始
原核生物には膜がありません。は核を内包している。 したがって、転写、翻訳、mRNAの分解のプロセスはすべて同時に起こる可能性があります。 細菌タンパク質の細胞内レベルは、同じDNAテンプレート上で同時に起こる複数の転写および翻訳イベントによって、迅速に増幅されることができる。 原核生物の転写は、しばしば複数の遺伝子をカバーし、複数のタンパク質を特定するポリシストロンmRNAを生成する
ここでの議論は、よく研究されている細菌種である大腸菌におけるこのプロセスを説明することによって、転写を例証する。 大腸菌の転写と古細菌の転写にはいくつかの違いがありますが、大腸菌の転写の理解は事実上すべての細菌種に適用することができます。 大腸菌では、ポリメラーゼは5つのポリペプチドサブユニットから構成され、そのうち2つは同一である。 これらのサブユニットのうち、α、β、β′と表記される4つのサブユニットがポリメラーゼのコア酵素を構成している。 これらのサブユニットは、遺伝子が転写されるたびに集合し、転写が完了すると分解する。 2つのα-サブユニットはポリメラーゼをDNA上で組み立てるのに必要であり、β-サブユニットは「生まれたばかりの」mRNA分子の一部となるリボヌクレオシド三リン酸に結合し、β′はDNA鋳型鎖に結合している。 5番目のサブユニットであるσは、転写の開始のみに関与している。 このサブユニットは、ポリメラーゼが適切な開始部位からmRNAの合成を開始するような転写特異性を与える。 σがなければ、コア酵素はランダムな部位から転写を行い、タンパク質のちんぷんかんぷんなmRNA分子を生成することになる。
Prokaryotic Promoters

図1. 原核生物RNAポリメラーゼのσサブユニットは、転写開始点上流のプロモーター領域に見出されるコンセンサス配列を認識する。
プロモーターとは、転写機構が結合して転写を開始するDNA配列のことである。 ほとんどの場合、プロモーターは、それが制御する遺伝子の上流に存在する。 プロモーターの配列は、対応する遺伝子が常に転写されるのか、ある程度転写されるのか、あるいはほとんど転写されないのかを決定するため、非常に重要である。 プロモーターは原核生物ゲノムによって異なるが、いくつかの要素は保存されている。 開始点上流の-10領域と-35領域には、2つのプロモーターコンセンサス配列、すなわちすべてのプロモーターや様々な細菌種で類似している領域がある(図1)。 35番目の配列であるTTGACAは、σによって認識され結合される。 この相互作用がなされると、コア酵素のサブユニットがその部位に結合する。 A-Tに富んだ-10領域はDNA鋳型の巻き戻しを容易にし、いくつかのホスホジエステル結合が作られる。 転写開始期は、約10ヌクレオチドのポリマーである頓挫転写物が作られ、放出されて終了する。
原核生物の伸長と終結
転写伸長期は、σサブユニットがポリメラーゼから解放されて開始される。 σの解離によりコア酵素はDNA鋳型に沿って進み、1秒間に約40ヌクレオチドの速度で5′から3′の方向にmRNAを合成する。 伸長の進行に伴い、DNAはコア酵素の前方で巻き戻され、後方で巻き戻されるという連続的な現象が起こる(図2)。 DNAとRNAの塩基対形成は、mRNA合成成分の安定性を維持するほどには安定ではない。 その代わり、RNAポリメラーゼはDNA鋳型と新生RNA鎖の間の安定したリンカーとして働き、伸長が早期に中断されないようにしている。
原核生物の終結シグナル
遺伝子が転写されると、原核生物のポリメラーゼはDNAテンプレートから解離し、新しく作られたmRNAを放出するように指示される必要がある。 転写される遺伝子によって、2種類の終結シグナルがある。 ひとつはタンパク質ベースで、もうひとつはRNAベースである。 Rho依存性の終結は、ポリメラーゼの後ろをついてくるrhoタンパク質によって制御されている。 遺伝子の末端付近で、ポリメラーゼはDNA鋳型上のGヌクレオチドの走りに遭遇し、停止してしまう。 その結果、rhoタンパク質がポリメラーゼに衝突する。 rhoとの相互作用によりmRNAは転写バブルから解放される。
Rho-independent terminationはDNA鋳型鎖の特定の配列によって制御される。 ポリメラーゼが転写される遺伝子の末端に近づくと、C-Gヌクレオチドが豊富な領域に遭遇する。 mRNAはそれ自身の上に折り返され、相補的なC-Gヌクレオチドが結合する。 その結果、安定したヘアピンができ、ポリメラーゼはA-Tヌクレオチドが豊富な領域を転写し始めるとすぐに失速してしまう。 mRNAの転写産物の相補的なU-A領域は、鋳型DNAと弱い相互作用を形成するのみである。 このことは、停止したポリメラーゼと相まって、コア酵素が分離して新しいmRNA転写物を遊離するのに十分な不安定性を誘発する。
終了すると、転写のプロセスは完了する。終了が起こるまでに、原核生物の転写物はすでに、コードされたタンパク質の多数のコピーの合成を開始するために使用されていただろう。 転写、翻訳、そしてmRNAの分解の統一が可能なのは、これらの過程がすべて同じ5′から3′の方向で起こり、原核生物細胞には膜上の区画がないからである(図3)。 一方、真核細胞では核があるため、転写と翻訳が同時に行われない。

図3. 複数のポリメラーゼが1つの細菌遺伝子を転写し、同時に多数のリボソームがmRNA転写物をポリペプチドに翻訳することができる。 このようにして、特定のタンパク質は急速に細菌細胞内で高濃度に達することができます。
BioStudioのアニメーションで、原核生物の転写のプロセスをご覧ください。
- α
- β′
- σ
原核生物のプロモーターの-10と-35の領域は、_______からコンセンサス配列と呼ばれている。
- それらはすべての細菌種で同一である
- それらはすべての細菌種で似ている
- それらはすべての生物で存在する
- それらはすべての生物で同じ機能を持つ
原核生物の翻訳
原核生物と真核生物では翻訳が似ています。 ここでは、代表的な原核生物である大腸菌でどのように翻訳が行われるのかを探り、細菌と真核生物の翻訳の違いを明記する。
開始
タンパク質合成の開始は開始複合体の形成で始まる。 大腸菌では、この複合体には、小さな30Sリボソーム、mRNAテンプレート、リボソームの正しい組み立てを助ける3つの開始因子、エネルギー源として働くグアノシン三リン酸(GTP)、N-ホルミル-メチオニンを運ぶ特別な開始因子tRNA(fMet-tRNAfMet)(図4) が含まれている。 イニシエーターtRNAは、mRNAの開始コドンAUGと相互作用し、ホルミル化メチオニン(fMet)を担持している。 fMetは開始に関わっているため、大腸菌が合成するすべてのポリペプチド鎖の先頭(N末端)に挿入されている。 大腸菌のmRNAでは、最初のAUGコドンの上流にShine-Dalgarno配列(リボソーム結合部位AGGAGGとも呼ばれる)と呼ばれるリーダー配列があり、リボソームを構成するrRNA分子と相補的に塩基対を形成して相互作用している。 この相互作用により、30SリボソームサブユニットはmRNA鋳型上の正しい位置に固定される。 この時点で、50Sリボソームサブユニットが開始複合体に結合し、無傷のリボソームが形成される。
真核生物では、開始複合体の形成は似ているが、次のような違いがある。
- 開始tRNAはMet-tRNAiと呼ばれるメチオニンを運ぶ異なる特殊なtRNAである
- Shine-Dalgarno配列でmRNAに結合するのではなく、真核生物の開始複合体は真核生物のmRNAの5分キャップを認識し、AUG開始コドンを認識するまでmRNAに沿って5分→3分方向へ追跡する。 この時、60SサブユニットはMet-tRNAi、mRNA、40Sサブユニットの複合体に結合する

図4.ポリペプチドが葉を出し、他のすべての構成要素が互いに解離する。 バクテリアの翻訳は、小リボソームサブユニット、mRNA、N-ホルミル-メチオニンを運ぶ開始点tRNA、および開始因子を含む開始複合体の形成で始まる。 その後、50Sサブユニットが結合し、無傷のリボソームを形成する。
伸長
原核生物と真核生物では、翻訳の伸長の基本は同じである。 大腸菌では、50Sリボソームサブユニットが結合して無傷のリボソームを生成する際に、機能的に重要な3つのリボソーム部位が形成される。 A(アミノアシル)部位は、荷電したアミノアシルtRNAを結合する。 P(ペプチジル)部位は、成長中のポリペプチド鎖とペプチド結合を形成しているが、まだ対応するtRNAから解離していないアミノ酸を運ぶ荷電tRNAを結合する。 E(exit)部位は、解離したtRNAを解放し、遊離アミノ酸を再充填する。 このようなtRNAの組み立てには、1つだけ顕著な例外がある。 開始複合体形成の際、バクテリアのfMet-tRNAfMetや真核生物のMet-tRNAiは、最初にAサイトに入ることなく直接Pサイトに入り、AUG後の最初のコドンに対応するtRNAを受け入れる準備ができている空きAサイトとなる。 各転座イベントにおいて、荷電したtRNAはA部位に入り、次にP部位に移動し、最後にE部位に移動して除去される。 リボソームの移動(ステップ)は、リボソームを3塩基ずつ3方向に前進させる構造変化によって引き起こされる。 AサイトのtRNAに結合したアミノ酸のアミノ基と、PサイトのtRNAに結合したアミノ酸のカルボキシル基の間にペプチド結合が形成される。 それぞれのペプチド結合の形成は、50Sリボソームサブユニットに組み込まれたRNAベースのリボザイムであるペプチジルトランスフェラーゼによって触媒される。 PサイトtRNAに結合したアミノ酸は、成長するポリペプチド鎖にも結合する。 リボソームがmRNAを横切るとき、かつてのPサイトtRNAはEサイトに入り、アミノ酸から切り離され、排出される。 伸長過程のいくつかのステップ(荷電したアミノアシルtRNAのA部位への結合や転位など)には、特定の伸長因子によって触媒されるGTP加水分解から得られるエネルギーが必要である。 大腸菌の翻訳装置では、驚くべきことに、各アミノ酸を加えるのにわずか 0.05 秒しかかからない。つまり、200 アミノ酸のタンパク質がわずか 10 秒で翻訳されることになる。 これらのナンセンスコドンは原核生物および真核生物の放出因子によって認識され、Pサイトアミノ酸がそのtRNAから切り離され、新しく作られたポリペプチドが放出される。 小リボソームと大リボソームサブユニットはmRNAとお互いから解離し、ほとんどすぐに別の翻訳開始複合体に採用される。
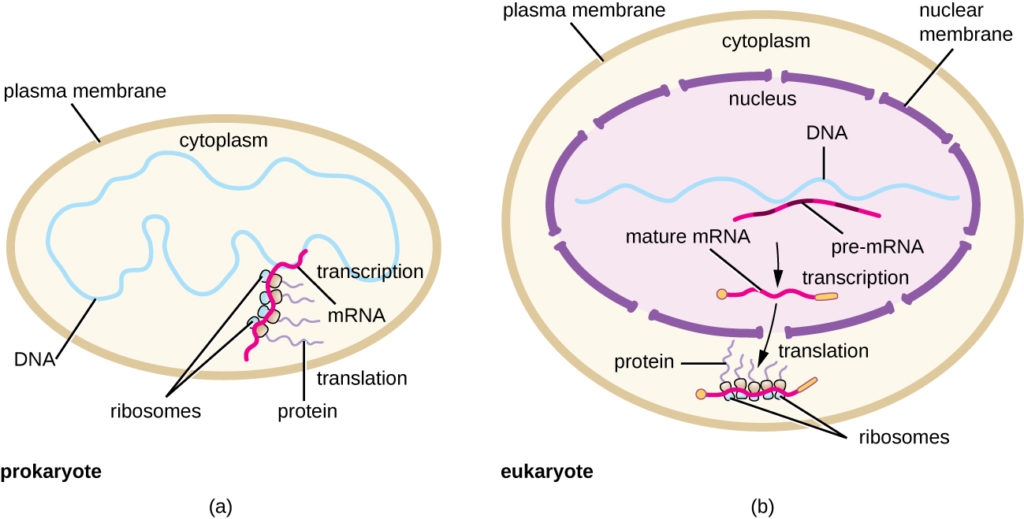
図5. (a)原核生物では、転写と翻訳のプロセスが細胞質で同時に行われるため、環境の手がかりに対して細胞が迅速に反応することができる。 (b)真核生物では、転写は核に、翻訳は細胞質に局在し、これらのプロセスは分離され、安定性のためにRNAプロセッシングが必要である。 細菌と真核生物の翻訳の比較
- 30S(真核生物)
30S - 50S (大サブユニット)と5Sおよび23S rRNAサブユニット
- 40S(小サブユニット)と18S rRNAサブユニット
- 60S(大サブユニット)5Sと。 5.8S, 28S rRNAサブユニット
70S - 30S
80S
Check Your Understanding
以下の質問に答えて、前のセクションでカバーしたトピックをどの程度理解しているかを確認してください。 このクイズは授業の成績には含まれません。また、何度でも再受験することができます。
このクイズで理解度を確認し、(1)前のセクションをさらに勉強するか、(2)次のセクションに進むかを決定してください。