Einführung
Gramm-positive Bakterien der Gattungen Bacillus und Clostridium können sich differenzieren, um eine Endospore (Spore) zu bilden, eine metabolisch ruhende Zelle, die als Reaktion auf Bedingungen produziert wird, die kein Zellwachstum erlauben (McKenney et al., 2012). Nach ihrer Freisetzung in die Umwelt überlebt die Spore in ihrem Ruhezustand extrem lange und widersteht einer Vielzahl von Stressfaktoren wie hohen Temperaturen, Austrocknung, Nährstoffmangel und dem Vorhandensein toxischer Chemikalien (McKenney et al., 2012). Die ruhende Spore ist jedoch in der Lage, die Umwelt kontinuierlich zu überwachen und auf das Vorhandensein von Wasser und Nährstoffen zu reagieren, indem sie keimt und eine vegetative Zelle hervorbringt, die wachsen und sporulieren kann (McKenney et al., 2012). Die Resistenz gegenüber unphysiologischen Bedingungen ist zum Teil auf die Oberflächenstrukturen der Sporen zurückzuführen. Bei Bacillus subtilis, dem Modellsystem für Sporenbildner, ist die Sporenoberfläche in einer mehrschichtigen Hülle und in einer Kruste organisiert, einer kürzlich entdeckten Schicht, die die Sporenhülle umgibt (McKenney et al., 2012). B. subtilis-Sporen sind negativ geladen (Huang et al., 2010; Pesce et al., 2014) und weisen eine relative Hydrophobie auf, die zum Teil auf die Glykosylierung einiger Sporenoberflächenproteine zurückzuführen ist (Cangiano et al., 2014; Rusciano et al., 2014). Bei mehreren Bacillus- und Clostridium-Arten, darunter B. cereus, B. anthracis, B. megaterium und C. difficile, ist die äußerste Sporenstruktur das Exosporium, eine morphologisch unterschiedliche Schicht aus Proteinen und Glykoproteinen, die die Hülle umgibt (Díaz-González et al., 2015; Manetsberger et al, 2015b; Stewart, 2015).
Die bakterielle Spore wurde als Plattform für die Darstellung heterologer Proteine vorgeschlagen, wobei die potenziellen Anwendungen von der Entwicklung von Schleimhautimpfstoffen bis hin zu wiederverwendbaren Biokatalysatoren, Diagnoseinstrumenten und Bioremediationsgeräten reichen (Knecht et al., 2011; Isticato und Ricca, 2014; Ricca et al., 2014). Die bemerkenswerte und gut dokumentierte Resistenz der Sporen (McKenney et al., 2012), die Eignung mehrerer sporenbildender Arten für genetische Manipulationen (Harwood und Cutting, 1990) und die Sicherheitsbilanz mehrerer Arten (Cutting, 2011; Baccigalupi et al., 2015) sprechen für die Verwendung der Sporen als Darstellungs- und Übertragungssystem. Es wurden zwei Strategien zur Darstellung heterologer Proteine auf der Sporenoberfläche entwickelt. Eine rekombinante Strategie, die auf der Konstruktion von Genfusionen zwischen DNA, die für ein ausgewähltes Sporenoberflächenprotein kodiert, und DNA, die für das darzustellende Protein kodiert, basiert, wurde im Laufe der Jahre zur Darstellung einer Vielzahl heterologer Proteine verwendet (Isticato und Ricca, 2014). Ein nicht rekombinanter Ansatz, der auf der spontanen Adsorption zwischen gereinigten Sporen und gereinigten Proteinen beruht, wurde ebenfalls zur Darstellung verschiedener Enzyme und Antigene verwendet (Ricca et al., 2014). Die molekularen Details, die die Adsorption von Sporen steuern, sind noch nicht vollständig geklärt. Es ist bekannt, dass die Adsorption effizienter ist, wenn der pH-Wert des Bindungspuffers sauer ist (pH 4) (Huang et al., 2010; Sirec et al., 2012) und dass wahrscheinlich eine Kombination aus elektrostatischen und hydrophoben Wechselwirkungen an der Interaktion beteiligt ist (Huang et al., 2010; Sirec et al., 2012). Es ist auch bekannt, dass mutierte Sporen mit stark veränderten Sporenoberflächen effizienter als isogene Wildtypsporen mit Modellproteinen interagieren (Sirec et al., 2012; Donadio et al, 2016).
Hier haben wir ein fluoreszierendes Protein, die monomere Form des rot fluoreszierenden Proteins (mRFP) der Koralle Discosoma sp. (Campbell et al., 2002), verwendet, um zu bewerten, ob Sporen von B. megaterium mit einem heterologen Modellprotein interagieren und es adsorbieren können. B. megaterium umfasst eine Reihe von morphologisch unterschiedlichen Stämmen, die die ungewöhnlich großen Abmessungen sowohl der Zellen (Länge bis zu 4 μm und Durchmesser von 1,5 μm) als auch der Sporen (Länge bis zu 3 μm und Durchmesser von 1 μm) gemeinsam haben (Di Luccia et al., 2016). Die Sporen einiger Stämme von B. megaterium sind von einem Exosporium umgeben, und da bisher nur Sporen von Arten ohne Exosporium als Adsorptionsplattformen in Betracht gezogen wurden, liegen keine Daten über den Einfluss des Exosporiums auf die Interaktion mit heterologen Proteinen vor.
Der Stamm QM B1551 ist der am besten charakterisierte Stamm von B. megaterium. Dieser Stamm trägt etwa 11 % seines Genoms auf sieben einheimischen Plasmiden (Rosso und Vary, 2005; Vary et al., 2007; Eppinger et al., 2011), von denen zwei – pBM500 und pBM600 – als Träger von Genen identifiziert wurden, die für die Bildung des charakteristischen „walnussförmigen“ Exosporiums dieses Stammes wesentlich sind (Manetsberger et al., 2015a). Die Proteinzusammensetzung des Exosporiums von QM B1551 ist bisher nur unzureichend charakterisiert, da nur wenige Gene, die für Orthologe anerkannter Exosporium-Proteine in Sporen anderer Arten kodieren, durch Genomanalysen identifiziert wurden. Dazu gehören Gene, die für BclA-Nap-Proteine kodieren, die in B. megaterium QM B1551-Sporen einen lokalisierten Nap bilden, sowie ein Ortholog des BxpB-Proteins, das in Sporen der B. cereus-Familie die Basalschicht des Exosporiums bildet. Letzteres scheint in B. megaterium QM B1551 Sporen eine andere Rolle zu spielen, da ein Null-Mutantenstamm ein scheinbar normales Exosporium behielt (Manetsberger et al, 2015a).
In dieser Arbeit präsentieren wir Daten, die zeigen, dass Sporen von B. megaterium QM B1551 effizient mRFP adsorbieren können, und liefern Beweise dafür, dass Proteinmoleküle in der Lage sind, die Permeabilitätsbarriere zu überwinden, die das Exosporium darstellt, um sich im Zwischenhüllenraum zu lokalisieren.
Materialien und Methoden
Bakterienstämme, Sporen und RFP-Produktion
Die in dieser Studie verwendeten B. megaterium-Stämme sind QM B1551 und sein plasmidloses Derivat PV361 (Rosso und Vary, 2005). Der in dieser Studie verwendete B. subtilis-Stamm war PY79 (Youngman et al., 1984). Die Sporulation aller Bacillus-Stämme wurde durch die Erschöpfungsmethode induziert (Cutting und Vander Horn, 1990). Nach 30 Stunden Wachstum in Difco-Sporulationsmedium (DS) bei 37°C und kräftigem Schütteln wurden die Sporen gesammelt und wie von Nicholson und Setlow (1990) beschrieben gereinigt, indem sie über Nacht in H2O bei 4°C inkubiert wurden, um die restlichen Sporangialzellen zu lysieren. Die Anzahl der erhaltenen gereinigten Sporen wurde durch direkte Zählung mit einer Bürker-Kammer unter einem optischen Mikroskop (Olympus BH-2 mit 40×-Objektiv) gemessen.
Für die mRFP-Produktion wurden Zellen des Escherichia coli-Stammes RH161 (Donadio et al., 2016), die den Expressionsvektor pRSET-A tragen, der eine In-Frame-Fusion des 5′-Endes der rfp-Kodierungsregion mit sechs Histidin-Kodons unter der transkriptionellen Kontrolle eines T7-Promotors trägt, wurden 18 Stunden lang bei 37 °C in 100 ml Autoinduktionsmedium kultiviert, um das heterologe Protein zu exprimieren (Isticato et al., 2010). Das His6-markierte RFP-Protein wurde unter nativen Bedingungen mit einer His-Trap-Säule gemäß den Empfehlungen des Herstellers (GE Healthcare Life Science) gereinigt. Gereinigtes Protein wurde mit einer PD10-Säule (GE Healthcare Life Science) entsalzt, um hohe NaCl- und Imidazol-Konzentrationen zu entfernen.
Adsorptionsreaktion
Wenn nicht anders angegeben, wurden 5 μg gereinigtes rekombinantes mRFP zu einer Suspension von Sporen (5 × 108) in 50 mM Natriumcitrat pH 4,0 bei 25°C in einem Endvolumen von 200 μl gegeben. Nach 1 h Inkubation wurde die Bindungsmischung zentrifugiert (10 min bei 13.000 g), um mRFP-gebundene Sporen im Pellet von freiem mRFP im Überstand zu trennen.
Western- und Dot-Blot-Analyse
Sporenpellets aus Adsorptionsreaktionen wurden in 40 μl Sporenmantel-Extraktionspuffer (Nicholson und Setlow, 1990; Giglio et al, 2011) resuspendiert, zur Solubilisierung der Sporenhüllenproteine 1 Stunde lang bei 68 °C inkubiert und auf ein 12 %iges SDS-PAGE-Gel geladen. Die Proteine wurden dann per Elektrotransfer auf Nitrocellulosefilter (Amersham Pharmacia Biotech) übertragen und für die Western-Blot-Analyse verwendet, wie zuvor berichtet (Isticato et al., 2008) unter Verwendung monoklonaler mRFP-erkennender Anti-His-Antikörper (Sigma). Eine quantitative Bestimmung der mRFP-Menge erfolgte durch Dot-Blot-Experimente, bei denen serielle Verdünnungen von gereinigtem mRFP und Überstand des Bindungstests verglichen wurden. Die Filter wurden anschließend mit der ECL-Prime-Methode (Amersham Pharmacia Biotech) visualisiert und mit der Quantity One 1-D Analysis Software (Bio-Rad) densitometrisch analysiert. Dot-Blot- und relative densitometrische Analysen wurden dreimal durchgeführt, um die Signifikanz der Ergebnisse zu überprüfen.
Fluoreszenz- und Immunfluoreszenzmikroskopie
Postadsorptionssporen wurden in 50 μl 1x PBS pH 4,0 resuspendiert und 5 μl der Suspension auf Objektträger gegeben und mit einem Deckglas abgedeckt, das zuvor 30 s lang mit Poly-L-Lysin (Sigma) behandelt wurde. Die Immunfluoreszenz wurde wie von Isticato et al. (2013) beschrieben durchgeführt, mit den folgenden Änderungen: 2,0 × 106 RFP-adsorbierte Sporen der B. megaterium-Stämme QM B1551 und PV361 wurden 30 Minuten lang mit 1% Rinderserumalbumin (BSA) – 1x PBS pH 4,0 vorbehandelt, bevor sie 2 Stunden lang bei 4°C mit den Anti-PolyHistidin-Antikörpern (Maus; Sigma), 1:20 verdünnt in 1x PBS pH 4,0-1% BSA, inkubiert wurden. Als Kontrolle für die Spezifität dieser Technik wurden nicht adsorbierte Sporen direkt mit Anti-His-Antikörpern behandelt. Nach dreimaligem Waschen wurden die Proben mit einem 64-fach verdünnten sekundären Anti-Maus-Antikörper inkubiert, der mit Fluoresceinisothiocyanat (FITC) (Santa Cruz Biotechnology, Inc.) konjugiert war, und viermal mit 1x PBS pH 4,0 gewaschen. Die gewaschenen Proben wurden in 20 μl 1x PBS pH 4,0 resuspendiert und 10 μl wurden analysiert. Alle Proben wurden mit einem Olympus BX51-Fluoreszenzmikroskop mit einem 100× UPlan F1-Ölobjektiv beobachtet; U-MNG- oder U-MWIBBP-Würfelfilter wurden verwendet, um die rote Fluoreszenzemission von mRFP bzw. die grüne Emission von FITC-konjugierten Antikörpern zu erkennen. Die Belichtungszeiten lagen im Bereich zwischen 200 und 3000 ms. Die Bilder wurden mit einer Olympus DP70 Digitalkamera aufgenommen und mit der Image Analysis Software (Olympus) für kleinere Anpassungen von Helligkeit, Kontrast und Farbbalance verarbeitet (McCloy et al., 2014). ImageJ (v1.48, NIH) wurde verwendet, um einen Umriss um 80 Sporen für jeden Stamm zu zeichnen, und die minimalen, maximalen und mittleren Fluoreszenzwerte pro Pixel wurden für jede Spore aufgezeichnet. Die Werte der Fluoreszenzintensität wurden anschließend als Box-Plots mit 5-95% Konfidenzintervallen dargestellt (McCloy et al., 2014).
Statistische Analyse
Die Ergebnisse der Dot-Blot- und Fluoreszenzmikroskopie-Analyse sind die Durchschnittswerte aus drei unabhängigen Experimenten. Die statistische Signifikanz wurde mit dem Student t-Test bestimmt, und das Signifikanzniveau wurde auf P < 0,05 festgelegt.
Ergebnisse
mRFP von Discosoma sp. wird von B. megaterium Spores
Um zu überprüfen, ob Sporen von B. megaterium QM B1551 in der Lage sind, mRFP zu adsorbieren, wurden 5 μg des gereinigten Proteins (Materialien und Methoden) mit 5,0 × 108 gereinigten Sporen inkubiert. Die Adsorptionsreaktion wurde in 50 mM Natriumcitrat bei pH 4,0 durchgeführt, wie zuvor beschrieben (Sirec et al., 2012). Nach der Reaktion wurden die Sporen ausgiebig mit 1x PBS pH 4,0 gewaschen, die Sporenoberflächenproteine wurden wie in Materialien und Methoden beschrieben extrahiert und durch Western Blotting mit anti-polyHistidin-Peroxidase-Antikörper (Sigma) analysiert, der den Histidin-markierten N-Terminus von mRFP erkennt. Wie in Abbildung 1A dargestellt, wurde mRFP aus Sporen extrahiert, was darauf hindeutet, dass es während der Reaktion absorbiert und dann durch die Extraktionsbehandlung freigesetzt wurde. Um die Effizienz der Adsorption zu bewerten, analysierten wir die Menge des ungebundenen mRFP, d. h. die Sporen wurden nach der Adsorption durch Zentrifugation gesammelt, der Überstand wurde seriell verdünnt und mittels Dot-Blotting analysiert (Abbildung 1B). Eine densitometrische Analyse des Dot-Blots (ergänzende Tabelle 1) zeigte, dass bei Verwendung von 5 μg mRFP in der Adsorptionsreaktion weniger als 1 % ungebunden blieb, was darauf hindeutet, dass etwa 99 % des heterologen Proteins an B. megaterium-Sporen adsorbiert wurden.

Abbildung 1. Bacillus megaterium QM B1551 Sporen adsorbieren mRFP. 5 × 108 Sporen wurden mit 5 μg gereinigtem mRFP inkubiert und die Proben anschließend zentrifugiert. (A) Sporenoberflächenproteine wurden durch SDS-DTT-Behandlung aus der Pelletfraktion extrahiert, durch SDS-PAGE fraktioniert und durch Western Blot analysiert. Aufgereinigtes mRFP (20 μg) wurde als Marker verwendet. (B) Der Überstand, der das ungebundene mRFP enthält, wurde seriell verdünnt und mittels Dot-Blot analysiert (QM B1551). Serielle Verdünnungen von gereinigtem mRFP (mRFP) wurden als Standard verwendet. (C) Mit mRFP adsorbierte Sporen wurden zweimal (W1 und W2) mit PBS-Puffer bei pH 3,0 oder pH 7,0 oder mit einer 1M NaCl, 0,1% Triton X-100-Lösung gewaschen. Serielle Verdünnungen von gereinigtem mRFP und ungebundenem mRFP (UN-mRFP) wurden als Standards verwendet. Immunreaktionen in allen Panels wurden mit mRFP-erkennenden Anti-His-Antikörpern durchgeführt, die mit Meerrettichperoxidase konjugiert waren.
Um zu analysieren, ob adsorbierte mRFP-Moleküle fest an die Sporenoberfläche gebunden waren, wurden die Sporen nach der Adsorptionsreaktion zweimal mit PBS-Puffer bei pH 3,0 oder pH 7,0 oder mit einer 1M NaCl, 0,1% Triton X-100-Lösung gewaschen, wie zuvor beschrieben (Donadio et al., 2016). Wie in Abbildung 1C gezeigt und durch die densitometrische Analyse des Dot-Blots (ergänzende Tabelle 2) unterstützt, verursachten die Waschungen bei pH 3,0 keine Freisetzung des adsorbierten mRFP, während die Waschungen bei pH 7,0 oder mit 1M NaCl, 0,1% Triton X-100 eine minimale, weniger als 1%, Freisetzung von mRFP-Molekülen verursachten. Die in Abbildung 1 dargestellten Ergebnisse deuten daher darauf hin, dass mRFP effizient adsorbiert und fest an B. megaterium-Sporen gebunden wurde. Um zu beurteilen, ob die an Sporen adsorbierten mRFP-Moleküle ihre Fluoreszenzeigenschaften beibehalten, führten wir eine fluoreszenzmikroskopische Analyse durch. Wie in Abbildung 2 dargestellt, waren die Sporen nach der Adsorptionsreaktion mit einem starken Fluoreszenzsignal verbunden, das um die gesamte Sporenoberfläche herum sichtbar war.
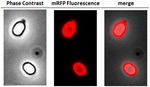
Abbildung 2. Fluoreszenzmikroskopische Analyse von B. megaterium QM B1551-mRFP Sporen. QM B1551-Sporen, die mit mRFP (5 μg) inkubiert und anschließend gewaschen wurden, wurden durch Fluoreszenzmikroskopie analysiert. Das gleiche Mikroskopiefeld wurde mit Phasenkontrast- und Fluoreszenzmikroskopie beobachtet. Maßstabsbalken 1 μm. Das Merge-Panel wird angezeigt. Die Belichtungszeit betrug 200 ms.
Das Exosporium spielt eine wesentliche Rolle bei der mRFP-Adsorption
Strain QM B1551 von B. megaterium enthält sieben einheimische Plasmide (Rosso und Vary, 2005; Eppinger et al., 2011), und die von den Plasmiden kodierten Gene sind für die Bildung des Exosporiums wesentlich (Manetsberger et al., 2015a). PV361 ist ein QM B1551-ausgehärteter Stamm, dem alle sieben Plasmide fehlen und dem folglich das Exosporium völlig fehlt (Manetsberger et al., 2015a). Wir verwendeten Sporen des Stammes PV361, um die Rolle des Exosporiums bei der mRFP-Adsorption zu analysieren. Parallel dazu verwendeten wir auch Sporen von B. subtilis PY79, von denen in einer früheren Studie gezeigt wurde, dass sie mRFP adsorbieren (Donadio et al., 2016). Um die Adsorptionseffizienz von Sporen der Stämme B. subtilis PY79 und B. megaterium QM B1551 und PV361 zu vergleichen, adsorbierten wir 5 μg gereinigtes mRFP mit 5,0 × 108 Sporen jedes der drei Stämme. Nach den Adsorptionsreaktionen wurden die Sporen durch Zentrifugation gesammelt, die Proteine durch SDS-DTT-Behandlung extrahiert und durch Western Blotting mit einem mRFP-erkennenden Anti-His-Antikörper analysiert. Wie in Abbildung 3A dargestellt, wurde mRFP offenbar in größeren Mengen aus Sporen von QM B1551 extrahiert als aus Sporen der beiden anderen Stämme. In einem Versuch, diese offensichtlichen Unterschiede zu quantifizieren, wurde ungebundenes mRFP aus den Adsorptionsreaktionen seriell verdünnt und mittels Dot-Blotting analysiert (Abbildung 3B). Eine densitometrische Analyse des Dot-Blots in Abbildung 3B (ergänzende Tabelle 3) zeigte, dass PY79- und PV361-Sporen etwa 90 % des gesamten mRFP adsorbierten, während QM B1551-Sporen fast das gesamte (über 99 %) gereinigte mRFP adsorbierten.
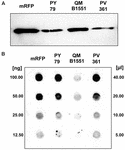
Abbildung 3. Adsorption von monomerem rot fluoreszierendem Protein an Sporen von B. megaterium QM B1551 und PV361 und B. subtilis PY79. 5 × 108 Sporen jedes Stammes wurden mit 5 μg gereinigtem mRFP inkubiert und die Proben anschließend zentrifugiert. Die Sporen in den Pelletfraktionen wurden zur Extraktion von Oberflächenproteinen verwendet, die anschließend mittels Western Blot analysiert wurden (A), während die Überstände seriell verdünnt und mittels Dot Blot analysiert wurden (B). Serielle Verdünnungen von gereinigtem mRFP wurden als Standards verwendet. Die Immunreaktionen in beiden Panels wurden mit Anti-His-Antikörpern durchgeführt, die mit Meerrettichperoxidase konjugiert waren.
Die Adsorptionseffizienz der Sporen der drei Stämme wurde auch durch Fluoreszenzmikroskopie analysiert (Abbildung 4A und ergänzende Abbildung S1). Die Mikroskopiebilder wurden mit der Software ImageJ (v1.48, NIH) analysiert, um eine quantitative Fluoreszenzbildanalyse durchzuführen, und die Sporenfluoreszenz wurde wie in Material und Methoden beschrieben berechnet. Die Analyse von 80 Sporen jedes Stammes ergab einen durchschnittlichen Fluoreszenzwert pro Pixel von 52,43 (± 5,94), 20,81 (± 2,71) und 32,33 (± 2.97; willkürliche Einheiten) für QM B1551, PV361 bzw. PY79 (Abbildung 4B), was ein weiterer Beweis dafür ist, dass die Sporen von QM B1551 mehr mRFP adsorbieren als die Sporen der anderen beiden Stämme.
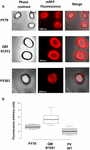
Abbildung 4. Effizienz der Adsorption von mRFP an Sporen von B. megaterium QM B1551 und PV361 und B. subtilis PY79. (A) Fluoreszenzmikroskopische Bilder von PY79-, QM B1551- und PV361-Sporen nach mRFP-Adsorption und Waschen. Die Belichtungszeiten sind angegeben. Phasenkontrast- und rote Fluoreszenzüberlagerungen werden gezeigt (Merge-Panel). Maßstabsbalken 1 μm. (B) Boxplots, die die Fluoreszenzintensität von achtzig verschiedenen Sporen jedes Stammes zeigen. Die Grenzen jedes Kästchens stellen das erste und das dritte Quartil (25 und 75 %) dar, die Werte außerhalb der Kästchen die Höchst- und Mindestwerte. Die Linie, die das Kästchen teilt, gibt den Medianwert für jeden Stamm an. Der P-Wert ist kleiner als 0,0001.
Aus den Ergebnissen der Abbildungen 3 und 4 schließen wir, dass das Exosporium, das in QM B1551 vorhanden ist und in PV361 fehlt, eine wichtige Rolle bei der Adsorption von mRFP spielt.
Darüber hinaus zeigen die Ergebnisse in Abbildung 4, dass die Sporen von B. subtilis PY79 bei der Adsorption von mRFP effizienter sind als die Sporen von B. megaterium PV361, während die in Abbildung 3B gezeigten Dot-Blotting-Untersuchungen für beide Stämme ähnliche Adsorptionseffizienzen ergeben. Wir glauben, dass diese Diskrepanz auf eine starke Verringerung der Fluoreszenz zurückzuführen ist, wenn mRFP an PV361, aber nicht an PY79 oder QM B1551-Sporen gebunden ist (siehe unten).
Quantitative Bewertung der mRFP-Adsorption an QM B1551-Sporen
Dot-Blot-Experimente (Abbildung 3B) zeigten, dass bei Verwendung von 5 μg gereinigtem mRFP in Adsorptionsreaktionen mit 5,0 × 108 Sporen des QM B1551-Stammes fast das gesamte heterologe Protein an die Spore gebunden wurde. Um die maximale Menge an mRFP zu bestimmen, die an QM B1551-Sporen adsorbiert werden kann, wiederholten wir die Reaktionen mit steigenden Konzentrationen von gereinigtem mRFP, d. h. 5,0 × 108 QM B1551-Sporen wurden mit 5, 10, 20, 40, 80 und 160 μg gereinigtem mRFP umgesetzt. Nach den Reaktionen wurden die Sporen durch Zentrifugation gesammelt, und die Überstände, die ungebundenes mRFP enthielten, wurden seriell verdünnt und mittels Dot-Blotting analysiert (Abbildung 5A). Abbildung 5B zeigt die Ergebnisse einer densitometrischen Analyse des Dot-Blots (??), aus der hervorgeht, dass bei der Reaktion von 5-80 μg mRFP mit 5 × 108 Sporen der Prozentsatz des an die Sporen gebundenen Proteins über 90 % betrug. Ein Rückgang des gebundenen mRFP wurde beobachtet, wenn 160 μg des gereinigten Proteins in der Reaktion verwendet wurden. Doch selbst bei Verwendung von 160 μg gereinigtem mRFP wurden über 60 % des Proteins absorbiert, was darauf hindeutet, dass 5,0 × 108 Sporen von QM B1551 etwa 100 μg mRFP adsorbieren können.
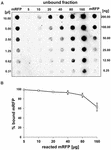
Abbildung 5. Quantitative Bewertung der Adsorption von mRFP an B. megaterium QM B1551 Sporen. 5 × 108 Sporen wurden mit 5, 10, 20, 40, 80 und 160 μg des gereinigten mRFP inkubiert. Die Reaktionsgemische wurden anschließend zentrifugiert und die Überstände seriell verdünnt und mittels Dot-Blot analysiert (A). Serielle Verdünnungen von gereinigtem mRFP wurden als Standards verwendet. Die Immunreaktionen in beiden Panels wurden mit Anti-His-Antikörpern durchgeführt, die mit Meerrettichperoxidase konjugiert waren. (B) Prozentualer Anteil des an Sporen adsorbierten mRFP nach Reaktion mit bestimmten Mengen endogenen mRFP. Die Fehlerbalken zeigen die Standardfehler des Mittelwerts aus drei Experimenten, wobei der P-Wert nie über 0,0025 liegt.
mRFP lokalisiert sich im Zwischenhüllenraum von B. megaterium QM B1551 Sporen
Mit Hilfe der Immunfluoreszenzmikroskopie wurde untersucht, ob adsorbierte mRFP-Moleküle auf der Oberfläche von B. megaterium QM B1551 Sporen exponiert waren. QM B1551-Sporen, die mit verschiedenen Mengen an mRFP adsorbiert waren, wurden mit einem monoklonalen Anti-His-Antikörper, der das rekombinante mRFP erkennt, und anschließend mit einem fluoreszierenden Anti-Maus-Sekundärantikörper (Santa Cruz Biotechnology, Inc.) zur Reaktion gebracht und unter dem Fluoreszenzmikroskop beobachtet (Abbildung 6). Bei der geringsten in diesem Experiment verwendeten mRFP-Menge (2 μg) wurde das mRFP-Fluoreszenzsignal (rot) rund um die Spore beobachtet, während das Immunfluoreszenzsignal (grün) schwach und hauptsächlich an den Sporenpolen konzentriert war, was darauf hindeutet, dass nur an diesen Stellen mRFP auf der Sporenoberfläche exponiert war. Mit zunehmender Menge des in der Reaktion verwendeten mRFP nahm die Anzahl der grünen Flecken zu (5 und 10 μg), und bei der höchsten in der Reaktion verwendeten mRFP-Menge (20 μg) wurde ein fast vollständiger grüner Ring um die Sporen beobachtet. Auf der Grundlage der in Abbildung 6 dargestellten Ergebnisse stellten wir die Hypothese auf, dass mRFP-Moleküle durch das Exosporium eindringen und sich im Zwischenraum zwischen der äußeren Hülle und dem Exosporium ansiedeln, d. h., wenn eine geringe Menge mRFP verwendet wird, befinden sich fast alle Proteinmoleküle innerhalb des Exosporiums und sind für den Antikörper nur an wenigen Stellen verfügbar. Eine Erhöhung der mRFP-Menge bei Adsorptionsreaktionen führt dazu, dass sich der Zwischenschichtraum „füllt“, bis schließlich mehr mRFP-Moleküle für den Antikörper auf der Sporenoberfläche verfügbar sind. Diese Hypothese impliziert, dass bei fehlendem Exosporium das gesamte mRFP für den Antikörper verfügbar sein sollte. Um dies zu prüfen, verglichen wir mittels Immunfluoreszenzmikroskopie die gleiche Anzahl von Sporen von QM B1551 (mit Exosporium) und von PV361 (ohne Exosporium), die mit der gleichen Menge mRFP (5 μg) inkubiert wurden. Wenn das Exosporium vorhanden war (QM B1551), war mRFP nur teilweise für den Antikörper verfügbar und es wurden grüne Flecken beobachtet (Abbildung 7 und ergänzende Abbildung S2). Wenn das Exosporium nicht vorhanden war (PV361), war das adsorbierte mRFP für den Antikörper rund um die Spore verfügbar, und es bildete sich ein vollständiger grüner Ring, was die Hypothese stützt, dass mRFP in QM B1551-Sporen innerhalb des Exosporiums liegt.
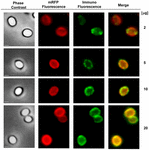
ABBILD 6: Immunfluoreszenzanalyse von mRFP-adsorbierten B. megaterium QM B1551-Sporen. Aliquots von 5 × 108 QM B1551-Sporen wurden mit unterschiedlichen Konzentrationen von mRFP inkubiert und anschließend mittels Phasenkontrast-, Fluoreszenz- und Immunfluoreszenzmikroskopie analysiert, wie in den Materialien und Methoden beschrieben. Das gleiche Mikroskopiefeld für jede Reaktion wird zusammen mit dem Merge-Panel dargestellt. Die Belichtungszeit betrug für alle Bilder 200 ms. Maßstabsbalken, 1 μm.
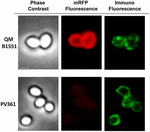
Abbildung 7. Immunfluoreszenz von mRFP adsorbiert an B. megaterium QM B1551 und PV361 Sporen. 5 × 108 QM B1551- und PV361-Sporen wurden mit 5 μg mRFP inkubiert und dann durch Immunfluoreszenzmikroskopie analysiert, wie in den Materialien und Methoden beschrieben. Für jedes Feld sind Phasenkontrast und Immunfluoreszenzmikroskopie dargestellt. Die Belichtungszeit betrug für alle Bilder 200 ms. Maßstabsbalken, 1 μm.
Während die in den Experimenten von Abbildung 7 verwendeten QM B1551-Sporen einen vollständigen rot fluoreszierenden Ring wie in Abbildung 2 zeigten, wiesen die PV361-Sporen ein sehr schwaches rot fluoreszierendes Signal auf. Bei PV361-Sporen wurde ein rotes Signal nur bei langen Belichtungszeiten am Fluoreszenzmikroskop beobachtet (Abbildung 4). Da mRFP in der Umgebung von PV361-Sporen vorhanden ist (Abbildungen 3 und 7), schließen wir daraus, dass die mRFP-Fluoreszenz geschwächt ist, wenn das Protein an PV361-Sporen adsorbiert wird. Weitere Experimente sind erforderlich, um diesen Punkt vollständig zu klären.
Diskussion
Die wichtigsten Ergebnisse dieses Berichts sind, dass Sporen von B. megaterium das heterologe Modellprotein mRFP äußerst effizient adsorbieren, dass das Exosporium bei diesem Prozess eine wichtige Rolle spielt und dass mRFP-Moleküle durch das Exosporium eindringen und zwischen der äußeren Hülle und dem Exosporium lokalisiert werden. Diese Ergebnisse erweitern frühere Arbeiten, die mit Sporen von B. subtilis durchgeführt wurden, und zeigen, dass auch Sporen einer anderen Spezies für die Übertragung heterologer Proteine durch die Adsorptionsmethode verwendet werden können. Die hohe Adsorptionseffizienz, die mit B. megaterium-Sporen beobachtet wurde, ist zum Teil auf die große Größe der Sporen im Vergleich zu B. subtilis zurückzuführen. In der Tat ist die Oberfläche der B. megaterium-Sporen etwa 2,5-mal größer als die der B. subtilis-Sporen, mit einer Oberfläche von 5,33 μm2 (h: 1,60 ± 0,16 w: 0,84 ± 0,07) gegenüber 1,98 μm2 (h: 1,07 ± 0,09 w: 0,48 ± 0,03). Die großen Abmessungen ermöglichen die Adsorption von bis zu 100 μg mRFP, wenn 160 μg Protein mit Sporen umgesetzt werden.
Die Beobachtung, dass mRFP das Exosporium durchquert, deutet darauf hin, dass es für mRFP, ein 27 kDa Protein, durchlässig ist. Die Durchlässigkeit des Exosporiums ist nicht völlig überraschend, da die in der Umgebung vorhandenen Keimstoffe die äußeren Schichten der Spore durchqueren müssen, um ihre Rezeptoren zu erreichen, auch wenn es sich bei den Keimstoffen in der Regel um kleine Moleküle mit Molekularmassen von typischerweise <200 Da handelt. Außerdem stimmen die mRFP-Daten weitgehend mit den Ergebnissen früherer Studien überein, die mit markierten Dextranen und verwandten Molekülen durchgeführt wurden (Koshikawa et al., 1984; Nishihara et al., 1989). In diesen Studien wurde angenommen, dass das Exosporium von B. megaterium QM B1551 eine Permeabilitätsbarriere für Moleküle mit einem Molekulargewicht von mehr als 100 KDa darstellt, während es die Passage von Molekülen mit einer Masse zwischen 2 und 40 kDa beeinflusst (Koshikawa et al., 1984; Nishihara et al., 1989).
Eine interessante Herausforderung für künftige Arbeiten wird es sein, den Mechanismus oder den Weg der Infiltration zu bestimmen, den mRFP und folglich auch andere heterologe Proteine von Interesse nehmen, um in den Zwischenschichtraum einzudringen. Die Untersuchung von geschnittenen B. megaterium QM B1551 Sporen mittels Transmissionselektronenmikroskopie zeigt, dass das Exosporium aus zwei identischen „Schalen“ besteht (Manetsberger et al., 2015a), und es könnte sein, dass die Grenzfläche zwischen jeder dieser Strukturen (in früheren Arbeiten als „apikale Öffnungen“ beschrieben) das Eindringen relativ großer Moleküle ermöglicht. Es wird auch von Interesse sein, die Grundlage für den offensichtlichen Verlust der mRFP-Fluoreszenz bei der Adsorption an PV361-Sporen zu ermitteln und ob mRFP-Moleküle in der Lage sind, die äußeren Hüllschichten zu infiltrieren, wie dies bei B. subtilis-Sporen beobachtet wurde (Donadio et al., 2016).
In der aktuellen Studie stellen wir die Hypothese auf, dass mRFP-Moleküle bevorzugt das Exosporium durchqueren und sich im Zwischenhüllraum zwischen der äußeren Hülle und dem Exosporium ansammeln. In diesem Modell werden mRFP-Moleküle nur dann adsorbiert und auf der Sporenoberfläche angezeigt, wenn die Adsorptionsstellen (oder die volumetrische Kapazität?) im Zwischenhüllenraum ausreichend besetzt sind. Dies bedeutet, dass der Adsorptionsansatz für die Oberflächendarstellung mit B. megaterium QM B1551 Sporen verwendet werden kann, obwohl das System vom Verhältnis zwischen Sporen und Proteinen bei Adsorptionsreaktionen abhängig ist. Da verschiedene Stämme von B. megaterium seit langem industriell für die Produktion von Enzymen wie Amylasen und Dehydrogenasen, Vitaminen und antimikrobiellen Molekülen verwendet werden (Vary et al., 2007), deuten unsere Daten auf eine neue biotechnologische Anwendung für die B. megaterium-Spore als Vehikel zur Bindung und Abgabe heterologer Proteine hin.
Beiträge der Autoren
ML und GD – führten die meisten Experimente durch; RA und AS – trugen zur Interpretation der Daten für die Arbeit bei; GiC, LB und GrC – trugen zum Entwurf der Arbeit bei und überarbeiteten sie kritisch; ER – trug zu Diskussionen und Vorschlägen während der Arbeit bei und schrieb das Manuskript mit; RI – leitete die Arbeit und schrieb das Manuskript mit. Alle Autoren lasen und genehmigten das endgültige Manuskript.
Finanzierung
Diese Arbeit wurde durch ein Stipendium (STAR Linea1- 2014) der Federico II Universität Neapel und der Compagnia di San Paolo, Italien an RI unterstützt.
Erklärung zu Interessenkonflikten
Die Autoren erklären, dass die Forschung in Abwesenheit jeglicher kommerzieller oder finanzieller Beziehungen durchgeführt wurde, die als potenzieller Interessenkonflikt ausgelegt werden könnten.
Ergänzendes Material
Das ergänzende Material zu diesem Artikel finden Sie online unter: https://www.frontiersin.org/article/10.3389/fmicb.2016.01752/full#supplementary-material
ABBILD S1 | Ganzfeldbilder der in Abbildung 4 dargestellten Fluoreszenzmikroskopie-Analyse. Das gleiche Mikroskopiefeld wurde mit Phasenkontrast- und Fluoreszenzmikroskopie beobachtet. Maßstabsbalken 5 μm. Das Merge-Panel wird angezeigt. Die Belichtungszeit ist angegeben.
Abbildung S2 | Ganzfeldbilder der in Abbildung 7 dargestellten Immunfluoreszenzanalyse. Das gleiche Mikroskopiefeld wurde mit Phasenkontrast- und Fluoreszenzmikroskopie beobachtet. Maßstabsbalken 5 μm. Die Belichtungszeit betrug 200 ms für alle Bilder.
TABELLE S1 | Densitometrische Analyse der Dot-Blot-Experimente mit den Überständen der Adsorptionsreaktion mit QM B1551-Sporen (Abbildung 1B).
TABLE S2 | Densitometrische Analyse von Dot-Blot-Experimenten mit den Überständen verschiedener Waschungen nach der Adsorptionsreaktion (Abbildung 1C).
TABLE S3 | Densitometrische Analyse von Dot-Blot-Experimenten mit den Überständen der Adsorptionsreaktion, die mit Sporen der Stämme PY79, QM B1551 und PV361 durchgeführt wurden (Abbildung 3B).
TABLE S4 | Densitometrische Analyse von Dot-Blot-Experimenten mit den Überständen der Adsorptionsreaktion, die mit verschiedenen Mengen von mRFP durchgeführt wurden.
Baccigalupi, L., Ricca, E., and Ghelardi, E. (2015). „Non-LAB probiotics: spore Formers,“ in Probiotics and Prebiotics: Current Research and Future Trends, eds K. Venema and A. P. do Carmo (Norfolk, VA: Caister Academic Press), 93-103. doi: 10.21775/9781910190098.06
CrossRef Full Text | Google Scholar
Campbell, R. E., Tour, O., Palmer, A. E., Steinbach, P. A., Baird, G. S., Zacharias, D. A., et al. (2002). Ein monomeres rot fluoreszierendes Protein. Proc. Natl. Acad. Sci. U.S.A. 99, 7877-7882. doi: 10.1073/pnas.082243699
PubMed Abstract | CrossRef Full Text | Google Scholar
Cangiano, G., Sirec, T., Panarella, C., Isticato, R., Baccigalupi, L., De Felice, M., et al. (2014). Die sps-Genprodukte beeinflussen die Keimung, Hydrophobizität und Proteinadsorption von Bacillus subtilis-Sporen. Appl. Environ. Microbiol. 80, 7293-7302. doi: 10.1128/AEM.02893-14
PubMed Abstract | CrossRef Full Text | Google Scholar
Cutting, S. M. (2011). Bacillus probiotics. Food Microbiol. 28, 214-220. doi: 10.1016/j.fm.2010.03.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Cutting, S., and Vander Horn, P. B. (1990). „Genetic analysis,“ in Molecular Biological Methods for Bacillus, eds C. Harwood and S. Cutting (Chichester: John Wiley and Sons), 27-74.
Google Scholar
Díaz-González, F., Milano, M., Olguin-Araneda, V., Pizarro-Cerda, J., Castro-Córdova, P., Tzeng, S. C., et al. (2015). Proteinzusammensetzung der äußersten exosporiumartigen Schicht von Clostridium difficile 630 Sporen. J. Proteom. 123, 1-13. doi: 10.1016/j.jprot.2015.03.035
PubMed Abstract | CrossRef Full Text | Google Scholar
Di Luccia, B., D’Apuzzo, E., Varriale, F., Baccigalupi, L., Ricca, E., and Pollice, A. (2016). Bacillus megaterium SF185 induziert Stresswege und beeinflusst die Zellzyklusverteilung von menschlichen Darmepithelzellen. Benef. Microb. 7, 609-620. doi: 10.3920/BM2016.0020
PubMed Abstract | CrossRef Full Text | Google Scholar
Donadio, G., Lanzilli, M., Sirec, T., Ricca, E., and Isticato, R. (2016) Protein adsorption and localization on wild type and mutant spores of Bacillus subtilis. Microb. Cell Fact. 15:153. doi: 10.1186/s12934-016-0551-2
PubMed Abstract | CrossRef Full Text
Eppinger, M., Bunk, B., Johns, M. A., Edirisinghe, J. N., Kutumbaka, K. K., Koenig, S. S., et al. (2011). Genomsequenzen der biotechnologisch wichtigen Bacillus megaterium-Stämme QM B1551 und DSM319. J. Bacteriol. 193, 4199-4213. doi: 10.1128/JB.00449-11
PubMed Abstract | CrossRef Full Text | Google Scholar
Giglio, R., Fani, R., Isticato, R., De Felice, M., Ricca, E., and Baccigalupi, L. (2011). Organisation und Evolution der cotG- und cotH-Gene von Bacillus subtilis. J. Bacteriol. 193, 6664-6673. doi: 10.1128/jb.06121-11
PubMed Abstract | CrossRef Full Text | Google Scholar
Harwood, C. R., and Cutting, S. M. (1990). Molekularbiologische Methoden für Bacillus. Chichester: John Wiley and Sons.
Google Scholar
Huang, J. M., Hong, H. A., Van Tong, H., Hoang, T. H., Brisson, A., and Cutting, S. M. (2010). Mucosal delivery of antigens using adsorption to bacterial spores. Vaccine 28, 1021-1030. doi: 10.1016/j.vaccine.2009.10.127
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., Pelosi, A., De Felice, M., and Ricca, E. (2010). CotE bindet an CotC und CotU und vermittelt deren Interaktion während der Sporenhüllenbildung in Bacillus subtilis. J. Bacteriol. 192, 949-954. doi: 10.1128/JB.01408-09
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., Pelosi, A., Zilhão, R., Baccigalupi, L., Henriques, A. O., De Felice, M., et al. (2008). CotC-CotU-Heterodimerisierung während des Aufbaus der Sporenhülle von Bacillus subtilis. J. Bacteriol. 190, 1267-1275. doi: 10.1128/JB.01425-07
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., and Ricca, E. (2014). Spore surface display. Microbiol. Spectr. 2, 351-366. doi: 10.1128/microbiolspec.TBS-0011-2012
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., Sirec, T., Treppiccione, L., Maurano, F., De Felice, M., Rossi, M., et al. (2013). Nonrecombinant display of the B subunit of the heat labile toxin of Escherichia coli on wild type and mutant spores of Bacillus subtilis. Microb. Cell Fact. 12:98. doi: 10.1186/1475-2859-12-98
PubMed Abstract | CrossRef Full Text | Google Scholar
Knecht, L. D., Pasini, P., and Daunert, S. (2011). Bakterielle Sporen als Plattformen für bioanalytische und biomedizinische Anwendungen. Anal. Bioanal. Chem. 400, 977-989. doi: 10.1007/s00216-011-4835-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Koshikawa, T., Beaman, T. C., Pankratz, H. S., Nakashio, S., Corner, T. R., and Gerhardt, P. (1984). Resistenz, Keimung und Permeabilitätskorrelate von Bacillus megaterium Sporen, die nacheinander von Integumentschichten befreit wurden. J. Bacteriol. 159, 624-632.
PubMed Abstract | Google Scholar
Manetsberger, J., Hall, E. A. H., and Christie, G. (2015a). Plasmid-kodierte Gene beeinflussen die Exosporium-Assemblierung und Morphologie in Bacillus megaterium QM B1551 Sporen. FEMS Microbiol. Lett. 362:fnv147. doi: 10.1093/femsle/fnv147
PubMed Abstract | CrossRef Full Text | Google Scholar
Manetsberger, J., Manton, J. D., Erdelyi, M. J., Lin, H., Rees, D., Christie, G., et al. (2015b). Ellipsoid-Lokalisierungsmikroskopie gibt Aufschluss über die Größe und Anordnung von Proteinschichten in Bacillus-Sporenhüllen. Biophys. J. 109, 2058-2066. doi: 10.1016/j.bpj.2015.09.023
PubMed Abstract | CrossRef Full Text | Google Scholar
McCloy, R., Rogers, S., Caldon, E., Lorca, T., Castro, A., and Burgess, A. (2014). Partielle Hemmung von Cdk1 in der G2-Phase hebt die SAC auf und entkoppelt mitotische Ereignisse. Cell Cycle 13, 1400-1412. doi: 10.4161/cc.28401
PubMed Abstract | CrossRef Full Text | Google Scholar
McKenney, P. T., Driks, A., and Eichemberger, P. (2012). Die Endospore von Bacillus subtilis: Aufbau und Funktionen der mehrschichtigen Hülle. Nat. Rev. Microbiol. 11, 33-44. doi: 10.1038/nrmicro2921
PubMed Abstract | CrossRef Full Text | Google Scholar
Nicholson, W. L., and Setlow, P. (1990). „Sporulation, germination and out-growth,“ in Molecular biological methods for Bacillus, eds C. Harwood and S. Cutting (Chichester: John Wiley and Sons), 391-450.
Google Scholar
Nishihara, T., Takubo, Y., Kawamata, E., Koshikawa, T., Ogaki, J., and Kondo, M. (1989). Die Rolle der äußeren Hülle bei der Resistenz von Bacillus megaterium Sporen. J. Biochem. 106, 270-273.
PubMed Abstract | Google Scholar
Pesce, G., Rusciano, G., Sirec, T., Isticato, R., Sasso, A., and Ricca, E. (2014). Surface charge and hydrodynamic coefficient measurements of Bacillus subtilis spore by optical tweezers. Colloids Surf. B Biointerfaces 116C, 568-575. doi: 10.1016/j.colsurfb.2014.01.039
PubMed Abstract | CrossRef Full Text | Google Scholar
Ricca, E., Baccigalupi, L., Cangiano, G., De Felice, M., and Isticato, R. (2014). Mucosal vaccine delivery by non-recombinant spores of Bacillus subtilis. Microb. Cell Fact. 13, 115. doi: 10.1186/s12934-014-0115-2
PubMed Abstract | CrossRef Full Text | Google Scholar
Rosso, M. L., and Vary, P. S. (2005). Verteilung von Bacillus megaterium QM B1551 Plasmiden unter anderen B. megaterium Stämmen und Bacillus Arten. Plasmid 53, 205-217. doi: 10.1016/j.plasmid.2004.10.005
PubMed Abstract |Ref Full Text | Google Scholar
Rusciano, G. Zito, G., Isticato, R., Sirec, T., Ricca, E., Bailo, E., und Sasso, A. (2014). Chemische Bildgebung im Nanobereich von Bacillus subtilis-Sporen durch die Kombination von spitzenverstärkter Raman-Streuung und fortschrittlichen statistischen Tools. ACS Nano 8, 12300-12309. doi: 10.1021/nn504595k
PubMed Abstract | CrossRef Full Text | Google Scholar
Sirec, T., Strazzulli, A., Isticato, R., De Felice, M., Moracci, M., and Ricca, E. (2012). Adsorption von Beta-Galactosidase von Alicyclobacillus acidocaldarius an Wildtyp- und Mutantensporen von Bacillus subtilis. Microb. Cell Fact. 11:100. doi: 10.1186/1475-2859-11-100
PubMed Abstract | CrossRef Full Text | Google Scholar
Stewart, G. C. (2015). Die Exosporium-Schicht von bakteriellen Sporen: eine Verbindung zur Umwelt und zum infizierten Wirt. Microbiol. Mol. Biol. Rev. 79, 437-457. doi: 10.1128/MMBR.00050-15
PubMed Abstract | CrossRef Full Text | Google Scholar
Vary, P. S., Biedendieck, R., Fuerch, T., Meinhardt, F., Rohde, M., Deckwer, W.-D., et al. (2007). Bacillus megaterium – vom einfachen Bodenbakterium zum Wirt für die industrielle Proteinproduktion. Appl. Microbiol. Biotechnol. 76, 957-967 doi: 10.1007/s00253-007-1089-3
PubMed Abstract | CrossRef Full Text | Google Scholar
Youngman, P., Perkins, J. B., and Losick, R. (1984). Eine neue Methode zur schnellen Klonierung der chromosomalen DNA von Bacillus subtilis in Escherichia coli in der Nähe der Insertion von Tn917. Mol. Gen. Genet. 195, 424-433. doi: 10.1007/BF00341443
CrossRef Full Text | Google Scholar