Allgemeine Einführung
Die meisten von uns wissen, dass Hefe ein sehr nützlicher Organismus ist, vor allem beim Backen, bei der Weinherstellung und beim Brauen. Aber was sind Hefen und warum stehen sie im Mittelpunkt so vieler Forschungen?

Hefen sind Pilze
Hefen sind einzellige Mikroorganismen, die zusammen mit Schimmelpilzen und Pilzen dem Königreich der Pilze zugeordnet werden. Hefen sind evolutionär vielfältig und werden daher in zwei getrennte Phyla eingeteilt, Ascomycota oder Schlauchpilze und Basidiomycota oder höhere Pilze, die zusammen das Unterkönigreich Dikarya bilden. Knospenhefen, auch „echte Hefen“ genannt, gehören zum Stamm der Ascomycota und zur Ordnung der Saccharomycetales. Solche Klassifizierungen basieren auf Merkmalen der Zelle, der Ascospore und der Kolonie sowie der Zellphysiologie.
Hefen sind einzellig, aber mit einer Zellorganisation, die höheren Organismen ähnelt
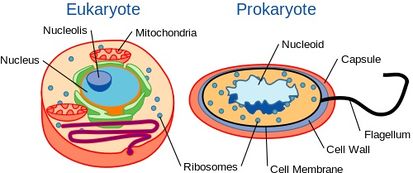
Obwohl Hefen Einzeller sind, besitzen sie eine ähnliche zelluläre Organisation wie höhere Organismen, einschließlich des Menschen. Insbesondere ist ihr genetischer Inhalt in einem Zellkern enthalten. Dadurch werden sie als eukaryotische Organismen eingestuft, im Gegensatz zu ihren einzelligen Gegenstücken, den Bakterien, die keinen Zellkern haben und als Prokaryoten gelten.
Natürliche Lebensräume
Hefen sind in der Natur weit verbreitet und haben eine Vielzahl von Lebensräumen. Man findet sie häufig auf Pflanzenblättern, Blüten und Früchten sowie im Boden. Hefen finden sich auch auf der Hautoberfläche und im Darmtrakt von Warmblütern, wo sie in Symbiose oder als Parasiten leben können. Die weit verbreitete „Hefeinfektion“ wird in der Regel durch Candida albicans verursacht. Candida ist nicht nur der Erreger vaginaler Hefeinfektionen, sondern auch die Ursache von Windelausschlag und Soor in Mund und Rachen.
Warum Hefe untersuchen?
Stellen Sie sich einen Organismus vor, der schnell in einem Kolben wächst und dessen DNA leicht manipuliert werden kann, der aber auch Einblicke in grundlegende biologische Prozesse des Menschen, einschließlich Krankheiten, bietet. Hefe passt auf diese Beschreibung und steht im Mittelpunkt des Interesses von Forschern auf der ganzen Welt, was zu mehr als 50.000 veröffentlichten wissenschaftlichen Artikeln geführt hat, in denen die Hefeforschung beschrieben wird!

Welche spezifischen Eigenschaften der Hefe machen sie zu einem „Modellorganismus“ für Studien und zum Mittelpunkt so vieler Forschungen? Hefe ist ein einzelliger Organismus, der einfach zu erforschen ist, aber eine ähnliche zelluläre Organisation aufweist wie höhere, mehrzellige Organismen wie der Mensch, d.h. sie besitzt einen Zellkern und ist daher ein Eukaryonten, wie oben beschrieben. Am wichtigsten ist, dass die Ähnlichkeit in der zellulären Organisation zwischen Hefe und höheren Eukaryoten zu Ähnlichkeiten in ihren grundlegenden zellulären Prozessen führt, so dass Entdeckungen in Hefe häufig direkte oder indirekte Hinweise darauf liefern, wie biologische Prozesse beim Menschen funktionieren.

Eine weitere wichtige Eigenschaft der Hefe, die für ihre Rolle als „Modellorganismus“ wesentlich ist, ist die Tatsache, dass sie relativ einfach zu bearbeiten ist. Hefen vermehren sich schnell und sind genetisch leicht zu manipulieren. Die Verdopplungszeit für Hefe (die Zeit, die eine Zelle benötigt, um sich zu verdoppeln und zu teilen) beträgt etwa 90 Minuten. Im Gegensatz dazu benötigen menschliche Zellen, die in Kulturen wachsen, etwa 24 Stunden, um sich zu verdoppeln. Es gibt auch gut definierte genetische Methoden für Hefe, die es den Forschern ermöglichen, auf einfache Weise Mutanten zu isolieren, sie mit anderen Mutanten oder mit anderen genetischen Hintergründen zu kreuzen und die Positionen von Genen zu kartieren. Genetische Karten, die auf der Grundlage des genetischen Abstands zwischen den Genen erstellt wurden, gaben den Forschern einen ersten Einblick in das Genom und seine Organisation und waren der Höhepunkt genetischer Studien, die bis in die erste Hälfte des 20. Jahrhunderts zurückreichen.
Eine Beschleunigung der Entdeckungen wurde möglich, nachdem das Genom der Bäckerhefe (S. cerevisiae), das ihr gesamtes genetisches Material enthält, 1996 als erstes eukaryontisches Genom sequenziert wurde. Es ist kleiner und kompakter als das menschliche Genom (12 Millionen Basenpaare und ~6.000 Gene, verglichen mit 3 Milliarden Basenpaaren und ~20-25.000 proteinkodierenden Genen). Dennoch zeigen Vergleiche der Genome, dass ~31 % der Hefegene menschlichen Genen sehr ähnlich sind und 20 % der menschlichen Krankheitsgene Entsprechungen in der Hefe haben. Außerdem können Hefezellen entweder als Haploide (ein Chromosomensatz) oder als Diploide (zwei Chromosomensätze) existieren. Da Haploide nur eine Kopie jedes Gens und eine effiziente Trennung und Wiedervereinigung von DNA-Strängen (Rekombination) haben, ist es sehr einfach, ein bestimmtes Gen in einem Haploiden zu löschen und die Auswirkungen auf die Zelle oder den „Phänotyp“ der Deletionsmutante zu beobachten. Diploide Zellen hingegen ermöglichen die Untersuchung essenzieller Gene (die für das Wachstum und die Lebensfähigkeit erforderlich sind), indem man eine Kopie des Gens löscht und in der anderen Kopie subtile Veränderungen vornimmt. Schließlich wurde mit den Informationen aus der Genomsequenz ein umfangreiches Instrumentarium an molekularen Reagenzien und genomweiten Sammlungen aufgebaut, das den Forschern leistungsfähige Mittel zur Untersuchung biologischer Probleme an die Hand gibt. Wenn bekannt ist, dass ein Hefegen in der DNA-Sequenz einem menschlichen Gen ähnelt, können Studien in Hefe wichtige Hinweise auf die Rolle des/der verwandten Gens/Gene beim Menschen liefern. Die relative Einfachheit der Untersuchung zellulärer Funktionen in der Hefe in Verbindung mit ihrer Relevanz für höhere Organismen macht sie daher zu einem sehr leistungsfähigen „Modellorganismus“ für Studien.
Leben und Zellzyklen der Hefe
Hefen wachsen normalerweise ungeschlechtlich durch Knospung. An der Mutterzelle bildet sich eine kleine Knospe, aus der sich die Tochterzelle entwickelt, die sich mit fortschreitendem Wachstum vergrößert. Während die Tochterzelle wächst, dupliziert sich die Mutterzelle und teilt dann ihre DNA. Der Zellkern teilt sich und wandert in die Tochterzelle ein. Sobald die Knospe einen Zellkern enthält und eine bestimmte Größe erreicht hat, trennt sie sich von der Mutterzelle. Die Abfolge der Ereignisse, die in einer Zelle stattfinden und zur Verdopplung und Teilung führen, wird als Zellzyklus bezeichnet. Der Zellzyklus besteht aus vier verschiedenen Phasen (G1, S, G2 und M) und wird ähnlich wie der Zellzyklus bei größeren Eukaryoten reguliert. Solange ausreichende Nährstoffe wie Zucker, Stickstoff und Phosphat vorhanden sind, teilen sich die Hefezellen ungeschlechtlich weiter.
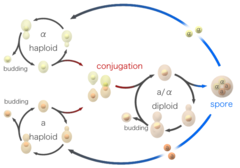
Hefezellen können sich auch geschlechtlich vermehren. Hefezellen existieren als einer von zwei verschiedenen Paarungstypen, a-Zellen und alpha-Zellen. Wenn Zellen entgegengesetzter Paarungstypen im Labor zusammengemischt werden oder in der Natur zufällig in Kontakt kommen, können sie sich paaren (konjugieren). Vor der Vereinigung verändern die Zellen ihre Form in einem Prozess, der „shmooing“ genannt wird. Der Begriff „shmoo“ wurde aufgrund der Ähnlichkeit der Form mit einer fiktiven Comicfigur gleichen Namens geprägt, die in den späten 40er Jahren von Al Capp geschaffen wurde und zuerst in seinem Comicstrip L’il Abner auftauchte. Bei der Konjugation verschmelzen zunächst die beiden haploiden Zellen und dann ihre Zellkerne, so dass eine diploide Zelle mit zwei Kopien von jedem Chromosom entsteht. Einmal gebildet, können sich diploide Zellen durch Knospung ungeschlechtlich vermehren, ähnlich wie Haploide. Wenn diploide Zellen jedoch keine Nährstoffe mehr erhalten, kommt es zur Sporulation. Während der Sporulation durchlaufen diploide Zellen die Meiose, eine besondere Form der Zellteilung, bei der die Anzahl der Chromosomen von zwei Kopien wieder auf eine Kopie reduziert wird. Nach der Meiose werden die in der Meiose entstandenen haploiden Kerne in vier Sporen verpackt, die veränderte Zellwände enthalten, was zu Strukturen führt, die sehr widerstandsfähig gegen Umweltbelastungen sind. Diese Sporen können lange Zeiträume überleben, bis die Bedingungen günstiger werden, z. B. durch verbesserte Nährstoffe, woraufhin sie keimen und sich ungeschlechtlich vermehren können. Diese verschiedenen Stadien, Knospung, Konjugation und Sporulation, bilden zusammen den Lebenszyklus der Hefe.

Wachstum und Stoffwechsel der Hefe
Wenn Hefezellen in kohlenstoffreichen Quellen wie Glukose gezüchtet werden, ziehen sie es vor, durch Gärung zu wachsen. Während der Gärung wird Glukose in Kohlendioxid und Ethanol umgewandelt. Im Allgemeinen findet die Gärung in Abwesenheit von Sauerstoff statt und ist daher von Natur aus anaerob. Selbst in Gegenwart von Sauerstoff ziehen Hefezellen ein fermentatives Wachstum vor, das nach dem Biologen, der diese Vorliebe entdeckte, als Crabtree-Effekt bezeichnet wird. Diese Form des Wachstums wird bei der Herstellung von Brot, Bier, Wein und anderen alkoholischen Getränken ausgenutzt. Obwohl die Hefezellen vorzugsweise durch Gärung wachsen, können sie bei Nährstoffmangel auch durch Zellatmung wachsen. Während der Atmung wandeln die Zellen Glukose in Kohlendioxid und Wasser um, wobei sie Sauerstoff verbrauchen und viel größere Mengen an Energie in Form von ATP produzieren.
Historische Entdeckungen

Hefe wird seit Tausenden von Jahren als industrieller Mikroorganismus verwendet. Die alten Ägypter nutzten die Hefegärung, um Brot zu säuern. Es gibt Hinweise auf Mahlsteine, Backkammern und Zeichnungen von 4000 Jahre alten Bäckereien. Archäologische Ausgrabungen haben Beweise in Form von Gläsern mit 7.000 Jahre alten Weinresten zutage gefördert.
Hefe wurde erstmals 1680 von Antoni van Leeuwenhoek mit hochwertigen Linsen sichtbar gemacht. Er glaubte jedoch, dass es sich bei diesen Kügelchen um stärkehaltige Partikel des Getreides handelte, die zur Herstellung der Würze, dem flüssigen Extrakt beim Brauen, verwendet wurden, und nicht um gärende Hefezellen. 1789 trug Antoine Lavoisier, ein französischer Chemiker, zu unserem Verständnis der grundlegenden chemischen Reaktionen bei, die zur Herstellung von Alkohol aus Zuckerrohr erforderlich sind. Durch Schätzung des Verhältnisses von Ausgangsstoffen und Produkten (Ethanol und Kohlendioxid) nach Zugabe von Hefeteig kam er zu dem Schluss, dass zwei chemische Wege genutzt wurden, wobei zwei Drittel des Zuckers zu Alkohol und ein Drittel zu Kohlendioxid reduziert wurden. Damals ging man jedoch davon aus, dass die Hefe lediglich dazu diente, die Reaktion einzuleiten, und nicht während des gesamten Prozesses erforderlich war.
Im Jahr 1815 entwickelte der französische Chemiker Joseph-Louis Gay-Lussac Methoden, um Traubensaft in einem unvergorenen Zustand zu halten, und entdeckte, dass die Einführung von „Ferment“ (das Hefe enthält) erforderlich war, um unvergorene Würze umzuwandeln, was die Bedeutung der Hefe für die alkoholische Gärung zeigte. 1835 konnte Charles Cagniard de la Tour mit Hilfe eines leistungsfähigeren Mikroskops zeigen, dass Hefen einzellig sind und sich durch Knospung vermehren. In den 1850er Jahren entdeckte Louis Pasteur, dass vergorene Getränke durch die Umwandlung von Glukose in Ethanol durch Hefe entstehen, und definierte Gärung als „Atmung ohne Luft“. Jahrhunderts verwendete Eduard Buchner zellfreie Extrakte, die durch Zerkleinern von Hefezellen gewonnen wurden, um Zymase nachzuweisen, eine Sammlung von Enzymen, die die Gärung fördern oder katalysieren, wofür er 1907 den Nobelpreis erhielt.
Ein Großteil der Pionierarbeit zur Hefegenetik wurde von Øjvind Winge geleistet. Er entdeckte, dass Hefe zwischen haploiden und diploiden Zuständen wechselt und dass Hefe heterothallisch ist, da zwei Stämme erforderlich sind, um Haploide in Diploide umzuwandeln (Konjugation). Er und sein Kollege Otto Laustsen entwickelten Techniken zur Mikromanipulation von Hefe, um sie genetisch untersuchen zu können. Bei dieser als „Tetrad-Analyse“ bezeichneten Technik wird mit Hilfe einer feinen Nadel und eines Mikroskops eine als Ascus bezeichnete Struktur isoliert, die die vier Sporenprodukte oder Tetraden enthält, die bei der Sporulation eines Diploiden entstehen. Sobald der Ascus isoliert ist, werden die Sporen in der Tetrade auseinandergezogen und zu Kolonien für die genetische Analyse heranwachsen gelassen. Diese Pionierarbeit brachte ihm den Titel „Vater der Hefegenetik“ ein. Einige dieser Arbeiten wurden von Carl Lindegren weiter aufgeklärt, der das Paarungstypsystem in der Knospenhefe aufklärte, indem er die Existenz von Mat a- und Mat alpha-Zellen nachwies, Methoden zur Durchführung von Massenverpaarungen zwischen Zellen dieser Paarungstypen entwickelte und dieses Wissen zur Untersuchung der Genetik der Zuckernutzung nutzte.
Seit dieser Zeit haben viele andere Forscher bahnbrechende Forschungen mit der Knospenhefe durchgeführt. Einige dieser Forscher wurden für ihre bedeutenden Entdeckungen mit dem Nobelpreis ausgezeichnet, darunter: Dr. Leland Hartwell (2001) für die Entdeckung von Genen, die den Zellzyklus regulieren (gemeinsam mit Paul Nurse und Tim Hunt); Roger Kornberg (2006) für seine Studien über den ersten Schritt der Genexpression, d. h. die Art und Weise, wie die DNA-Sequenz eines Gens in die Boten-RNA (mRNA) kopiert wird; Dr. Elizabeth Blackburn, Carol Greider und Dr. Michael Hunt. Elizabeth Blackburn, Carol Greider und Jack Szostak (2009) für die Entdeckung und Aufklärung der Gene und Mittel, mit denen die Zellen die Chromosomenenden oder Telomere vor dem Abbau schützen, und Dr. Randy Schekman, James Rothman und Thomas Südhof (2013) für ihre Forschungen über die Maschinerie, die den Blasenverkehr reguliert. Zuletzt erhielt Dr. Yoshinori Ohsumi den Preis für seine Arbeit zur Autophagie, die mit Studien an Hefe begann.
Kommerzielle Anwendungen

Hefe gilt seit langem als der Organismus der Wahl für die Herstellung von alkoholischen Getränken, Brot und einer Vielzahl von Industrieprodukten. Dies beruht auf der Leichtigkeit, mit der der Stoffwechsel der Hefe mit Hilfe genetischer Techniken manipuliert werden kann, der Schnelligkeit, mit der sie zu hohen Zellausbeuten (Biomasse) gezüchtet werden kann, der Leichtigkeit, mit der diese Biomasse von Produkten getrennt werden kann, und dem Wissen, dass sie allgemein als sicher anerkannt ist (GRAS).
Die Sprossenhefe S. cerevisiae und andere Hefearten werden seit langem zur Fermentierung des Zuckers von Reis, Weizen, Gerste und Mais verwendet, um alkoholische Getränke wie Bier und Wein herzustellen. Es gibt zwei Hauptarten von Bierhefe: obergärige Bierhefe und untergärige Lagerhefe. Obergärige Hefe wie z. B. S. cerevisiae steigen während der Gärung an die Oberfläche und werden zum Brauen von Ales, Porters, Stouts und Weizenbieren verwendet. Im Gegensatz dazu ist S. pastorianus (früher bekannt als S. carlsbergensis) eine untergärige Hefe, die zur Herstellung von Lagerbier verwendet wird. Lagerhefen wachsen am besten bei niedrigeren Temperaturen. Daher wachsen sie langsamer, produzieren weniger Oberflächenschaum und setzen sich daher in der Regel am Boden des Gärbehälters ab. Pilsner, Märzen, Bocks und amerikanische Malzliköre sind allesamt Lagerbierstile. In der modernen Brauerei wurden viele der ursprünglichen obergärigen Stämme so verändert, dass sie zu Untergärern wurden.
Hefen erzeugen Wein, indem sie Zucker aus Traubensaft (Most) zu Ethanol vergären. Obwohl die Gärung von Wein durch natürlich vorkommende Hefen in den Weinbergen eingeleitet werden kann, entscheiden sich viele Weinkellereien für die Zugabe einer reinen Hefekultur, um die Gärung zu beherrschen und zu kontrollieren. Die Bläschen in Champagner und Schaumweinen entstehen durch eine zweite Gärung, in der Regel in der Flasche, bei der das Kohlendioxid eingeschlossen wird. Kohlendioxid, das bei der Weinherstellung entsteht, wird als Nebenprodukt freigesetzt. Eine Hefezelle kann etwa ihr Eigengewicht an Glukose pro Stunde vergären. Unter optimalen Bedingungen kann S. cerevisiae bis zu 18 Volumenprozent Ethanol produzieren, wobei 15 – 16 % die Norm sind. Das Schwefeldioxid, das in kommerziell hergestelltem Wein enthalten ist, wird unmittelbar nach dem Zerquetschen der Trauben zugesetzt, um die natürlich vorhandenen Bakterien, Schimmelpilze und Hefen abzutöten.

Saccharomyces cerevisiae oder Bäckerhefe wird seit langem als Triebmittel beim Backen verwendet. Die Bäckerhefe vergärt den im Teig enthaltenen Zucker und erzeugt dabei Kohlendioxid und Ethanol. Das Kohlendioxid wird in kleinen Bläschen im Teig eingeschlossen, was dazu führt, dass der Teig aufgeht. Eine Ausnahme bildet das Sauerteigbrot, das nicht mit Bäckerhefe, sondern mit einer Kombination aus wilder Hefe und Bakterien hergestellt wird. Die Hefe Candida milleri wird zur Stärkung des Klebers und ein säurebildendes Bakterium „Lactobacillus sanfranciscensis“ zur Fermentierung des Malzzuckers verwendet.
Neben diesen traditionellen Verwendungen wird Hefe auch für viele andere kommerzielle Zwecke eingesetzt. Veganer verwenden Hefe oft als Käseersatz, und sie wird häufig als Topping für Produkte wie Popcorn verwendet. In der petrochemischen Industrie wird sie zur Herstellung von Biokraftstoffen wie Ethanol und Farnesen, einem Vorprodukt für Diesel und Düsentreibstoff, eingesetzt. Sie wird auch bei der Herstellung von Schmiermitteln und Reinigungsmitteln verwendet. Hefe wird in der Lebensmittelindustrie für die Herstellung von Lebensmittelzusatzstoffen wie Farbstoffen, Antioxidantien und Geschmacksverstärkern verwendet. Sie wird häufig bei der Herstellung von Arzneimitteln wie Antiparasitika, Krebsmitteln, Biopharmazeutika wie Insulin, Impfstoffen und Nutrazeutika verwendet. Hefe wird häufig für die Herstellung von industriellen Enzymen und Chemikalien verwendet. Auf dem Gebiet der Umweltsanierung werden Hefestämme sogar zur Entfernung von Metallen aus Bergbauabfällen eingesetzt.
Anwendung bei menschlichen Krankheiten und in der Forschung

Aufgrund der großen Ähnlichkeit zwischen Hefegenen und ihren menschlichen Gegenstücken und der konservierten grundlegenden Zellbiologie ist Hefe zu einem beliebten Modellsystem für die Untersuchung menschlicher Krankheitsgene geworden. Es gibt verschiedene Ansätze, um mehr über menschliche Gene zu erfahren, sobald eine Verbindung zwischen einem menschlichen und einem Hefegen hergestellt ist. Bei einem Ansatz wird nach der Entdeckung eines mit einer menschlichen Krankheit assoziierten Gens die Sequenz mit den Sequenzen aller Gene im Hefegenom verglichen, um das oder die ähnlichsten Hefegene zu ermitteln. Um zu untersuchen, ob die Gene funktionell verwandt sind, wird das menschliche Gen dann in einem Hefekeim exprimiert, in dem das Hefegen zuvor durch Mutation inaktiviert wurde. Auf diese Weise können die Forscher feststellen, ob das menschliche Gen in der Lage ist, die Lebensfähigkeit, das Wachstum oder spezifischere Defekte, die mit dem Verlust des Hefegens einhergehen, zu beheben, eine Methode, die als funktionelle Komplementierung bezeichnet wird. Wenn die Wege und/oder Prozesse, an denen ein Hefegen beteiligt ist, konserviert sind, kann man viel über die Funktion des menschlichen Gens auf der Grundlage dessen lernen, was bereits über das entsprechende Hefegen bekannt ist. Sobald eine funktionelle Komplementierung festgestellt wurde, können Forscher dieses System nutzen, um die Funktion des verwandten menschlichen Genprodukts weiter zu charakterisieren. Weniger gezielte Ansätze, bei denen häufig Hochdurchsatztechniken (HTP) eingesetzt werden, um Tausende von menschlichen Genen gleichzeitig nach dem Zufallsprinzip zu durchsuchen, um Gene mit komplementärer Aktivität zu identifizieren. Solche Ansätze wurden erfolgreich eingesetzt, um konservierte Zellzyklusregulatoren (CDC2), Gene, die an Krebs beteiligt sind, und Gene, die an neurodegenerativen Erkrankungen beteiligt sind, zu identifizieren.
Es gibt viele Szenarien, in denen Studien den Forschern wertvolle Informationen über die zellulären Wege und/oder Prozesse liefern können, an denen ein menschliches Gen beteiligt ist, wenn ein verwandtes Hefegen nicht vorhanden ist. Beispielsweise treten einige neurodegenerative Krankheiten wie Alzheimer und Parkinson dadurch auf, dass sich aufgrund von Proteinfehlfaltung Proteinaggregate, so genanntes Amyloid, ansammeln, die für die Nervenzellen toxisch sind. Die Untersuchung von fehlgefalteten Hefeproteinen mit ähnlichem Amyloidbildungspotenzial, den so genannten Prionen, hat den Forschern Einblicke in diese neurodegenerativen Krankheiten verschafft. Alternativ kann auch eine erhöhte Expression eines krankheitsassoziierten Gens in Hefe zu einem Phänotyp führen. So ist beispielsweise Alpha-Synuclein, ein mit der Parkinson-Krankheit assoziiertes Gen, toxisch, wenn es in ausreichender Menge exprimiert wird. Ein solcher Stamm kann dann verwendet werden, um nach Hefegenen oder kleinen Molekülen zu suchen, die die Synuclein-induzierte Toxizität unterdrücken oder verstärken, was oft Hinweise auf die relevanten zellulären Signalwege liefert. Patienten mit Amyotropher Lateralsklerose (ALS) oder der Lou-Gehrig-Krankheit haben oft Mutationen in einer Reihe von RNA-bindenden Proteinen, die zur Bildung von Aggregaten neigen, die den RNA-Stoffwechsel stören. Mit Hilfe eines Hefe-Screens konnte eine Reihe von Hefegenen mit ähnlichen Eigenschaften (Bildung toxischer Aggregate) identifiziert werden, was den Forschern neue Kandidatengene zur Untersuchung lieferte. Umgekehrt bilden die menschlichen RNA-bindenden Proteine toxische Aggregate, wenn sie in Hefe exprimiert werden, und dieser Stamm wurde verwendet, um ein Hefegen zu identifizieren, das, wenn es mutiert ist, die Produktion dieser Aggregate blockiert.
Hefe wird zum Organismus der Wahl bei Studien, die auf die Identifizierung von Arzneimittelzielen und die Wirkungsweise verschiedener Arzneimittel abzielen. Chemogenomik oder chemische Genomik bezieht sich auf die Untersuchungen, bei denen eine Kombination aus Chemikalien und Genomik verwendet wird, um Arzneimittelziele zu untersuchen und potenziell neue Arzneimittel zu identifizieren. Bei diesen chemisch-genomischen Studien wurden hauptsächlich zwei Ansätze verwendet. Beim ersten Ansatz wird eine genomweite Sammlung diploider Stämme angelegt, bei denen eine der beiden identischen Kopien eines Gens entfernt wird, wodurch die Konzentration eines bestimmten Genprodukts gesenkt wird. Zielgene und Gene, die an dem Zielweg beteiligt sind, reagieren empfindlicher auf die Substanz und werden bei dieser Art von Screening bevorzugt identifiziert. Bei einem zweiten Ansatz werden nicht essenzielle Gene systematisch entfernt und die Sammlung mit einem Medikament gescreent, um nach Genen zu suchen, die den Zielpfad des Medikaments puffern. Mit diesem Ansatz sollen Gene identifiziert werden, die für das Wachstum in Gegenwart des Wirkstoffs erforderlich sind. Weitere Ansätze mit Hilfe von Überexpressionsscreens wurden angewandt, um Gene zu identifizieren, die an der Arzneimittelresistenz beteiligt sind, einschließlich des potenziellen Zielgenoms. Der Vergleich des Expressionsprofils von Hefezellen, bei denen ein Gen deletiert wurde, mit dem von Wildtyp-Hefezellen, die mit einem bestimmten Medikament behandelt wurden, kann ebenfalls ein wirksames Mittel sein, um Gene zu identifizieren, die den Forschern etwas darüber verraten, wie das Medikament in den Zellen wirkt.
Dies sind nur einige Beispiele dafür, wie Hefe zur Erforschung menschlicher Krankheiten eingesetzt werden kann. Studien in Hefe können Forschern dabei helfen, mehr über die zugrunde liegende Biologie zu erfahren, indem sie dieses Modellsystem nutzen, oder sie können ihnen dabei helfen, Arzneimittelziele oder die Wirkungsweise von Arzneimitteln zu identifizieren.
Ressourcen
- Die Saccharomyces Genome Database (SGD) bietet umfassende integrierte biologische Informationen für die Knospenhefe Saccharomyces cerevisiae zusammen mit Such- und Analysewerkzeugen zur Erforschung dieser Daten.
- MIPS Comprehensive Yeast Genome Database (CYPD) bietet Informationen über die molekulare Struktur und das funktionelle Netzwerk des vollständig sequenzierten, gut untersuchten Modell-Eukaryoten, der Knospenhefe Saccharomyces cerevisiae.
- Candida Genome Database (CGD), eine Ressource für genomische Sequenzdaten sowie Gen- und Proteininformationen für Candida albicans.
- PomBase, eine umfassende Datenbank für die Spalthefe Schizosaccharomyces pombe, die strukturelle und funktionelle Annotation, Literaturkuratierung und Zugang zu großen Datensätzen bietet.
- Fungal genome resources at NCBI, ein Leitfaden für Pilzgenom-Ressourcen beim National Center for Biotechnology Information (NCBI).
- Eine umfangreiche Liste hefebezogener Ressourcen zu Themen, die von allgemeinen Informationen über Hefe bis hin zu Nukleinsäuren, Genomen und Proteinen, Expressionsdaten, Lokalisierung, Phänotypen und mehr reichen.
Suggested Reading
Books
- YeastBook. (2011) Ein umfassendes Kompendium von Übersichtsarbeiten, das den aktuellen Stand des Wissens über die Molekularbiologie, Zellbiologie und Genetik der Hefe Saccharomyces cerevisiae darstellt, Genetik
- Von a bis alpha: Yeast as a Model for Cellular Differentiation. (2007) Hiten D. Madhani, Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York.
- Landmark Papers in Yeast Biology. (2006), herausgegeben von Patrick Linder, David Shore, und Michael N. Hall, Cold Spring Harbor Press, Cold Spring Harbor, New York.
- Methods in Yeast Genetics: A Cold Spring Harbor Laboratory Course Manual. (2005) David C. Amberg, Daniel J. Burke, and Jeffrey N. Strathern. Cold Spring Laboratory Press, Cold Spring Harbor, New York.
- The Early Days of Yeast Genetics. (1993) herausgegeben von Michael N. Hall und Patrick Linder. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York.
- Band I: The Molecular and Cellular Biology of the Yeast Saccharomyces cerevisiae: Genome Dynamics, Protein Synthesis, and Energetics. (1991) herausgegeben von James R. Broach, John R. Pringle, und Elizabeth W. Jones. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York.
- Band II: Die molekulare und zelluläre Biologie der Hefe Saccharomyces cerevisiae: Gene Expression. (1992) herausgegeben von Elizabeth W. Jones, John R. Pringle und James R. Broach. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York.
- Band III: The Molecular and Cellular Biology of the Yeast Saccharomyces cerevisiae: Cell Cycle and Cell Biology. (1997) herausgegeben von John R. Pringle, James R. Broach und Elizabeth W. Jones. Cold Spring Harbor Laboratory Press, Cold Spring Harbor, New York.
- Yeast: A Practical Approach. (1988) edited by I. Campbell and , and John H. Duffus, IRL Press, Ithaca, New York.
Journal Articles
- Duina A.A., Miller M.E., and J.B. Keeney (2014) Budding Yeast for Budding Geneticists: A Primer on the Saccharomyces cerevisiae Model System. Genetics. 197:33-48.
- Botstein D. and G.R. Fink (2011) Yeast: Ein experimenteller Organismus für die Biologie des 21. Jahrhunderts. Genetics. 189:695-704.
- Cherry J.M., Hong E.L., Amundsen C., Balakrishnan R., Binkley G., Chan E.T., Christie K.R., Costanzo M.C., Dwight S.S., Engel S.R., Fisk D.G., Hirschman J.E., Hitz B.C., Karra K., Krieger C.J., Miyasato S.R., Nash R.S., Park J., Skrzypek M.S., Simison M., Weng S., and E.D. Wong (2011) Saccharomyces Genome Database: the genomics resource of budding yeast. Nucleic Acids Res. 40:D700-D705.
- Altman T.J., Boone C., Churchill G.A., Hengartner M.O., Mackay T.F., and D.L. Stemple (2011) The future of model organisms in human disease research. Nat. Rev. Genet. 18:575-582.
- Ho C.H., Piotrowski J., Dixon S.J., Baryshnikova A., Costanzo M., and C. Boone (2011) Combining functional genomics and chemical biology to identify targets of bioactive compounds. Curr Opin Chem Biol. 15:66-78.
- Smith A.M., Ammar R., Nislow C., and G. Giaever (2010) A survey of yeast genomic assays for drug and target discovery. Pharmacol Ther. 127:156-164.
- Dolinski K., and D. Botstein (2007) Orthology and functional conservation in eukaryotes. Annu. Rev. Genet. 41:465-507.
- Barnett J.A. (2007) A history of research on yeasts 10: foundations of yeast genetics. Yeast 24:799-845.
- Spradling A., Ganetsky B., Hieter P., Johnston M., Olson M., Orr-Weaver T., Rossant J., Sanchez A., and R. Waterston (2006) New roles for model genetic organisms in understanding and treating human disease: report from the 2006 Genetics Society of America meeting. Genetics 172:2025-2032.
- Sherman F. (2002) Getting started with yeast Methods Enzymol. 350:3-41.