Souligner le processus de transcription et de traduction procaryotes
Les procaryotes, qui comprennent les bactéries et les archées, sont pour la plupart des organismes unicellulaires qui, par définition, n’ont pas de noyau lié à une membrane ni d’autres organites. Un chromosome bactérien est un cercle fermé de manière covalente qui, contrairement aux chromosomes eucaryotes, n’est pas organisé autour de protéines histones. La région centrale de la cellule dans laquelle réside l’ADN procaryote est appelée le nucléoïde. En outre, les procaryotes possèdent souvent d’abondants plasmides, qui sont des molécules d’ADN circulaires plus courtes pouvant ne contenir qu’un ou quelques gènes. Les plasmides peuvent être transférés indépendamment du chromosome bactérien pendant la division cellulaire et sont souvent porteurs de caractéristiques telles que la résistance aux antibiotiques. En raison de ces caractéristiques uniques, la transcription et la régulation des gènes sont quelque peu différentes entre les cellules procaryotes et les cellules eucaryotes.
Objectifs d’apprentissage
- Comprendre les étapes de base de la transcription de l’ADN en ARN dans les cellules procaryotes
- Comprendre les bases de la traduction procaryote. et comment elle diffère de la traduction eucaryote
Transcription procaryote
Initiation de la transcription chez les procaryotes
Les procaryotes n’ont pas de noyau enfermé dans une membrane.de membrane. Par conséquent, les processus de transcription, de traduction et de dégradation de l’ARNm peuvent tous se produire simultanément. Le niveau intracellulaire d’une protéine bactérienne peut rapidement être amplifié par de multiples événements de transcription et de traduction se produisant simultanément sur la même matrice d’ADN. La transcription procaryote couvre souvent plus d’un gène et produit des ARNm polycistroniques qui spécifient plus d’une protéine.
Notre discussion ici exemplifiera la transcription en décrivant ce processus chez Escherichia coli, une espèce bactérienne bien étudiée. Bien que certaines différences existent entre la transcription chez E. coli et la transcription chez les archées, une compréhension de la transcription chez E. coli peut être appliquée à pratiquement toutes les espèces bactériennes.
Arn polymérase procaryote
Les procaryotes utilisent la même ARN polymérase pour transcrire tous leurs gènes. Chez E. coli, la polymérase est composée de cinq sous-unités polypeptidiques, dont deux sont identiques. Quatre de ces sous-unités, dénommées α, α, β et β′ constituent l’enzyme centrale de la polymérase. Ces sous-unités s’assemblent chaque fois qu’un gène est transcrit, et se désassemblent une fois la transcription terminée. Chaque sous-unité a un rôle unique ; les deux sous-unités α sont nécessaires pour assembler la polymérase sur l’ADN ; la sous-unité β se lie au ribonucléoside triphosphate qui fera partie de la molécule d’ARNm naissante « récemment née » ; et la β′ se lie au brin d’ADN matrice. La cinquième sous-unité, σ, n’intervient que dans l’initiation de la transcription. Elle confère une spécificité transcriptionnelle telle que la polymérase commence à synthétiser l’ARNm à partir d’un site d’initiation approprié. Sans σ, l’enzyme centrale transcrirait à partir de sites aléatoires et produirait des molécules d’ARNm qui spécifieraient un charabia de protéines. La polymérase composée des cinq sous-unités est appelée l’holoenzyme (un holoenzyme est un composé biochimiquement actif composé d’une enzyme et de sa coenzyme).
Prokaryotic Promoters

Figure 1. La sous-unité σ de l’ARN polymérase procaryote reconnaît les séquences consensus présentes dans la région du promoteur en amont du site de début de transcription. La sous-unité σ se dissocie de la polymérase après que la transcription ait été initiée.
Un promoteur est une séquence d’ADN sur laquelle la machinerie de transcription se lie et initie la transcription. Dans la plupart des cas, les promoteurs existent en amont des gènes qu’ils régulent. La séquence spécifique d’un promoteur est très importante car elle détermine si le gène correspondant est transcrit tout le temps, parfois ou rarement. Bien que les promoteurs varient selon les génomes procaryotes, quelques éléments sont conservés. Dans les régions -10 et -35 en amont du site d’initiation, il existe deux séquences consensus de promoteur, ou des régions qui sont similaires dans tous les promoteurs et dans diverses espèces bactériennes (Figure 1).
La séquence consensus -10, appelée région -10, est TATAAT. La séquence -35, TTGACA, est reconnue et liée par le σ. Une fois cette interaction réalisée, les sous-unités de l’enzyme centrale se fixent sur le site. La région -10 riche en A-T facilite le déroulement de la matrice d’ADN, et plusieurs liaisons phosphodiesters sont réalisées. La phase d’initiation de la transcription se termine par la production de transcrits abortifs, qui sont des polymères d’environ 10 nucléotides fabriqués et libérés.
Elongation et terminaison chez les procaryotes
La phase d’élongation de la transcription commence avec la libération de la sous-unité σ de la polymérase. La dissociation de σ permet à l’enzyme centrale de progresser le long de la matrice d’ADN, synthétisant l’ARNm dans la direction 5′ à 3′ à un rythme d’environ 40 nucléotides par seconde. Au fur et à mesure de l’élongation, l’ADN est continuellement déroulé devant l’enzyme centrale et rembobiné derrière elle (figure 2). L’appariement des bases entre l’ADN et l’ARN n’est pas suffisamment stable pour maintenir la stabilité des composants de la synthèse de l’ARNm. Au lieu de cela, l’ARN polymérase agit comme un lieur stable entre la matrice d’ADN et les brins d’ARN naissants pour s’assurer que l’élongation n’est pas interrompue prématurément.

Figure 2. Cliquez pour agrandir l’image. Pendant l’élongation, l’ARN polymérase procaryote suit la matrice d’ADN, synthétise l’ARNm dans le sens 5′ à 3′, et déroule et rembobine l’ADN au fur et à mesure de sa lecture.
Signaux de terminaison procaryotes
Une fois qu’un gène est transcrit, la polymérase procaryote doit recevoir l’ordre de se dissocier de la matrice d’ADN et de libérer l’ARNm nouvellement fabriqué. Selon le gène transcrit, il existe deux types de signaux de terminaison. L’un est basé sur les protéines et l’autre sur l’ARN. La terminaison dépendante de Rho est contrôlée par la protéine rho, qui suit la polymérase sur la chaîne d’ARNm en croissance. Vers la fin du gène, la polymérase rencontre une série de nucléotides G sur la matrice d’ADN et elle s’arrête. En conséquence, la protéine rho entre en collision avec la polymérase. L’interaction avec rho libère l’ARNm de la bulle de transcription.
La terminaison indépendante de rho est contrôlée par des séquences spécifiques du brin d’ADN matrice. Lorsque la polymérase s’approche de la fin du gène en cours de transcription, elle rencontre une région riche en nucléotides C-G. L’ARNm se replie sur lui-même, et les nucléotides C-G complémentaires se lient entre eux. Il en résulte une épingle à cheveux stable qui provoque l’arrêt de la polymérase dès qu’elle commence à transcrire une région riche en nucléotides A-T. La région complémentaire U-A de l’ARNm transcrit ne forme qu’une faible interaction avec l’ADN matrice. Ceci, couplé à la polymérase bloquée, induit une instabilité suffisante pour que l’enzyme centrale se détache et libère le nouveau transcrit d’ARNm.
Lors de la terminaison, le processus de transcription est terminé. Au moment où la terminaison se produit, le transcrit procaryote aurait déjà été utilisé pour commencer la synthèse de nombreuses copies de la protéine codée, car ces processus peuvent se produire simultanément. L’unification de la transcription, de la traduction et même de la dégradation de l’ARNm est possible parce que tous ces processus se produisent dans la même direction 5′ à 3′, et parce qu’il n’y a pas de compartimentation membranaire dans la cellule procaryote (Figure 3). En revanche, la présence d’un noyau dans les cellules eucaryotes empêche la transcription et la traduction simultanées.

Figure 3. Plusieurs polymérases peuvent transcrire un seul gène bactérien tandis que de nombreux ribosomes traduisent simultanément les transcriptions d’ARNm en polypeptides. De cette façon, une protéine spécifique peut rapidement atteindre une concentration élevée dans la cellule bactérienne.
Visitez cette animation BioStudio pour voir le processus de transcription procaryote.
Questions pratiques
Quelle sous-unité de la polymérase de E. coli confère une spécificité à la transcription ?
- α
- β
- β′
- σ
Les régions -10 et -35 des promoteurs procaryotes sont appelées séquences consensus car ________.
- elles sont identiques dans toutes les espèces bactériennes
- elles sont similaires dans toutes les espèces bactériennes
- elles existent dans tous les organismes
- elles ont la même fonction dans tous les organismes
La traduction procaryote
La traduction est similaire chez les procaryotes et les eucaryotes. Nous allons ici explorer comment la traduction se déroule chez E. coli, un procaryote représentatif, et préciser les éventuelles différences entre la traduction bactérienne et eucaryote.
Initiation
L’initiation de la synthèse protéique commence par la formation d’un complexe d’initiation. Chez E. coli, ce complexe implique le petit ribosome 30S, la matrice d’ARNm, trois facteurs d’initiation qui aident le ribosome à s’assembler correctement, la guanosine triphosphate (GTP) qui agit comme source d’énergie, et un ARNt initiateur spécial portant la N-formyl-méthionine (fMet-tRNAfMet) (Figure 4). L’ARNt initiateur interagit avec le codon d’initiation AUG de l’ARNm et porte une méthionine formylée (fMet). En raison de son implication dans l’initiation, la fMet est insérée au début (terminus N) de chaque chaîne polypeptidique synthétisée par E. coli. Dans l’ARNm d’E. coli, une séquence leader en amont du premier codon AUG, appelée séquence Shine-Dalgarno (également connue sous le nom de site de liaison ribosomal AGGAGG), interagit par appariement de bases complémentaires avec les molécules d’ARNr qui composent le ribosome. Cette interaction ancre la sous-unité ribosomique 30S au bon endroit sur la matrice d’ARNm. À ce stade, la sous-unité ribosomique 50S se lie ensuite au complexe d’initiation, formant ainsi un ribosome intact.
Chez les eucaryotes, la formation du complexe d’initiation est similaire, avec les différences suivantes :
- L’ARNt initiateur est un ARNt spécialisé différent portant la méthionine, appelé Met-tRNAi
- Au lieu de se lier à l’ARNm au niveau de la séquence Shine-Dalgarno, le complexe d’initiation eucaryote reconnaît la coiffe 5′ de l’ARNm eucaryote, puis se déplace le long de l’ARNm dans la direction 5′ à 3′ jusqu’à ce que le codon d’initiation AUG soit reconnu. À ce stade, la sous-unité 60S se lie au complexe de Met-tRNAi, de l’ARNm et de la sous-unité 40S.

Figure 4. La traduction chez les bactéries commence par la formation du complexe d’initiation, qui comprend la petite sous-unité ribosomale, l’ARNm, l’ARNt initiateur portant la N-formyl-méthionine, et les facteurs d’initiation. Puis la sous-unité 50S se lie, formant ainsi un ribosome intact.
Elongation
Chez les procaryotes et les eucaryotes, les bases de l’élongation de la traduction sont les mêmes. Chez E. coli, la liaison de la sous-unité ribosomique 50S pour produire le ribosome intact forme trois sites ribosomiques fonctionnellement importants : Le site A (aminoacyle) fixe les ARNt aminoacyle chargés entrants. Le site P (peptidyle) lie les ARNt chargés portant des acides aminés qui ont formé des liaisons peptidiques avec la chaîne polypeptidique en croissance mais qui ne se sont pas encore dissociés de leur ARNt correspondant. Le site E (exit) libère les ARNt dissociés afin qu’ils puissent être rechargés en acides aminés libres. Il existe une exception notable à cette chaîne d’assemblage des ARNt : Au cours de la formation du complexe d’initiation, l’ARNfMet bactérien ou l’ARNmt eucaryote entre directement dans le site P sans entrer d’abord dans le site A, fournissant un site A libre prêt à accepter l’ARNt correspondant au premier codon après l’AUG.
L’élongation se poursuit par des mouvements monocodon du ribosome appelés chacun événement de translocation. Au cours de chaque événement de translocation, les ARNt chargés entrent au niveau du site A, puis se déplacent vers le site P, et enfin vers le site E pour être éliminés. Les mouvements ribosomiques, ou étapes, sont induits par des changements de conformation qui font avancer le ribosome de trois bases dans la direction 3′. Des liaisons peptidiques se forment entre le groupe amino de l’acide aminé fixé à l’ARNt du site A et le groupe carboxyle de l’acide aminé fixé à l’ARNt du site P. La formation de chaque liaison peptidique est catalysée par la peptidyl transférase, une ribozyme à base d’ARN qui est intégrée dans la sous-unité ribosomique 50S. L’acide aminé lié à l’ARNt du site P est également lié à la chaîne polypeptidique en croissance. Lorsque le ribosome traverse l’ARNm, l’ancien ARNt du site P entre dans le site E, se détache de l’acide aminé et est expulsé. Plusieurs des étapes de l’élongation, notamment la liaison d’un ARNt aminoacyle chargé au site A et la translocation, nécessitent de l’énergie dérivée de l’hydrolyse du GTP, catalysée par des facteurs d’élongation spécifiques. Étonnamment, l’appareil de traduction d’E. coli ne prend que 0,05 seconde pour ajouter chaque acide aminé, ce qui signifie qu’une protéine de 200 acides aminés peut être traduite en seulement 10 secondes.
Termination
La terminaison de la traduction se produit lorsqu’un codon non-sens (UAA, UAG ou UGA) est rencontré pour lequel il n’y a pas d’ARNt complémentaire. En s’alignant sur le site A, ces codons non sens sont reconnus par des facteurs de libération chez les procaryotes et les eucaryotes, ce qui entraîne le détachement de l’acide aminé du site P de son ARNt, libérant ainsi le polypeptide nouvellement fabriqué. Les petites et grandes sous-unités ribosomiques se dissocient de l’ARNm et les unes des autres ; elles sont recrutées presque immédiatement dans un autre complexe d’init iation de la traduction.
En résumé, il existe plusieurs caractéristiques clés qui distinguent l’expression génique procaryote de celle observée chez les eucaryotes. Elles sont illustrées dans la figure 5 et énumérées dans le tableau 1.
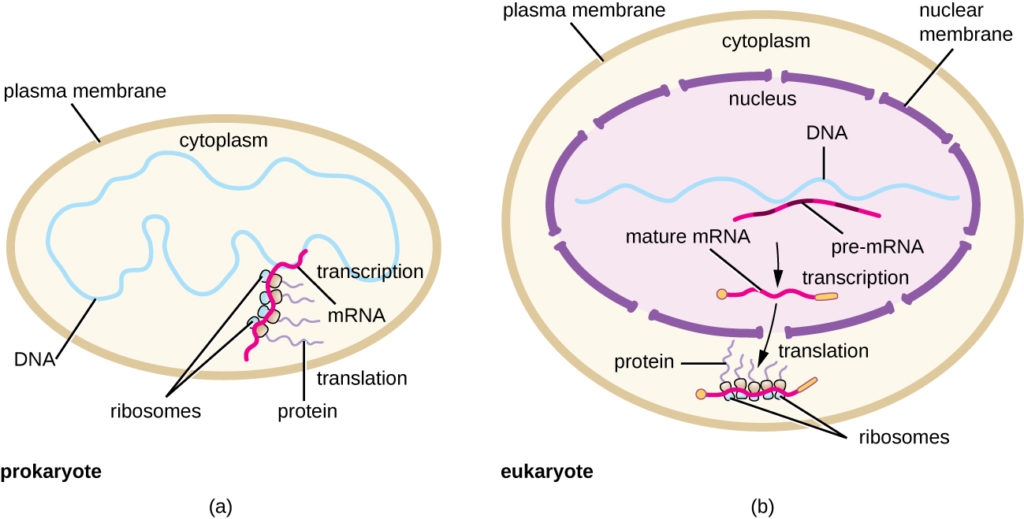
Figure 5. (a) Chez les procaryotes, les processus de transcription et de traduction se produisent simultanément dans le cytoplasme, ce qui permet une réponse cellulaire rapide à un indice environnemental. (b) Chez les eucaryotes, la transcription est localisée dans le noyau et la traduction dans le cytoplasme, séparant ces processus et nécessitant le traitement de l’ARN pour la stabilité.
| Tableau 1. Comparaison de la traduction chez les bactéries par rapport aux eucaryotes | ||
|---|---|---|
| Propriété | Bactéries | Eucaryotes |
| Ribosomes | 70S
|
80S
|
| Acide aminé porté par l’initiateur tRNA | fMet | Met |
| Shine-.Séquence Dalgarno dans l’ARNm | Présent | Absent |
| Transcription et traduction simultanées | Oui | Non |
Vérifiez votre compréhension
Répondez à la ou aux questions ci-dessous pour voir dans quelle mesure vous comprenez les sujets traités dans la section précédente. Ce court questionnaire ne compte pas pour votre note dans le cours, et vous pouvez le reprendre un nombre illimité de fois.
Utilisez ce questionnaire pour vérifier votre compréhension et décider si (1) vous devez approfondir la section précédente ou (2) passer à la section suivante.
.