Depuis les découvertes séminales des principes de base qui sous-tendent le clonage moléculaire, un certain nombre de stratégies de clonage ont été développées pour améliorer la facilité et la vitesse à laquelle les fragments d’ADN peuvent être recombinés. Découvrez les stratégies les plus courantes utilisées dans le clonage moléculaire ou obtenez votre gène souhaité dans le vecteur que vous voulez, de manière simple, avec les clones ORF GenEZ™. Commencez par une recherche de votre gène.
Clonage traditionnel
Le clonage traditionnel, également appelé clonage par PCR, nécessite l’utilisation de la réaction en chaîne par polymérase (PCR) pour amplifier la séquence matrice d’intérêt (généralement le gène d’intérêt) et ajouter des sites de restriction aux extrémités de la séquence. Les enzymes de restriction sont utilisées pour couper à la fois la matrice d’intérêt et le vecteur cible, et l’ADN ligase est utilisée pour joindre les extrémités collantes de la matrice et du vecteur. Le clonage traditionnel permet une manipulation flexible de la séquence d’ADN, ce qui facilite la construction de presque toutes les structures souhaitées. Cependant, les points de contrôle et les procédures d’optimisation requis pour le clonage traditionnel peuvent être lourds, et les réactifs nécessaires peuvent être coûteux.
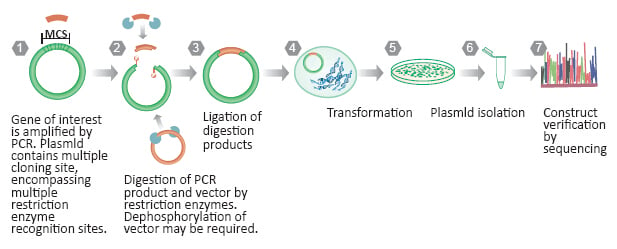
Clonage AT
Le clonage AT est l’une des formes les plus simples de clonage. Dans cette méthode, des vecteurs contenant des surplombs de thymine 5′ sont utilisés pour accepter des produits PCR dans lesquels des surplombs d’adénosine 3′ supplémentaires ont été ajoutés par la nature de l’amplification de la polymérase TAQ. Le clonage TA présente l’avantage de la facilité et de la rapidité, car aucune étape de digestion de restriction n’est nécessaire. En outre, les kits de clonage TA contiennent des tampons de réaction qui contiennent le vecteur pré-mélangé, la ligase et le tampon, ce qui réduit le temps de réaction de ligature à seulement 5 minutes. L’inconvénient de la technologie de clonage TA est que le clonage n’est pas directionnel, ce qui signifie que le gène d’intérêt peut être inséré dans le vecteur cible dans l’orientation sens ou antisens. Normalement, la moitié des transformants ultérieurs contiendront le gène dans le sens et l’autre moitié dans le sens contraire. Cependant, les cellules transformées avec des gènes toxiques peuvent toutes présenter les gènes dans la direction antisens, puisque les cellules contenant les gènes dirigés dans le sens ne survivront pas. En outre, les cellules survivantes contenant des gènes toxiques orientés dans le sens peuvent être mutées pour coder une protéine moins toxique.
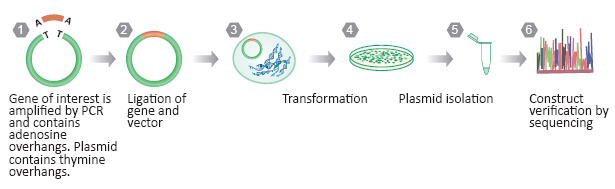
Clonage sans soudure
Les technologies de clonage sans soudure éliminent le besoin d’enzymes de restriction. Cela peut être avantageux lorsqu’un insert contient un certain nombre de sites de restriction dans sa séquence, rendant difficultes l’identification des enzymes de restriction qui ne couperont pas le gène d’intérêt pendant la procédure de clonage. Le clonage sans soudure tire parti de la recombinaison homologue et il existe de nombreuses variantes de cette technique. En général, la procédure consiste à ajouter des séquences d’ancrage d’environ 15 pb de long à l’insert et au vecteur par PCR. Des exonucléases sont utilisées pour mastiquer les séquences de l’insert et du vecteur et l’ADN est joint à l’aide d’enzymes recombinases ou d’ADN ligase. Le clonage sans faille a été simplifié par le développement de kits qui contiennent déjà le vecteur cible et un mélange propriétaire d’enzymes nécessaires à la réaction de recombinaison. Par exemple, le kit GenBuilder™ de GenScript peut cloner des inserts jusqu’à 10 kb en 30 minutes, et peut également être utilisé pour des projets de clonage à haut débit.
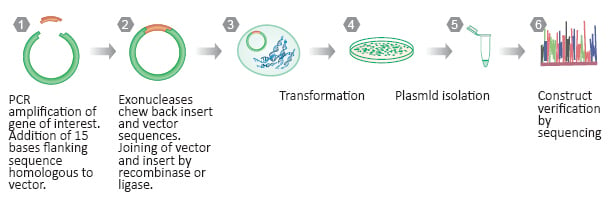
.