Introduzione
I batteri Gram-positivi dei generi Bacillus e Clostridium possono differenziarsi per formare un endospora (spora), una cellula metabolicamente quiescente prodotta in risposta a condizioni che non permettono la crescita cellulare (McKenney et al., 2012). Una volta rilasciata nell’ambiente, la spora sopravvive nel suo stato dormiente per periodi estremamente lunghi, resistendo a una vasta gamma di stress come le alte temperature, la disidratazione, l’assenza di nutrienti e la presenza di sostanze chimiche tossiche (McKenney et al., 2012). Tuttavia, la spora quiescente è in grado di monitorare continuamente l’ambiente e rispondere alla presenza di acqua e nutrienti germinando e originando una cellula vegetativa in grado di crescere e sporulare (McKenney et al., 2012). La resistenza a condizioni non fisiologiche è, in parte, dovuta alle strutture superficiali delle spore. In Bacillus subtilis, il sistema modello per i formatori di spore, la superficie della spora è organizzata in un mantello multistrato e in una crosta, uno strato scoperto di recente che circonda il mantello della spora (McKenney et al., 2012). Le spore di B. subtilis sono caricate negativamente (Huang et al., 2010; Pesce et al., 2014) e hanno una relativa idrofobicità che è in parte dovuta alla glicosilazione di alcune proteine di superficie delle spore (Cangiano et al., 2014; Rusciano et al., 2014). In diverse specie di Bacillus e Clostridium, tra cui B. cereus, B. anthracis, B. megaterium e C. difficile, la struttura più esterna della spora è l’esosporio, uno strato morfologicamente distinto composto da proteine e glicoproteine che circonda il mantello (Díaz-González et al., 2015; Manetsberger et al, 2015b; Stewart, 2015).
La spora batterica è stata proposta come piattaforma per visualizzare proteine eterologhe, con potenziali applicazioni che vanno dallo sviluppo di vaccini mucosali a biocatalizzatori riutilizzabili, strumenti diagnostici e dispositivi di bioremediation (Knecht et al., 2011; Isticato e Ricca, 2014; Ricca et al., 2014). La notevole e ben documentata resistenza della spora (McKenney et al., 2012), l’attitudine di diverse specie che formano spore alla manipolazione genetica (Harwood e Cutting, 1990) e il record di sicurezza di diverse specie (Cutting, 2011; Baccigalupi et al., 2015) supportano l’uso della spora come sistema di visualizzazione e consegna. Due strategie sono state sviluppate per visualizzare le proteine eterologhe sulla superficie della spora. Una strategia ricombinante, basata sulla costruzione di fusioni geniche tra il DNA che codifica per una proteina di superficie della spora selezionata e il DNA che codifica per la proteina da visualizzare, è stata utilizzata negli anni per visualizzare una varietà di proteine eterologhe (Isticato e Ricca, 2014). Un approccio non ricombinante, basato sull’adsorbimento spontaneo tra spore purificate e proteine purificate, è stato utilizzato anche per visualizzare vari enzimi e antigeni (Ricca et al., 2014). I dettagli molecolari che controllano l’adsorbimento delle spore non sono stati completamente chiariti. È noto che l’adsorbimento è più efficiente quando il pH del tampone di legame è acido (pH 4) (Huang et al., 2010; Sirec et al., 2012) e che una combinazione di interazioni elettrostatiche e idrofobiche sono probabilmente coinvolte nell’interazione (Huang et al., 2010; Sirec et al., 2012). È anche noto che le spore mutanti con superfici spore gravemente alterate interagiscono in modo più efficiente rispetto alle spore isogeniche wild type con proteine modello (Sirec et al., 2012; Donadio et al, 2016).
Qui, abbiamo usato una proteina fluorescente, la forma monomerica della proteina fluorescente rossa (mRFP) del corallo Discosoma sp. (Campbell et al., 2002), per valutare se le spore di B. megaterium sono in grado di interagire con e assorbire una proteina eterologa modello. B. megaterium comprende una serie di ceppi morfologicamente distinti che condividono le insolite grandi dimensioni sia delle cellule (lunghezza fino a 4 μm e un diametro di 1,5 μm) che delle spore (lunghezza fino a 3 μm e diametro di 1 μm) (Di Luccia et al., 2016). Le spore di alcuni ceppi di B. megaterium sono circondate da un esosporio, e poiché finora solo le spore di specie prive di esosporio sono state considerate come piattaforme di adsorbimento, non sono disponibili dati sull’impatto dell’esosporio nell’interazione con proteine eterologhe.
Il ceppo QM B1551 è il ceppo meglio caratterizzato di B. megaterium. Questo ceppo porta circa l’11% del suo genoma su sette plasmidi indigeni (Rosso e Vary, 2005; Vary et al., 2007; Eppinger et al., 2011), due dei quali – pBM500 e pBM600 – sono stati identificati come portatori di geni che sono essenziali per la formazione del caratteristico esosporio “a forma di noce” di questo ceppo (Manetsberger et al., 2015a). La composizione proteica dell’esosporio di QM B1551 è ancora poco caratterizzata, con solo alcuni geni che codificano gli ortologhi delle proteine dell’esosporio riconosciute nelle spore di altre specie che sono stati identificati dalle analisi genomiche. Questi includono i geni che codificano le proteine BclA nap, che formano un nap localizzato nelle spore di B. megaterium QM B1551, più un ortologo della proteina BxpB che forma lo strato basale dell’esosporio nelle spore della famiglia B. cereus. Quest’ultimo sembra svolgere un ruolo diverso nelle spore di B. megaterium QM B1551, poiché un ceppo mutante nullo ha mantenuto un esosporio apparentemente normale (Manetsberger et al, 2015a).
In questo lavoro, presentiamo dati che dimostrano che le spore di B. megaterium QM B1551 possono efficientemente adsorbire mRFP, e fornire la prova che le molecole di proteine sono in grado di attraversare la barriera di permeabilità presentata dal exosporium per localizzarsi nello spazio intercoat.
Materiali e metodi
Razze batteriche, spore e produzione di RFP
I ceppi di B. megaterium impiegati in questo studio sono QM B1551 e il suo derivato PV361 senza plasmide (Rosso e Vary, 2005). Il ceppo di B. subtilis utilizzato in questo studio è PY79 (Youngman et al., 1984). La sporulazione di tutti i ceppi di Bacillus è stata indotta dal metodo dell’esaurimento (Cutting e Vander Horn, 1990). Dopo 30 ore di crescita nel terreno Difco Sporulation (DS) a 37°C con agitazione vigorosa, le spore sono state raccolte e purificate come descritto da Nicholson e Setlow (1990) utilizzando un’incubazione notturna in H2O a 4°C per lisare le cellule sporangiali residue. Il numero di spore purificate ottenute è stato misurato mediante conteggio diretto con una camera Bürker sotto un microscopio ottico (Olympus BH-2 con obiettivo 40×).
Per la produzione di mRFP, cellule di Escherichia coli ceppo RH161 (Donadio et al, 2016), recanti il vettore di espressione pRSET-A che porta una fusione in-frame dell’estremità 5′ della regione codificante rfp a sei codoni di istidina sotto il controllo trascrizionale di un promotore T7, sono state coltivate per 18 h a 37°C in 100 ml di terreno di autoinduzione per esprimere la proteina eterologa (Isticato et al., 2010). La proteina RFP legata a His6 è stata purificata in condizioni native utilizzando una colonna His-Trap come raccomandato dal produttore (GE Healthcare Life Science). La proteina purificata è stata desalinizzata utilizzando una colonna PD10 (GE Healthcare Life Science) per rimuovere alte concentrazioni di NaCl e imidazolo.
Reazione di adsorbimento
Non diversamente specificato 5 μg di mRFP ricombinante purificato è stato aggiunto ad una sospensione di spore (5 × 108) in 50 mM Sodium Citrate pH 4.0 a 25 ° C in un volume finale di 200 μl. Dopo 1 h di incubazione, la miscela di legame è stata centrifugata (10 min a 13.000 g) per frazionare mRFP legato-spore nel pellet da mRFP libero nel surnatante.
Western e Dot-Blot Analysis
I pellet di spore da reazioni di adsorbimento sono stati risospesi in 40 μl di tampone di estrazione di cappotto spore (Nicholson e Setlow, 1990; Giglio et al, 2011), incubato a 68 ° C per 1 h per solubilizzare le proteine del cappotto spore e caricato su un gel SDS-PAGE al 12%. Le proteine sono state poi elettro-trasferite su filtri di nitrocellulosa (Amersham Pharmacia Biotech) e utilizzati per l’analisi Western blot come precedentemente riportato (Isticato et al., 2008) utilizzando monoclonale mRFP-riconoscimento anticorpo anti-His (Sigma). Una determinazione quantitativa della quantità di mRFP è stata ottenuta da esperimenti di dot blot confrontando diluizioni seriali di mRFP purificato e supernatante del saggio di legame. I filtri sono stati poi visualizzati con il metodo ECL-prime (Amersham Pharmacia Biotech) e sottoposti ad analisi densitometrica da Quantity One 1-D Analysis Software (Bio-Rad). Dot blot e analisi densitometriche relative sono state eseguite tre volte per verificare la significatività dei risultati.
Microscopia di fluorescenza e immunofluorescenza
Le spore post-adsorbimento sono state risospese in 50 μl di 1x PBS pH 4.0 e 5 μl della sospensione posti su vetrini da microscopio e coperti con un coprioggetto precedentemente trattato per 30 s con poli-L-lisina (Sigma). L’immunofluorescenza è stata eseguita come descritto da Isticato et al. (2013), con le seguenti modifiche: 2,0 × 106 RFP-adsorbito spore di QM B1551 e PV361 B. megaterium ceppi sono stati pretrattati con 1% di sieroalbumina bovina (BSA) – 1x PBS pH 4.0 per 30 min prima di 2 h-incubazione a 4 ° C con gli anticorpi anti-poliHistidina (mouse; Sigma) diluito 1:20 in 1x PBS pH 4.0-1% BSA. Come controllo della specificità di questa tecnica, le spore non adsorbite sono state trattate direttamente con anticorpi anti-His. Dopo tre lavaggi, i campioni sono stati incubati con un anticorpo secondario anti-mouse diluito 64 volte coniugato con isotiocianato di fluoresceina, FITC (Santa Cruz Biotechnology, Inc.) e lavato quattro volte con 1x PBS pH 4.0. I campioni lavati sono stati risospesi in 20 μl di 1x PBS pH 4.0 e 10 μl sono stati analizzati. Tutti i campioni sono stati osservati con un microscopio a fluorescenza Olympus BX51 dotato di un obiettivo UPlan F1 100 × olio; U-MNG o U-MWIBBP cubo-filtri sono stati utilizzati per rilevare l’emissione di fluorescenza rossa di mRFP o l’emissione verde di FITC-anticorpi coniugati, rispettivamente. I tempi di esposizione erano nell’intervallo tra 200 e 3000 ms. Le immagini sono state catturate utilizzando una fotocamera digitale Olympus DP70 e trattati con il software di analisi delle immagini (Olympus) per regolazioni minori di luminosità, contrasto e bilanciamento del colore (McCloy et al., 2014). ImageJ (v1.48, NIH) è stato utilizzato per disegnare un contorno intorno a 80 spore per ogni ceppo e valori di fluorescenza minimi, massimi e medi per pixel sono stati registrati per ogni spora. I valori di intensità di fluorescenza sono stati visualizzati successivamente come box-plot con intervalli di confidenza del 5-95% (McCloy et al., 2014).
Analisi statistica
I risultati delle analisi in dot blot e microscopia a fluorescenza sono le medie di tre esperimenti indipendenti. La significatività statistica è stata determinata dal test t di Student, e il livello di significatività è stato fissato a P < 0.05.
Risultati
mRFP di Discosoma sp. è assorbito da B. megaterium Spores
Per verificare se le spore di B. megaterium QM B1551 erano in grado di adsorbire mRFP, 5 μg della proteina purificata (materiali e metodi) sono stati incubati con 5,0 × 108 spore purificate. La reazione di adsorbimento è stata eseguita in 50 mM citrato di sodio a pH 4,0, come precedentemente descritto (Sirec et al., 2012). Dopo la reazione, le spore sono stati ampiamente lavati con 1x PBS pH 4.0, proteine di superficie spore sono stati estratti come descritto nei materiali e metodi e analizzati mediante western blotting con anti-poliHistidine-Peroxidase anticorpo (Sigma), che riconosce la istidina-tagged N terminus di mRFP. Come mostrato nella Figura 1A, mRFP è stato estratto dalle spore, indicando che è stato assorbito durante la reazione e poi rilasciato dal trattamento di estrazione. Per valutare l’efficienza di adsorbimento, abbiamo analizzato la quantità di mRFP lasciato non legato, cioè, post-adsorbito spore sono stati raccolti per centrifugazione e il surnatante diluito in serie e analizzati da dot blotting (Figura 1B). Un’analisi densitometrica del dot blot (Tabella 1 supplementare) ha mostrato che quando 5 μg di mRFP è stato utilizzato nella reazione di adsorbimento meno dell’1% è stato lasciato non legato, indicando che circa il 99% della proteina eterologa è stato adsorbito a B. megaterium spore.

FIGURA 1. Le spore di Bacillus megaterium QM B1551 adsorbono mRFP. 5 × 108 spore sono state incubate con 5 μg di mRFP purificato e poi i campioni sottoposti a centrifugazione. (A) Le proteine di superficie delle spore sono state estratte dalla frazione del pellet mediante trattamento SDS-DTT, frazionate su SDS-PAGE e analizzate mediante Western blot. Il mRFP purificato (20 μg) è stato usato come marcatore. (B) Il surnatante, contenente il mRFP non legato, è stato diluito in serie e analizzato mediante dot blot (QM B1551). Diluizioni seriali di mRFP purificato (mRFP) sono state usate come standard. (C) Le spore adsorbite con mRFP sono state lavate due volte (W1 e W2) con tampone PBS a pH 3.0 o pH 7.0, o con una soluzione 1M NaCl, 0.1% Triton X-100. Diluizioni seriali di mRFP purificato e mRFP non legato (UN-mRFP) sono stati utilizzati come standard. Immuno-reazioni in tutti i pannelli sono stati eseguiti con mRFP-riconoscimento anticorpo anti-His coniugato con perossidasi di rafano.
Per analizzare se molecole mRFP adsorbiti erano strettamente legati alla superficie della spora, post-adsorbimento spore reazione sono stati lavati due volte con tampone PBS a pH 3.0 o pH 7.0, o con una soluzione 1M NaCl, 0,1% Triton X-100 come precedentemente descritto (Donadio et al., 2016). Come mostrato in Figura 1C, e supportato da analisi densitometrica del dot blot (Tabella 2 supplementare), i lavaggi a pH 3.0 non ha causato alcun rilascio del mRFP adsorbito, mentre i lavaggi a pH 7.0 o con 1M NaCl, 0,1% Triton X-100 causato un minimo, meno dell’1%, rilascio di molecole mRFP. Pertanto, i risultati presentati nella Figura 1 suggeriscono che mRFP è stato efficacemente adsorbito e strettamente legato alle spore di B. megaterium. Per valutare se le molecole mRFP adsorbite dalle spore hanno mantenuto le loro proprietà di fluorescenza, abbiamo eseguito un’analisi di microscopia a fluorescenza. Come mostrato nella Figura 2, le spore di reazione post-adsorbimento erano associate ad un forte segnale di fluorescenza visibile intorno all’intera superficie della spora.
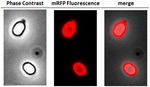
FIGURA 2. Analisi al microscopio a fluorescenza delle spore di B. megaterium QM B1551-mRFP. Le spore QM B1551 incubate con mRFP (5 μg), e successivamente lavate, sono state analizzate al microscopio a fluorescenza. Lo stesso campo di microscopia è stato osservato da contrasto di fase e microscopia a fluorescenza. Scala bar 1 μm. Il pannello di fusione è riportato. Il tempo di esposizione è stato di 200 ms.
L’esosporio ha un ruolo essenziale nell’adsorbimento di mRFP
Strain QM B1551 di B. megaterium contiene sette plasmidi indigeni (Rosso e Vary, 2005; Eppinger et al., 2011) e i geni codificati dai plasmidi sono essenziali per la formazione dell’esosporio (Manetsberger et al., 2015a). PV361 è un ceppo curato da QM B1551 privo di tutti e sette i plasmidi e, di conseguenza, totalmente privo dell’esosporio (Manetsberger et al., 2015a). Abbiamo usato spore del ceppo PV361 per analizzare il ruolo dell’esosporio nell’adsorbimento di mRFP. In parallelo, abbiamo anche usato spore di B. subtilis PY79 che in uno studio precedente hanno dimostrato di adsorbire mRFP (Donadio et al., 2016). Per confrontare l’efficienza di adsorbimento delle spore dei ceppi B. subtilis PY79 e B. megaterium QM B1551 e PV361, abbiamo adsorbito 5 μg di mRFP purificato con 5,0 × 108 spore di ciascuno dei tre ceppi. Dopo le reazioni di adsorbimento le spore sono state raccolte per centrifugazione, le proteine estratte dal trattamento SDS-DTT e analizzate mediante western blotting con mRFP riconoscendo l’anticorpo anti-His. Come mostrato nella Figura 3A, mRFP è stato apparentemente estratto in quantità maggiori dalle spore di QM B1551 che dalle spore degli altri due ceppi. Nel tentativo di quantificare queste differenze apparenti, mRFP non legato dalle reazioni di adsorbimento è stato diluito in serie e analizzato da dot blotting (Figura 3B). Un’analisi densitometrica del dot blot di Figura 3B (Tabella 3 supplementare) ha indicato che le spore PY79 e PV361 hanno adsorbito circa il 90% del totale mRFP mentre le spore QM B1551 hanno adsorbito quasi tutto (oltre il 99%) il mRFP purificato.
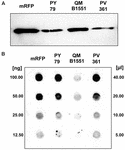
FIGURA 3. Adsorbimento della proteina fluorescente rossa monomerica su spore di B. megaterium QM B1551 e PV361 e B. subtilis PY79. 5 × 108 spore di ogni ceppo sono state incubate con 5 μg di mRFP purificato e poi i campioni sottoposti a centrifugazione. Spore nelle frazioni di pellet sono stati utilizzati per estrarre le proteine di superficie che sono stati successivamente analizzati da western blot (A), mentre i surnatanti sono stati diluiti in serie e analizzati da dot blot (B). Diluizioni seriali di mRFP purificato sono stati utilizzati come standard. Le immuno-reazioni in entrambi i pannelli sono state eseguite con anticorpo anti-His coniugato con perossidasi di rafano.
L’efficienza di adsorbimento delle spore dei tre ceppi è stata analizzata anche mediante microscopia a fluorescenza (Figura 4A e Figura S1 supplementare). Le immagini di microscopia sono state analizzate dal software ImageJ (v1.48, NIH) per eseguire l’analisi quantitativa delle immagini di fluorescenza e la fluorescenza delle spore è stata calcolata come descritto in Materiali e metodi. L’analisi di 80 spore di ogni ceppo indicato un valore medio di fluorescenza per pixel di 52,43 (± 5,94), 20,81 (± 2,71), e 32,33 (± 2.97; unità arbitrarie) per QM B1551, PV361, e PY79, rispettivamente (Figura 4B), conferendo ulteriori prove che le spore QM B1551 adsorbito più mRFP che spore degli altri due ceppi.
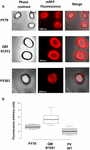
FIGURA 4. Efficienza di adsorbimento di mRFP alle spore di B. megaterium QM B1551 e PV361 e B. subtilis PY79. (A) Immagini al microscopio a fluorescenza di spore PY79, QM B1551 e PV361 dopo l’adsorbimento di mRFP e il lavaggio. I tempi di esposizione sono indicati. Contrasto di fase e sovrapposizioni di fluorescenza rossa sono mostrati (pannello di fusione). Scale bar 1 μm. (B) Box plot che visualizza l’intensità di fluorescenza di ottanta diverse spore di ogni ceppo. I limiti di ogni casella rappresentano il primo e il terzo quartile (25 e 75%) e i valori al di fuori delle caselle rappresentano i valori massimi e minimi. La linea che divide la casella indica il valore mediano per ogni ceppo. Il valore P è inferiore a 0,0001.
In base ai risultati delle figure 3 e 4, concludiamo che l’esosporio, presente in QM B1551 e assente in PV361 ha un ruolo rilevante nell’adsorbimento di mRFP.
Inoltre, i risultati della Figura 4 indicano che le spore di B. subtilis PY79 sono più efficienti di quelle di B. megaterium PV361 nell’adsorbimento di mRFP, mentre il dot blotting riportato nella Figura 3B indicava efficienze di adsorbimento simili per i due ceppi. Crediamo che questa discrepanza sia dovuta a una forte riduzione della fluorescenza quando mRFP è legato a PV361 ma non a PY79 o alle spore QM B1551 (vedi sotto).
Valutazione quantitativa dell’assorbimento di mRFP alle spore di QM B1551
Esperimenti di spot blot (Figura 3B) hanno indicato che quando 5 μg di mRFP purificato sono stati utilizzati in reazioni di assorbimento con 5,0 × 108 spore del ceppo QM B1551 quasi tutta la proteina eterologa è stata legata alla spora. Al fine di definire la quantità massima di mRFP che può essere adsorbita alle spore QM B1551, abbiamo ripetuto le reazioni con concentrazioni crescenti di mRFP purificato, cioè, 5,0 × 108 spore QM B1551 sono stati fatti reagire con 5, 10, 20, 40, 80, e 160 μg di mRFP purificato. Dopo le reazioni le spore sono state raccolte per centrifugazione e i surnatanti contenenti mRFP non legato sono stati diluiti in serie e analizzati mediante dot blotting (Figura 5A). La Figura 5B mostra i risultati di un’analisi densitometrica del dot blot (???), che indica che quando 5-80 μg di mRFP sono stati fatti reagire con 5 × 108 spore, la percentuale di proteina legata alle spore era superiore al 90%. Una diminuzione di mRFP legata è stata osservata quando 160 μg di proteina purificata sono stati utilizzati nella reazione. Tuttavia, anche quando 160 μg di mRFP purificato è stato utilizzato oltre il 60% della proteina è stato assorbito, indicando che 5,0 × 108 spore di QM B1551 può adsorbire circa 100 μg di mRFP.
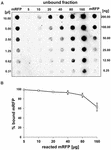
FIGURA 5. Valutazione quantitativa dell’adsorbimento di mRFP alle spore di B. megaterium QM B1551. 5 × 108 spore sono state incubate con 5, 10, 20, 40, 80 e 160 μg di mRFP purificato. Le miscele di reazione sono state successivamente sottoposte a centrifugazione e i surnatanti diluiti in serie e analizzati mediante dot blot (A). Diluizioni seriali di mRFP purificato sono stati utilizzati come standard. Le immunoreazioni in entrambi i pannelli sono state eseguite con anticorpo anti-His coniugato con perossidasi di rafano. (B) Percentuale di mRFP adsorbita alle spore dopo la reazione con quantità definite di mRFP endogeno. Le barre di errore mostrano gli errori standard della media di tre esperimenti e il valore P non è mai superiore a 0.0025.
mRFP si localizza nello spazio tra le spore di B. megaterium QM B1551
Un approccio di microscopia a immuno-fluorescenza è stato impiegato per valutare se le molecole mRFP adsorbite fossero esposte sulla superficie delle spore di B. megaterium QM B1551. Le spore QM B1551 adsorbite con varie quantità di mRFP sono state fatte reagire con un anticorpo monoclonale anti-His che riconosce la mRFP ricombinante, poi con un anticorpo secondario fluorescente anti-mouse (Santa Cruz Biotechnology, Inc.) e osservate al microscopio a fluorescenza (Figura 6). Con la quantità più bassa di mRFP usata in questo esperimento (2 μg) il segnale di fluorescenza mRFP (rosso) è stato osservato tutto intorno alla spora mentre il segnale di immunofluorescenza (verde) era debole e principalmente concentrato ai poli della spora, suggerendo che solo in quei punti mRFP era esposto sulla superficie della spora. Aumentando la quantità di mRFP usata nella reazione il numero di macchie verdi aumentava (5 e 10 μg) e con la più alta quantità di mRFP usata nella reazione (20 μg) un anello verde quasi completo è stato osservato intorno alle spore. Sulla base dei risultati presentati nella Figura 6, abbiamo ipotizzato che le molecole mRFP si infiltrino attraverso l’esosporio e si localizzino nello spazio intercobientale tra il mantello esterno e l’esosporio, cioè, quando viene utilizzata una bassa quantità di mRFP quasi tutte le molecole proteiche sono interne all’esosporio e sono disponibili per l’anticorpo solo in alcuni punti. Aumentando la quantità di mRFP nelle reazioni di adsorbimento, lo spazio tra le spore si “riempie”, finché alla fine più molecole mRFP sono disponibili per l’anticorpo sulla superficie della spora. Questa ipotesi implica che se l’esosporium manca, allora tutta la mRFP dovrebbe essere disponibile per l’anticorpo. Per testare questo, abbiamo confrontato al microscopio a immunofluorescenza un numero uguale di spore di QM B1551 (con esosporio) e di PV361 (senza esosporio) incubate con la stessa quantità di mRFP (5 μg). Quando l’esosporio era presente (QM B1551) mRFP era solo parzialmente disponibile per l’anticorpo e sono state osservate macchie verdi (Figura 7 e Figura supplementare S2). Quando l’esosporio non era presente (PV361) mRFP adsorbito era disponibile per l’anticorpo tutto intorno alla spora e un anello verde completo è stato formato, sostenendo l’ipotesi che mRFP è interno al esosporio in spore QM B1551.
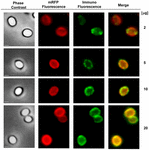
FIGURA 6. Analisi immunofluorescenza di mRFP-adsorbito B. megaterium QM B1551 spore. Aliquote di 5 × 108 spore QM B1551 sono state incubate con concentrazioni variabili di mRFP e sono state successivamente analizzate con microscopia a contrasto di fase, fluorescenza e immunofluorescenza, come descritto nei materiali e metodi. Lo stesso campo di microscopia per ogni reazione è riportato insieme al pannello di unione. Il tempo di esposizione è stato di 200 ms per tutte le immagini. Barra della scala, 1 μm.
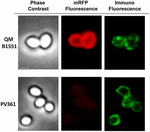
FIGURA 7. Immunofluorescenza di mRFP adsorbita alle spore di B. megaterium QM B1551 e PV361. 5 × 108 spore QM B1551 e PV361 sono stati incubati con 5 μg mRFP e poi analizzati mediante microscopia ad immunofluorescenza, come descritto nei materiali e metodi. Per ogni campo sono mostrati il contrasto di fase e la microscopia ad immunofluorescenza. Il tempo di esposizione era di 200 ms per tutte le immagini. Barra della scala, 1 μm.
Mentre le spore QM B1551 utilizzate negli esperimenti della Figura 7 hanno mostrato un anello rosso fluorescente completo come nella Figura 2, le spore PV361 hanno mostrato un segnale rosso fluorescente molto debole. Con le spore PV361 un segnale rosso è stato osservato solo utilizzando lunghi tempi di esposizione al microscopio a fluorescenza (Figura 4). Poiché mRFP è presente intorno alle spore PV361 (Figure 3 e 7), concludiamo che la fluorescenza mRFP è indebolita quando la proteina è adsorbita alle spore PV361. Ulteriori esperimenti saranno necessari per affrontare completamente questo punto.
Discussione
I risultati principali di questo rapporto sono che le spore di B. megaterium sono estremamente efficienti nell’adsorbire la proteina modello eterologa mRFP, che l’esosporio ha un ruolo importante in questo processo, e che le molecole mRFP si infiltrano attraverso l’esosporio localizzandosi tra il mantello esterno e l’esosporio. Questi risultati espandono il lavoro precedente eseguito sulle spore di B. subtilis e dimostrano che anche le spore di una specie diversa possono essere utilizzate per fornire proteine eterologhe attraverso il metodo di adsorbimento. L’alta efficienza di adsorbimento osservata con le spore di B. megaterium è in parte dovuta alle grandi dimensioni delle sue spore rispetto a quelle di B. subtilis. Infatti, la superficie della spora di B. megaterium è circa 2,5 volte più grande della spora di B. subtilis, con una superficie di 5,33 μm2 (h: 1,60 ± 0,16 w: 0,84 ± 0,07) contro 1,98 μm2 (h: 1,07 ± 0,09 w: 0,48 ± 0,03). Le grandi dimensioni permettono l’adsorbimento di fino a 100 μg di mRFP quando 160 μg di proteina reagiscono con le spore.
L’osservazione che mRFP attraversa l’esosporio indica che è permeabile a mRFP, una proteina di 27 kDa. La permeabilità dell’esosporio non è del tutto sorprendente poiché i germinanti presenti nell’ambiente devono attraversare gli strati esterni della spora per raggiungere i loro recettori, anche se i germinanti sono tipicamente piccole molecole con masse molecolari tipicamente <200 Da. Inoltre, i dati mRFP sono ampiamente in accordo con i risultati di studi precedenti condotti con destrani marcati e molecole correlate (Koshikawa et al., 1984; Nishihara et al., 1989). In quegli studi, l’esosporio di B. megaterium QM B1551 è stato suggerito per rappresentare una barriera di permeabilità alle molecole con peso molecolare superiore a 100 KDa, mentre influenza il passaggio di molecole con massa compresa tra 2 e 40 kDa (Koshikawa et al, 1984; Nishihara et al., 1989).
Un’interessante sfida per il lavoro futuro sarà quella di stabilire il meccanismo o il percorso di infiltrazione che mRFP, e per deduzione altre proteine eterologhe di interesse, prende per entrare nello spazio inter-rivestimento. L’esame al microscopio elettronico a trasmissione di spore sezionate di B. megaterium QM B1551 indica che l’esosporio comprende due “gusci” identici (Manetsberger et al., 2015a), e può essere che l’interfaccia tra ciascuna di queste strutture (descritta come “aperture apicali” nei primi articoli) permetta l’ingresso di molecole relativamente grandi. Discernere la base per l’apparente perdita di fluorescenza mRFP dopo l’adsorbimento alle spore PV361, e se le molecole mRFP sono in grado di infiltrarsi negli strati esterni del mantello, come osservato per le spore di B. subtilis (Donadio et al., 2016), sarà anche di interesse.
Nello studio attuale, si ipotizza che le molecole mRFP attraversano preferenzialmente l’esosporio e si accumulano nello spazio inter-rivestimento tra il mantello esterno e l’esosporio. In questo modello, le molecole mRFP sono adsorbite e visualizzate sulla superficie della spora solo una volta che i siti di adsorbimento (o la capacità volumetrica?) nello spazio intercoat sono sufficientemente occupati. Questo implica che l’approccio di adsorbimento alla visualizzazione della superficie può essere utilizzato con le spore di B. megaterium QM B1551, anche se il sistema dipende dal rapporto spore/proteine utilizzato nelle reazioni di adsorbimento. Poiché vari ceppi di B. megaterium sono stati a lungo utilizzati industrialmente per la produzione di enzimi come amilasi e deidrogenasi, vitamine e molecole antimicrobiche (Vary et al, 2007), i nostri dati suggeriscono una nuova applicazione biotecnologica per la spora di B. megaterium come veicolo per legare e fornire proteine eterologhe.
Contributi degli autori
ML e GD – hanno eseguito la maggior parte degli esperimenti; RA e AS – hanno contribuito all’interpretazione dei dati per il lavoro; GiC, LB, e GrC – hanno contribuito alla stesura del lavoro e alla sua revisione critica; ER – ha contribuito alle discussioni e ai suggerimenti durante il lavoro e ha contribuito alla stesura del manoscritto; RI – ha condotto il lavoro e ha contribuito alla scrittura del manoscritto. Tutti gli autori hanno letto e approvato il manoscritto finale.
Finanziamento
Questo lavoro è stato sostenuto da una borsa di studio (STAR Linea1- 2014) dell’Università Federico II di Napoli e Compagnia di San Paolo, Italia a RI.
Dichiarazione di conflitto di interessi
Gli autori dichiarano che la ricerca è stata condotta in assenza di relazioni commerciali o finanziarie che possano essere interpretate come un potenziale conflitto di interessi.
Materiale supplementare
Il materiale supplementare per questo articolo può essere trovato online su: https://www.frontiersin.org/article/10.3389/fmicb.2016.01752/full#supplementary-material
FIGURA S1 | Immagini a tutto campo dell’analisi di microscopia a fluorescenza riportata in Figura 4. Lo stesso campo di microscopia è stato osservato da contrasto di fase e microscopia a fluorescenza. Scala bar 5 μm. Il pannello di fusione è riportato. Il tempo di esposizione è indicato.
FIGURA S2 | Immagini dell’intero campo di analisi Immunofluorescenza riportato in Figura 7. Lo stesso campo di microscopia è stato osservato da contrasto di fase e microscopia a fluorescenza. Scala bar 5 μm. Il tempo di esposizione era 200 ms per tutte le immagini.
TABELLA S1 | Analisi densitometrica degli esperimenti dot blot con i surnatanti della reazione di adsorbimento con spore QM B1551 (Figura 1B).
Tabella S2 | Analisi densitometrica degli esperimenti di dot blot con i surnatanti di vari lavaggi dopo la reazione di adsorbimento (Figura 1C).
Tabella S3 | Analisi densitometrica degli esperimenti di dot blot con i surnatanti della reazione di adsorbimento eseguita con spore dei ceppi PY79, QM B1551, e PV361 (Figura 3B).
TABELLA S4 | Analisi densitometrica degli esperimenti di dot blot con i surnatanti della reazione di adsorbimento eseguita con diverse quantità di mRFP.
Baccigalupi, L., Ricca, E., e Ghelardi, E. (2015). “I probiotici non-LAB: formatori di spore”, in Probiotici e prebiotici: Current Research and Future Trends, eds K. Venema and A. P. do Carmo (Norfolk, VA: Caister Academic Press), 93-103. doi: 10.21775/9781910190098.06
CrossRef Full Text | Google Scholar
Campbell, R. E, Tour, O., Palmer, A. E., Steinbach, P. A., Baird, G. S., Zacharias, D. A., et al. (2002). Una proteina fluorescente rossa monomerica. Proc. Natl. Acad. Sci. U.S.A. 99, 7877-7882. doi: 10.1073/pnas.082243699
PubMed Abstract | CrossRef Full Text | Google Scholar
Cangiano, G., Sirec, T., Panarella, C., Isticato, R., Baccigalupi, L., De Felice, M., et al. (2014). I prodotti del gene sps influenzano la germinazione, l’idrofobicità e l’adsorbimento delle proteine delle spore di Bacillus subtilis. Appl. Environ. Microbiol. 80, 7293-7302. doi: 10.1128/AEM.02893-14
PubMed Abstract | CrossRef Full Text | Google Scholar
Taglio, S. M. (2011). Bacillus probiotici. Cibo Microbiol. 28, 214-220. doi: 10.1016/j.fm.2010.03.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Cutting, S., and Vander Horn, P. B. (1990). “Analisi genetica”, in Molecular Biological Methods for Bacillus, eds C. Harwood and S. Cutting (Chichester: John Wiley and Sons), 27-74.
Google Scholar
Díaz-González, F., Milano, M., Olguin-Araneda, V., Pizarro-Cerda, J., Castro-Córdova, P., Tzeng, S. C., et al. (2015). Composizione proteica dello strato più esterno exosporium-like delle spore di Clostridium difficile 630. J. Proteom. 123, 1-13. doi: 10.1016/j.jprot.2015.03.035
PubMed Abstract | CrossRef Full Text | Google Scholar
Di Luccia, B., D’Apuzzo, E., Varriale, F., Baccigalupi, L., Ricca, E., and Pollice, A. (2016). Bacillus megaterium SF185 induce vie di stress e influenza la distribuzione del ciclo cellulare delle cellule epiteliali intestinali umane. Benef. Microb. 7, 609-620. doi: 10.3920/BM2016.0020
PubMed Abstract | CrossRef Full Text | Google Scholar
Donadio, G., Lanzilli, M., Sirec, T., Ricca, E., and Isticato, R. (2016) Adsorbimento e localizzazione di proteine su spore wild type e mutanti di Bacillus subtilis. Microb. Cell Fact. 15:153. doi: 10.1186/s12934-016-0551-2
PubMed Abstract | CrossRef Full Text
Eppinger, M., Bunk, B., Johns, M. A., Edirisinghe, J. N., Kutumbaka, K. K., Koenig, S. S., et al. (2011). Sequenze del genoma dei ceppi di Bacillus megaterium biotecnologicamente importanti QM B1551 e DSM319. J. Bacteriol. 193, 4199-4213. doi: 10.1128/JB.00449-11
PubMed Abstract | CrossRef Full Text | Google Scholar
Giglio, R., Fani, R., Isticato, R., De Felice, M., Ricca, E., e Baccigalupi, L. (2011). Organizzazione ed evoluzione dei geni cotG e cotH di Bacillus subtilis. J. Bacteriol. 193, 6664-6673. doi: 10.1128/jb.06121-11
PubMed Abstract | CrossRef Full Text | Google Scholar
Harwood, C. R., e taglio, S. M. (1990). Metodi biologici molecolari per Bacillus. Chichester: John Wiley and Sons.
Google Scholar
Huang, J. M., Hong, H. A., Van Tong, H., Hoang, T. H., Brisson, A., and Cutting, S. M. (2010). Consegna mucosale di antigeni utilizzando l’adsorbimento alle spore batteriche. Vaccine 28, 1021-1030. doi: 10.1016/j.vaccine.2009.10.127
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., Pelosi, A., De Felice, M., e Ricca, E. (2010). CotE si lega a CotC e CotU e media la loro interazione durante la formazione del mantello della spora in Bacillus subtilis. J. Bacteriol. 192, 949-954. doi: 10.1128/JB.01408-09
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., Pelosi, A., Zilhão, R., Baccigalupi, L., Henriques, A. O., De Felice, M., et al. CotC-CotU eterodimerizzazione durante l’assemblaggio del cappotto della spora di Bacillus subtilis. J. Bacteriol. 190, 1267-1275. doi: 10.1128/JB.01425-07
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., e Ricca, E. (2014). Visualizzazione della superficie delle spore. Microbiol. Spectr. 2, 351-366. doi: 10.1128/microbiolspec.TBS-0011-2012
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., Sirec, T., Treppiccione, L., Maurano, F., De Felice, M., Rossi, M., et al. (2013). Visualizzazione non ricombinante della subunità B della tossina termolabile di Escherichia coli su spore wild type e mutanti di Bacillus subtilis. Microb. Cell Fact. 12:98. doi: 10.1186/1475-2859-12-98
PubMed Abstract | CrossRef Full Text | Google Scholar
Knecht, L. D., Pasini, P., e Daunert, S. (2011). Spore batteriche come piattaforme per applicazioni bioanalitiche e biomediche. Anal. Bioanal. Chem. 400, 977-989. doi: 10.1007/s00216-011-4835-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Koshikawa, T., Beaman, T. C., Pankratz, H. S., Nakashio, S., Corner, T. R., e Gerhardt, P. (1984). Resistenza, germinazione e correlati di permeabilità delle spore di Bacillus megaterium successivamente private degli strati di tegumento. J. Bacteriol. 159, 624-632.
PubMed Abstract | Google Scholar
Manetsberger, J., Hall, E. A. H., e Christie, G. (2015a). I geni codificati dal plasmide influenzano l’assemblaggio e la morfologia dell’esosporio nelle spore di Bacillus megaterium QM B1551. FEMS Microbiol. Lett. 362:fnv147. doi: 10.1093/femsle/fnv147
PubMed Abstract | CrossRef Full Text | Google Scholar
Manetsberger, J., Manton, J. D., Erdelyi, M. J., Lin, H., Rees, D., Christie, G., et al. (2015b). La microscopia di localizzazione ellissoidale infrange la dimensione e l’ordine degli strati di proteine nei cappotti delle spore di Bacillus. Biophys. J. 109, 2058-2066. doi: 10.1016/j.bpj.2015.09.023
PubMed Abstract | CrossRef Full Text | Google Scholar
McCloy, R., Rogers, S., Caldon, E., Lorca, T., Castro, A., e Burgess, A. (2014). Inibizione parziale di Cdk1 in fase G2 scavalca il SAC e disaccoppia gli eventi mitotici. Cell Cycle 13, 1400-1412. doi: 10.4161/cc.28401
PubMed Abstract | CrossRef Full Text | Google Scholar
McKenney, P. T., Driks, A., e Eichemberger, P. (2012). L’endospora di Bacillus subtilis: assemblaggio e funzioni del cappotto multistrato. Nat. Rev. Microbiol. 11, 33-44. doi: 10.1038/nrmicro2921
PubMed Abstract | CrossRef Full Text | Google Scholar
Nicholson, W. L., and Setlow, P. (1990). “Sporulazione, germinazione e out-growth,” in Molecular biological methods for Bacillus, eds C. Harwood and S. Cutting (Chichester: John Wiley and Sons), 391-450.
Google Scholar
Nishihara, T., Takubo, Y., Kawamata, E., Koshikawa, T., Ogaki, J., e Kondo, M. (1989). Ruolo del cappotto esterno nella resistenza della spora di Bacillus megaterium. J. Biochem. 106, 270-273.
PubMed Abstract | Google Scholar
Pesce, G., Rusciano, G., Sirec, T., Isticato, R., Sasso, A., e Ricca, E. (2014). Misure di carica superficiale e coefficiente idrodinamico di spore di Bacillus subtilis mediante pinzette ottiche. Colloids Surf. B Biointerfaces 116C, 568-575. doi: 10.1016/j.colsurfb.2014.01.039
PubMed Abstract | CrossRef Full Text | Google Scholar
Ricca, E., Baccigalupi, L., Cangiano, G., De Felice, M., e Isticato, R. (2014). Consegna del vaccino mucosale mediante spore non ricombinanti di Bacillus subtilis. Microb. Cell Fact. 13, 115. doi: 10.1186/s12934-014-0115-2
PubMed Abstract | CrossRef Full Text | Google Scholar
Rosso, M. L., e Vary, P. S. (2005). Distribuzione dei plasmidi di Bacillus megaterium QM B1551 tra altri ceppi di B. megaterium e specie di Bacillus. Plasmid 53, 205-217. doi: 10.1016/j.plasmid.2004.10.005
PubMed Abstract | CrossRef Full Text | Google Scholar
Rusciano, G. Zito, G., Isticato, R., Sirec, T., Ricca, E., Bailo, E., e Sasso, A. (2014). Imaging chimico su nanoscala di spore di Bacillus subtilis combinando tip-enhanced Raman scattering e strumenti statistici avanzati. ACS Nano 8, 12300-12309. doi: 10.1021/nn504595k
PubMed Abstract | CrossRef Full Text | Google Scholar
Sirec, T., Strazzulli, A., Isticato, R., De Felice, M., Moracci, M., e Ricca, E. (2012). Assorbimento di beta-galattosidasi di Alicyclobacillus acidocaldarius su spore wild type e mutanti di Bacillus subtilis. Microb. Cell Fact. 11:100. doi: 10.1186/1475-2859-11-100
PubMed Abstract | CrossRef Full Text | Google Scholar
Stewart, G. C. (2015). Lo strato Exosporium delle spore batteriche: una connessione con l’ambiente e l’ospite infetto. Microbiol. Mol. Biol. Rev. 79, 437-457. doi: 10.1128/MMBR.00050-15
PubMed Abstract | CrossRef Full Text | Google Scholar
Vary, P. S., Biedendieck, R., Fuerch, T., Meinhardt, F., Rohde, M., Deckwer, W. -D., et al. (2007). Bacillus megaterium: da semplice batterio del suolo a ospite per la produzione industriale di proteine. Appl. Microbiol. Biotechnol. 76, 957-967 doi: 10.1007/s00253-007-1089-3
PubMed Abstract | CrossRef Full Text | Google Scholar
Youngman, P., Perkins, J. B., and Losick, R. (1984). Un nuovo metodo per la clonazione rapida in Escherichia coli del DNA cromosomico di Bacillus subtilis adiacente all’inserzione Tn917. Mol. Gen. Genet. 195, 424-433. doi: 10.1007/BF00341443
CrossRef Full Text | Google Scholar