Opgave van het proces van prokaryote transcriptie en translatie
De prokaryoten, waartoe bacteriën en archaea behoren, zijn meestal eencellige organismen die, per definitie, geen membraangebonden kernen en andere organellen hebben. Een bacterieel chromosoom is een covalent gesloten cirkel die, in tegenstelling tot eukaryote chromosomen, niet is georganiseerd rond histon-eiwitten. Het centrale deel van de cel waarin prokaryotisch DNA zich bevindt, wordt de nucleoïde genoemd. Bovendien hebben prokaryoten vaak een overvloed aan plasmiden, kortere cirkelvormige DNA-moleculen die slechts één of enkele genen kunnen bevatten. Plasmiden kunnen onafhankelijk van het bacteriële chromosoom worden overgedragen tijdens de celdeling en dragen vaak eigenschappen zoals resistentie tegen antibiotica. Door deze unieke kenmerken zijn transcriptie en genregulatie enigszins verschillend tussen prokaryote en eukaryote cellen.
Leerdoelen
- Begrijpen wat de basisstappen zijn bij de transcriptie van DNA in RNA in prokaryote cellen
- Begrijpen wat de basisprincipes zijn van prokaryotische translatie en hoe het verschilt van eukaryotische translatie
Prokaryotische Transcriptie
Initiëring van Transcriptie in Prokaryoten
Prokaryoten hebben geen membraan-ingesloten kernen. Daarom kunnen de processen van transcriptie, translatie en mRNA-afbraak alle gelijktijdig plaatsvinden. Het intracellulaire niveau van een bacterieel eiwit kan snel worden verhoogd door meervoudige transcriptie- en translatiegebeurtenissen die gelijktijdig op hetzelfde DNA-sjabloon plaatsvinden. Prokaryotische transcriptie omvat vaak meer dan één gen en produceert polycistronische mRNA’s die meer dan één eiwit specificeren.
Onze discussie hier zal transcriptie illustreren door dit proces te beschrijven in Escherichia coli, een goed bestudeerde bacteriesoort. Hoewel er enkele verschillen bestaan tussen transcriptie in E. coli en transcriptie in archaea, kan een begrip van E. coli-transcriptie worden toegepast op vrijwel alle bacteriesoorten.
Prokaryotisch RNA-polymerase
Prokaryoten gebruiken hetzelfde RNA-polymerase om al hun genen te transcriberen. In E. coli bestaat het polymerase uit vijf polypeptide-subeenheden, waarvan er twee identiek zijn. Vier van deze subeenheden, die α, α, β en β′ worden genoemd, vormen het polymerase-kernenzym. Deze subeenheden verzamelen zich telkens wanneer een gen wordt getranscribeerd, en zij ontmantelen zich wanneer de transcriptie voltooid is. Elke subeenheid heeft een unieke rol; de twee α-subeenheden zijn nodig om de polymerase op het DNA te monteren; de β-subeenheid bindt zich aan het ribonucleosidetrifosfaat dat deel zal gaan uitmaken van het “pasgeboren” mRNA-molecuul; en de β′ bindt de DNA-sjabloonstreng. De vijfde subeenheid, σ, is alleen betrokken bij het initiëren van de transcriptie. Zij verleent transcriptionele specificiteit zodat de polymerase begint met de synthese van mRNA vanaf een geschikte initiatieplaats. Zonder σ zou het kernenzym transcriberen vanaf willekeurige plaatsen en mRNA-moleculen produceren die eiwitgebrabbel specificeren. Het polymerase bestaande uit alle vijf subeenheden wordt het holoenzym genoemd (een holoenzym is een biochemisch actieve verbinding bestaande uit een enzym en zijn co-enzym).
Prokaryotic Promoters

Figuur 1. De σ-subeenheid van prokaryotisch RNA-polymerase herkent consensussequenties die in het promotorgebied stroomopwaarts van de transcriptiestartplaats worden aangetroffen. De σ-subeenheid dissocieert van de polymerase nadat de transcriptie is begonnen.
Een promotor is een DNA-sequentie waaraan de transcriptie-machinerie zich bindt en de transcriptie initieert. In de meeste gevallen bevinden de promotors zich stroomopwaarts van de genen die zij reguleren. De specifieke sequentie van een promotor is zeer belangrijk omdat deze bepaalt of het corresponderende gen altijd, soms, of zelden wordt getranscribeerd. Hoewel promotors variëren tussen prokaryote genomen, zijn een paar elementen geconserveerd. In de -10 en -35 regio’s stroomopwaarts van de initiatieplaats zijn er twee consensussequenties van de promotor, of regio’s die vergelijkbaar zijn in alle promotors en in verschillende bacteriesoorten (figuur 1).
De -10 consensussequentie, de -10 regio genoemd, is TATAAT. De -35 sequentie, TTGACA, wordt herkend en gebonden door σ. Zodra deze interactie tot stand is gebracht, binden de subeenheden van het kernenzym zich aan de plaats. De A-T-rijke -10 regio vergemakkelijkt het afwikkelen van het DNA-sjabloon, en er worden verschillende fosfodiësterbindingen gemaakt. De transcriptie-initiatiefase eindigt met de productie van abortieve transcripten, dat zijn polymeren van ongeveer 10 nucleotiden die worden gemaakt en vrijgegeven.
Verlenging en Beëindiging bij Prokaryoten
De transcriptie-verlengingsfase begint met het loslaten van de σ-subeenheid van het polymerase. Door de dissociatie van σ kan het kernenzym langs het DNA-sjabloon gaan en mRNA synthetiseren in de 5′ tot 3′ richting met een snelheid van ongeveer 40 nucleotiden per seconde. Tijdens de elongatie wordt het DNA voortdurend voor het kernenzym afgewikkeld en achter het kernenzym weer opgewikkeld (figuur 2). De basenparing tussen DNA en RNA is niet stabiel genoeg om de stabiliteit van de mRNA-synthesecomponenten te handhaven. In plaats daarvan fungeert het RNA-polymerase als een stabiele link tussen het DNA-sjabloon en de ontluikende RNA-strengen om ervoor te zorgen dat de elongatie niet voortijdig wordt onderbroken.

Figuur 2. Klik voor een grotere afbeelding. Tijdens de elongatie volgt het prokaryotische RNA-polymerase het DNA-sjabloon, synthetiseert mRNA in de 5′ tot 3′ richting, en rolt het DNA af en terug terwijl het wordt afgelezen.
Prokaryotische eindsignalen
Als een gen eenmaal getranscribeerd is, moet het prokaryotische polymerase de opdracht krijgen zich van het DNA-sjabloon los te maken en het nieuw gemaakte mRNA vrij te maken. Afhankelijk van het gen dat getranscribeerd wordt, zijn er twee soorten terminatiesignalen. Het ene is gebaseerd op eiwitten en het andere op RNA. Rho-afhankelijke terminatie wordt gecontroleerd door het rho-eiwit, dat achter het polymerase op de groeiende mRNA-keten meeloopt. Tegen het einde van het gen stuit het polymerase op een reeks G-nucleotiden op het DNA-sjabloon en stopt. Als gevolg daarvan botst het rho-eiwit tegen het polymerase. De interactie met rho bevrijdt het mRNA uit de transcriptiebel.
Rho-onafhankelijke beëindiging wordt gecontroleerd door specifieke sequenties in de DNA-sjabloonstreng. Als het polymerase het einde van het getranscribeerde gen nadert, stuit het op een gebied dat rijk is aan C-G nucleotiden. Het mRNA vouwt terug op zichzelf, en de complementaire C-G nucleotiden binden zich aan elkaar. Het resultaat is een stabiele haarspeld die het polymerase doet afslaan zodra het begint met de transcriptie van een gebied dat rijk is aan A-T-nucleotiden. De complementaire U-A-regio van het mRNA-transcript vormt slechts een zwakke interactie met het sjabloon-DNA. Dit, in combinatie met het vastgelopen polymerase, veroorzaakt voldoende instabiliteit voor het kernenzym om los te breken en het nieuwe mRNA-transcript vrij te maken.
Up het moment van beëindiging is het transcriptieproces voltooid. Tegen de tijd dat het proces wordt beëindigd, is het prokaryotisch transcript reeds gebruikt voor de synthese van talrijke kopieën van het gecodeerde eiwit, omdat deze processen gelijktijdig kunnen plaatsvinden. De vereniging van transcriptie, translatie en zelfs mRNA-afbraak is mogelijk omdat al deze processen in dezelfde 5′ tot 3′ richting verlopen, en omdat er in de prokaryote cel geen membraneuze compartimentering is (figuur 3). De aanwezigheid van een kern in eukaryote cellen verhindert daarentegen gelijktijdige transcriptie en translatie.

Figuur 3. Meerdere polymerasen kunnen één bacterieel gen transcriberen terwijl talrijke ribosomen tegelijkertijd de mRNA-transcripten omzetten in polypeptiden. Op deze manier kan een specifiek eiwit snel een hoge concentratie in de bacteriële cel bereiken.
Bekijk deze BioStudio-animatie om het proces van prokaryotische transcriptie te zien.
Praktijkvragen
Welke subeenheid van het E. coli polymerase verleent specificiteit aan transcriptie?
- α
- β
- β′
- σ
De -10 en -35 regio’s van prokaryote promotors worden consensus sequenties genoemd omdat ________.
- ze in alle bacteriesoorten identiek zijn
- ze in alle bacteriesoorten vergelijkbaar zijn
- ze in alle organismen bestaan
- ze in alle organismen dezelfde functie hebben
Prokaryotische translatie
De translatie is vergelijkbaar in prokaryoten en eukaryoten. Hier zullen we onderzoeken hoe translatie plaatsvindt in E. coli, een representatieve prokaryoot, en eventuele verschillen tussen bacteriële en eukaryotische translatie specificeren.
Initiatie
De initiatie van eiwitsynthese begint met de vorming van een initiatiecomplex. In E. coli bestaat dit complex uit het kleine 30S ribosoom, het mRNA-sjabloon, drie initiatiefactoren die helpen het ribosoom correct te assembleren, guanosinetrifosfaat (GTP) dat als energiebron fungeert, en een speciaal initiator-tRNA dat N-formyl-methionine (fMet-tRNAfMet) bevat (figuur 4). Het initiator-tRNA staat in wisselwerking met het startcodon AUG van het mRNA en bevat een geformyleerd methionine (fMet). Omdat fMet een rol speelt bij de initiatie, wordt het ingebracht aan het begin (N-terminus) van elke polypeptideketen die door E. coli wordt gesynthetiseerd. In E. coli mRNA is er een leader-sequentie stroomopwaarts van het eerste AUG codon, de Shine-Dalgarno-sequentie (ook bekend als de ribosomale bindingsplaats AGGAGG), die via complementaire basenparen een interactie aangaat met de rRNA-moleculen waaruit het ribosoom is opgebouwd. Deze interactie verankert de 30S ribosomale subeenheid op de juiste plaats op het mRNA-sjabloon. Op dit punt bindt de 50S ribosomale subeenheid zich vervolgens aan het initiatiecomplex, waardoor een intact ribosoom wordt gevormd.
In eukaryoten verloopt de vorming van het initiatiecomplex op soortgelijke wijze, met de volgende verschillen:
- De initiator tRNA is een ander gespecialiseerd tRNA dat methionine bevat, genaamd Met-tRNAi
- In plaats van binding aan het mRNA op de Shine-Dalgarno sequentie, herkent het eukaryotische initiatie complex de 5′ cap van het eukaryotische mRNA, en volgt dan het mRNA in de 5′ tot 3′ richting, totdat het AUG startcodon wordt herkend. Op dit punt bindt de 60S subeenheid aan het complex van Met-tRNAi, mRNA, en de 40S subeenheid.

Figuur 4. De translatie in bacteriën begint met de vorming van het initiatiecomplex, dat bestaat uit de kleine ribosomale subeenheid, het mRNA, het initiator-tRNA dat N-formyl-methionine bevat, en initiatiefactoren. Dan bindt de 50S subeenheid zich en vormt een intact ribosoom.
Elongatie
In prokaryoten en eukaryoten zijn de grondbeginselen van elongatie van translatie hetzelfde. In E. coli vormt de binding van de 50S ribosomale subeenheid om het intacte ribosoom te produceren drie functioneel belangrijke ribosomale sites: De A (aminoacyl) plaats bindt inkomende geladen aminoacyl tRNA’s. De P (peptidyl)-plaats bindt geladen tRNA’s met aminozuren die peptidebindingen met de groeiende polypeptideketen hebben gevormd, maar nog niet van hun corresponderende tRNA zijn losgemaakt. De E (exit) site maakt gedissocieerde tRNA’s vrij, zodat zij weer kunnen worden opgeladen met vrije aminozuren. Er is één opmerkelijke uitzondering op deze assemblagelijn van tRNA’s: Tijdens de vorming van het initiatiecomplex gaat bacterieel fMet-tRNAfMet of eukaryotisch Met-tRNAi direct de P-site binnen zonder eerst de A-site binnen te gaan, waardoor een vrije A-site ontstaat die klaar is om het tRNA te accepteren dat correspondeert met het eerste codon na de AUG.
Elongatie verloopt met single-codon bewegingen van het ribosoom, die elk een translocation event worden genoemd. Tijdens elke translocatiegebeurtenis komen de geladen tRNA’s binnen op de A-locatie, verschuiven dan naar de P-locatie, en tenslotte naar de E-locatie voor verwijdering. Ribosomale bewegingen, of stappen, worden geïnduceerd door conformatieveranderingen die het ribosoom drie basen opschuiven in de 3′-richting. Peptide bindingen vormen zich tussen de aminogroep van het aminozuur dat vastzit aan het A-site tRNA en de carboxylgroep van het aminozuur dat vastzit aan het P-site tRNA. De vorming van elke peptidebinding wordt gekatalyseerd door peptidyltransferase, een op RNA gebaseerd ribozyme dat in de 50S ribosomale subeenheid is geïntegreerd. Het aminozuur dat aan het P-site tRNA is gebonden, is ook verbonden met de groeiende polypeptideketen. Wanneer het ribosoom over het mRNA stapt, komt het voormalige P-site tRNA in de E-site, maakt zich los van het aminozuur, en wordt uitgestoten. Verscheidene van de stappen tijdens elongatie, waaronder binding van een geladen aminoacyl tRNA aan de A site en translocatie, vereisen energie afkomstig van GTP hydrolyse, die wordt gekatalyseerd door specifieke elongatiefactoren. Verbazingwekkend genoeg heeft het vertaalapparaat van E. coli slechts 0,05 seconden nodig om elk aminozuur toe te voegen, wat betekent dat een eiwit van 200 aminozuren in slechts 10 seconden kan worden vertaald.
Terminatie
De translatie wordt beëindigd wanneer een nonsense codon (UAA, UAG, of UGA) wordt aangetroffen waarvoor geen complementair tRNA bestaat. Wanneer deze nonsense codons op één lijn liggen met de A-site, worden ze herkend door releasefactoren in prokaryoten en eukaryoten, die ertoe leiden dat het P-site aminozuur loskomt van zijn tRNA, waardoor het nieuw gemaakte polypeptide vrijkomt. De kleine en grote ribosomale subeenheden scheiden zich van het mRNA en van elkaar; zij worden vrijwel onmiddellijk gerekruteerd in een ander complex voor translatie-initiatie.
Samenvattend zijn er verschillende hoofdkenmerken die prokaryote genexpressie onderscheiden van die bij eukaryoten. Deze worden geïllustreerd in figuur 5 en opgesomd in tabel 1.
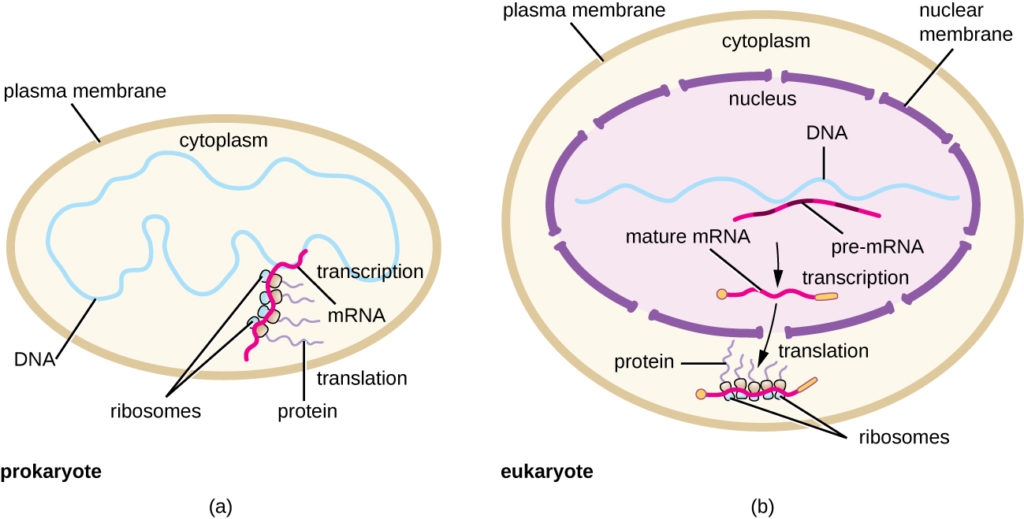
Figuur 5. (a) Bij prokaryoten vinden transcriptie en translatie gelijktijdig plaats in het cytoplasma, zodat de cel snel kan reageren op een omgevingsprikkel. (b) Bij eukaryoten is de transcriptie gelokaliseerd in de celkern en de translatie in het cytoplasma, waardoor deze processen gescheiden zijn en verwerking van het RNA nodig is voor de stabiliteit.
| Tabel 1. Vergelijking van translatie in Bacteria Versus Eukaryoten | ||
|---|---|---|
| Property | Bacteria | Eukaryoten |
| Ribosomes | 70S
|
80S
|
| Amino zuur gedragen door initiator tRNA | fMet | Met |
| Shine-Dalgarno-sequentie in mRNA | Aanwezig | Absistent |
| Simultane transcriptie en translatie | Ja | Nee |
Check Your Understanding
Beantwoord de onderstaande vraag (vragen) om te zien hoe goed u de onderwerpen uit het vorige deel begrijpt. Deze korte quiz telt niet mee voor uw cijfer en u kunt hem een onbeperkt aantal keren overdoen.
Gebruik deze quiz om uw begrip te toetsen en te beslissen of u (1) de vorige paragraaf verder wilt bestuderen of (2) verder wilt gaan met de volgende paragraaf.