Zarys procesu transkrypcji i translacji u prokariotów
Prokariota, do którego zaliczamy bakterie i archaidy, to w większości jednokomórkowe organizmy, które z definicji nie posiadają jąder związanych z błoną i innych organelli. Chromosom bakteryjny jest kowalencyjnie zamkniętym kołem, które, w przeciwieństwie do chromosomów eukariotycznych, nie jest zorganizowane wokół białek histonowych. Centralny obszar komórki, w którym znajduje się prokariotyczne DNA, nazywany jest nukleoidem. Ponadto u prokariotów często występują duże ilości plazmidów, które są krótszymi, kolistymi cząsteczkami DNA, mogącymi zawierać tylko jeden lub kilka genów. Plazmidy mogą być przenoszone niezależnie od chromosomu bakteryjnego podczas podziału komórki i często przenoszą takie cechy, jak oporność na antybiotyki. Ze względu na te unikalne cechy, transkrypcja i regulacja genów jest nieco inna w komórkach prokariotycznych i eukariotycznych.
Cele nauczania
- Zrozumienie podstawowych kroków w transkrypcji DNA na RNA w komórkach prokariotycznych
- Zrozumienie podstaw prokariotycznej translacji i czym się różni od translacji eukariotycznej
Prokariotyczna transkrypcja
Początkowanie transkrypcji u prokariotów
Prokariota nie ma jądra otoczonego błoną.zamkniętych w błonie jąder. Dlatego procesy transkrypcji, translacji i degradacji mRNA mogą zachodzić jednocześnie. Wewnątrzkomórkowy poziom białka bakteryjnego może być szybko wzmocniony przez wiele zdarzeń transkrypcji i translacji zachodzących jednocześnie na tym samym szablonie DNA. Transkrypcja u prokariotów często obejmuje więcej niż jeden gen i produkuje polirecystronowe mRNA, które określa więcej niż jedno białko.
Nasza dyskusja tutaj będzie przykładem transkrypcji opisując ten proces u Escherichia coli, dobrze zbadanego gatunku bakterii. Chociaż istnieją pewne różnice między transkrypcją u E. coli a transkrypcją u archaea, zrozumienie transkrypcji u E. coli może być zastosowane do praktycznie wszystkich gatunków bakterii.
Prokariotyczna polimeraza RNA
Prokariota używa tej samej polimerazy RNA do transkrypcji wszystkich swoich genów. W E. coli, polimeraza składa się z pięciu podjednostek polipeptydowych, z których dwie są identyczne. Cztery z tych podjednostek, oznaczone jako α, α, β i β′ składają się na rdzeń enzymu polimerazy. Podjednostki te gromadzą się podczas każdej transkrypcji genu i rozpadają się po zakończeniu transkrypcji. Każda podjednostka ma swoją unikalną rolę; dwie podjednostki α są niezbędne do złożenia polimerazy na DNA; podjednostka β wiąże się z trifosforanem rybonukleozydu, który stanie się częścią powstającej „niedawno urodzonej” cząsteczki mRNA; a β′ wiąże się z nicią szablonu DNA. Piąta podjednostka, σ, jest zaangażowana tylko w inicjację transkrypcji. Nadaje ona specyficzność transkrypcyjną w taki sposób, że polimeraza rozpoczyna syntezę mRNA od odpowiedniego miejsca inicjacji. Bez σ, enzym rdzeniowy transkrybowałby z przypadkowych miejsc i produkowałby cząsteczki mRNA, które określałyby białkowy bełkot. Polimeraza złożona z wszystkich pięciu podjednostek nazywana jest holoenzymem (holoenzym to aktywny biochemicznie związek składający się z enzymu i jego koenzymu).
Prokariotyczne promotory

Rysunek 1. Podjednostka σ prokariotycznej polimerazy RNA rozpoznaje sekwencje konsensusowe znajdujące się w regionie promotora przed miejscem startu transkrypcji. Podjednostka σ odłącza się od polimerazy po zainicjowaniu transkrypcji.
Promotor jest sekwencją DNA, na której maszyneria transkrypcyjna wiąże się i inicjuje transkrypcję. W większości przypadków, promotory istnieją przed genami, które regulują. Specyficzna sekwencja promotora jest bardzo ważna, ponieważ decyduje o tym, czy odpowiadający mu gen jest transkrybowany przez cały czas, przez pewien czas, czy też rzadko. Chociaż promotory różnią się między genomami prokariotycznymi, kilka elementów jest konserwowanych. W regionach -10 i -35 przed miejscem inicjacji znajdują się dwie sekwencje konsensusu promotora, czyli regiony, które są podobne we wszystkich promotorach i u różnych gatunków bakterii (Rycina 1).
Sekwencja konsensusu -10, zwana regionem -10, to TATAAT. Sekwencja -35, TTGACA, jest rozpoznawana i wiązana przez σ. Gdy ta interakcja jest wykonana, podjednostki enzymu rdzeniowego wiążą się do miejsca. Bogaty w A-T region -10 ułatwia odwijanie szablonu DNA, w wyniku czego powstaje kilka wiązań fosfodiestrowych. Faza inicjacji transkrypcji kończy się produkcją przerwanych transkryptów, które są polimerami około 10 nukleotydów, które są tworzone i uwalniane.
Elongation and Termination in Prokaryotes
Faza elongacji transkrypcji rozpoczyna się od uwolnienia podjednostki σ z polimerazy. Dysocjacja σ pozwala enzymowi rdzeniowemu postępować wzdłuż szablonu DNA, syntetyzując mRNA w kierunku 5′ do 3′ z szybkością około 40 nukleotydów na sekundę. W trakcie elongacji, DNA jest stale odwijane przed enzymem rdzeniowym i ponownie za nim (Rysunek 2). Parowanie zasad między DNA i RNA nie jest wystarczająco stabilne, aby utrzymać stabilność składników syntezy mRNA. Zamiast tego polimeraza RNA działa jako stabilny łącznik między szablonem DNA a powstającymi nićmi RNA, aby zapewnić, że elongacja nie zostanie przedwcześnie przerwana.

Rysunek 2. Kliknij, aby powiększyć obraz. Podczas elongacji prokariotyczna polimeraza RNA podąża wzdłuż szablonu DNA, syntetyzuje mRNA w kierunku od 5′ do 3′ oraz odwija i przewija DNA w miarę odczytu.
Prokariotyczne sygnały zakończenia
Po transkrypcji genu prokariotyczna polimeraza musi zostać poinstruowana, aby oddzielić się od szablonu DNA i uwolnić nowo wytworzony mRNA. W zależności od transkrybowanego genu, istnieją dwa rodzaje sygnałów zakończenia. Jeden z nich jest oparty na białkach, a drugi na RNA. Terminacja zależna od Rho jest kontrolowana przez białko rho, które podąża za polimerazą na rosnącym łańcuchu mRNA. W pobliżu końca genu polimeraza natrafia na ciąg nukleotydów G na szablonie DNA i zatrzymuje się. W wyniku tego białko rho zderza się z polimerazą. Interakcja z rho uwalnia mRNA z pęcherzyka transkrypcyjnego.
Rho-niezależna terminacja jest kontrolowana przez specyficzne sekwencje w nici szablonu DNA. Gdy polimeraza zbliża się do końca transkrybowanego genu, napotyka region bogaty w nukleotydy C-G. MRNA zagina się na siebie, a komplementarne nukleotydy C-G wiążą się ze sobą. W rezultacie powstaje stabilna spinka do włosów, która powoduje zatrzymanie polimerazy w momencie, gdy zaczyna ona transkrybować region bogaty w nukleotydy A-T. Komplementarny region U-A transkryptu mRNA tworzy jedynie słabe oddziaływanie z szablonowym DNA. To, w połączeniu z zatrzymaniem polimerazy, wywołuje wystarczającą niestabilność dla enzymu rdzeniowego, aby oderwać się i uwolnić nowy transkrypt mRNA.
Po zakończeniu, proces transkrypcji jest kompletny. Do czasu zakończenia występuje, transkrypt prokariotyczny byłby już używany do rozpoczęcia syntezy licznych kopii zakodowanego białka, ponieważ procesy te mogą zachodzić jednocześnie. Ujednolicenie transkrypcji, translacji, a nawet degradacji mRNA jest możliwe, ponieważ wszystkie te procesy zachodzą w tym samym kierunku 5′ do 3′, a także dlatego, że w komórce prokariotycznej nie ma przedziałów błonowych (Rysunek 3). W przeciwieństwie do tego, obecność jądra w komórkach eukariotycznych wyklucza jednoczesną transkrypcję i translację.

Rysunek 3. Wiele polimeraz może transkrybować pojedynczy gen bakteryjny, podczas gdy liczne rybosomy jednocześnie przekształcają transkrypty mRNA w polipeptydy. W ten sposób specyficzne białko może szybko osiągnąć wysokie stężenie w komórce bakteryjnej.
Odwiedź animację BioStudio, aby zobaczyć proces transkrypcji u prokariotów.
Pytania praktyczne
Która podjednostka polimerazy E. coli nadaje specyficzność transkrypcji?
- α
- β
- β′
- σ
Regiony -10 i -35 promotorów prokariotycznych są nazywane sekwencjami konsensusowymi, ponieważ ________.
- są identyczne we wszystkich gatunkach bakterii
- są podobne we wszystkich gatunkach bakterii
- istnieją we wszystkich organizmach
- pełnią tę samą funkcję we wszystkich organizmach
Prokariotyczna translacja
Translacja jest podobna u prokariotów i eukariotów. Tutaj zbadamy, jak translacja zachodzi u E. coli, reprezentatywnego prokariota, i określimy wszelkie różnice między translacją bakteryjną i eukariotyczną.
Inicjacja
Inicjacja syntezy białka rozpoczyna się od utworzenia kompleksu inicjacyjnego. W E. coli kompleks ten obejmuje mały rybosom 30S, szablon mRNA, trzy czynniki inicjujące, które pomagają rybosomowi prawidłowo się złożyć, trifosforan guanozyny (GTP), który działa jako źródło energii, oraz specjalny inicjujący tRNA niosący N-formylo-metioninę (fMet-tRNAfMet) (Rysunek 4). Inicjatorowy tRNA oddziałuje z kodonem startu AUG mRNA i niesie ze sobą formylowaną metioninę (fMet). Ze względu na swój udział w inicjacji, fMet jest wstawiany na początku (N terminus) każdego łańcucha polipeptydowego syntetyzowanego przez E. coli. W mRNA E. coli sekwencja prowadząca, znajdująca się przed pierwszym kodonem AUG, zwana sekwencją Shine-Dalgarno (znana również jako rybosomalne miejsce wiążące AGGAGG), oddziałuje poprzez komplementarne parowanie zasad z cząsteczkami rRNA tworzącymi rybosom. Interakcja ta zakotwicza podjednostkę rybosomalną 30S w odpowiednim miejscu na matrycy mRNA. W tym momencie podjednostka rybosomalna 50S łączy się z kompleksem inicjacyjnym, tworząc nienaruszony rybosom.
U eukariotów tworzenie kompleksu inicjacyjnego przebiega podobnie, z następującymi różnicami:
- TRNA inicjatora jest innym wyspecjalizowanym tRNA niosącym metioninę, zwanym Met-tRNAi
- Zamiast wiązać się z mRNA przy sekwencji Shine-Dalgarno, eukariotyczny kompleks inicjacyjny rozpoznaje czapeczkę 5′ eukariotycznego mRNA, a następnie podąża wzdłuż mRNA w kierunku od 5′ do 3′, aż do rozpoznania kodonu startowego AUG. W tym momencie podjednostka 60S wiąże się z kompleksem Met-tRNAi, mRNA i podjednostką 40S.

Rysunek 4. Translacja u bakterii rozpoczyna się od utworzenia kompleksu inicjacyjnego, w skład którego wchodzi mała podjednostka rybosomalna, mRNA, inicjatorowy tRNA niosący N-formylo-metioninę oraz czynniki inicjacyjne. Następnie wiąże się podjednostka 50S, tworząc nienaruszony rybosom.
Elongacja
W prokariotach i eukariotach podstawy wydłużania translacji są takie same. U E. coli wiązanie podjednostki rybosomalnej 50S w celu wytworzenia nienaruszonego rybosomu tworzy trzy funkcjonalnie ważne miejsca rybosomalne: Miejsce A (aminoacylowe) wiąże przychodzące naładowane aminoacylowe tRNA. Miejsce P (peptydylowe) wiąże naładowane tRNA niosące aminokwasy, które utworzyły wiązania peptydowe z rosnącym łańcuchem polipeptydowym, ale nie odłączyły się jeszcze od odpowiadającego im tRNA. Miejsce E (exit) uwalnia zdysocjowane tRNA, tak że mogą one zostać ponownie naładowane wolnymi aminokwasami. Istnieje jeden godny uwagi wyjątek od tej linii montażowej tRNA: Podczas tworzenia kompleksu inicjacyjnego, bakteryjny fMet-tRNAfMet lub eukariotyczny Met-tRNAi wchodzi bezpośrednio do miejsca P bez uprzedniego wejścia do miejsca A, zapewniając wolne miejsce A gotowe do przyjęcia tRNA odpowiadającego pierwszemu kodonowi po AUG.
Elongacja przebiega z jednokodonowymi ruchami rybosomu, z których każdy nazywany jest zdarzeniem translokacji. Podczas każdego zdarzenia translokacji, naładowane tRNA wchodzi w miejscu A, następnie przesuwa się do miejsca P, a następnie ostatecznie do miejsca E w celu usunięcia. Ruchy rybosomalne, czyli kroki, są wywoływane przez zmiany konformacyjne, które przesuwają rybosom o trzy zasady w kierunku 3′. Wiązania peptydowe tworzą się między grupą aminową aminokwasu przyłączonego do tRNA w miejscu A a grupą karboksylową aminokwasu przyłączonego do tRNA w miejscu P. Tworzenie każdego wiązania peptydowego jest katalizowane przez transferazę peptydylową, rybozym oparty na RNA, który jest wbudowany w podjednostkę 50S rybosomalu. Aminokwas związany z tRNA w miejscu P jest również przyłączany do rosnącego łańcucha polipeptydowego. Gdy rybosom przechodzi przez mRNA, dawny tRNA z miejsca P wchodzi do miejsca E, odłącza się od aminokwasu i zostaje wydalony. Kilka etapów podczas elongacji, w tym wiązanie naładowanego aminoacylo tRNA do miejsca A i translokacja, wymaga energii pochodzącej z hydrolizy GTP, która jest katalizowana przez specyficzne czynniki elongacyjne. Co zadziwiające, aparat translacyjny E. coli potrzebuje tylko 0,05 sekundy na dodanie każdego aminokwasu, co oznacza, że białko o masie 200 aminokwasów może zostać przetłumaczone w ciągu zaledwie 10 sekund.
Terminacja
Terminacja translacji następuje, gdy napotkany zostanie kodon nonsensowny (UAA, UAG lub UGA), dla którego nie ma komplementarnego tRNA. Na wyrównanie z miejscem A, te nonsensowne kodony są rozpoznawane przez czynniki uwalniania w prokariota i eukariota, które powodują w miejscu P aminokwas odłącza się od jego tRNA, uwalniając nowo wykonane polipeptyd. Małe i duże podjednostki rybosomalne odłączają się od mRNA i od siebie nawzajem; są niemal natychmiast rekrutowane do innego kompleksu inicjującego translację.
Podsumowując, istnieje kilka kluczowych cech, które odróżniają ekspresję genów u prokariotów od tej obserwowanej u eukariotów. Są one zilustrowane na Rysunku 5 i wymienione w Tabeli 1.
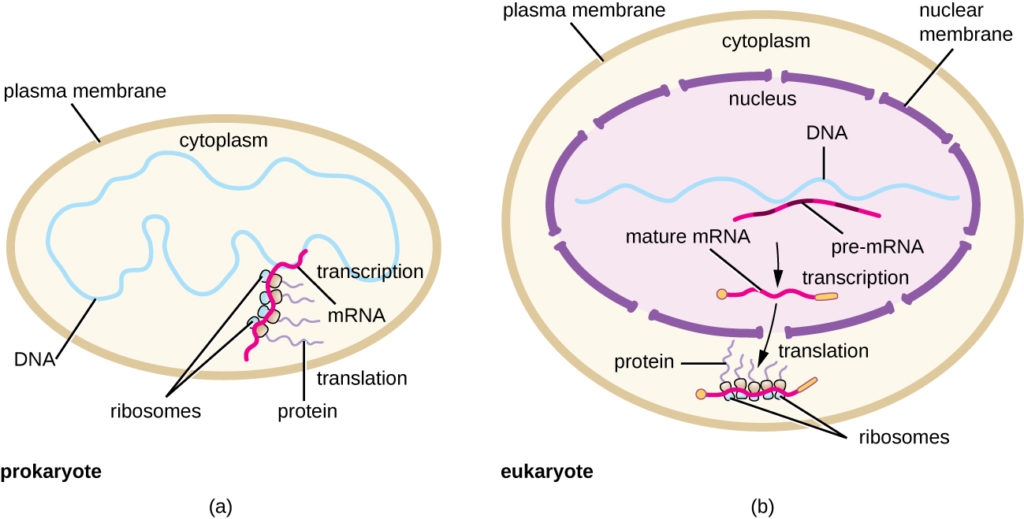
Rys. 5. (a) U prokariotów procesy transkrypcji i translacji zachodzą jednocześnie w cytoplazmie, co pozwala na szybką odpowiedź komórki na bodziec środowiskowy. (b) U eukariotów transkrypcja jest zlokalizowana w jądrze, a translacja w cytoplazmie, co oddziela te procesy i wymaga przetwarzania RNA w celu zapewnienia stabilności.
| Tabela 1. Comparison of Translation in Bacteria Versus Eukaryotes | ||
|---|---|---|
| Property | Bacteria | Eukaryotes |
| Ribosomy | 70S
|
80S
|
| Aminokwas przenoszony przez inicjator tRNA | fMet | Met |
| Sekwencja Shine-.Sekwencja Dalgarno w mRNA | obecna | nieobecna |
| Równoczesna transkrypcja i translacja | Tak | Nie |
Check Your Understanding
Odpowiedz na poniższe pytanie(a), aby sprawdzić, jak dobrze rozumiesz tematy poruszone w poprzedniej sekcji. Ten krótki quiz nie wlicza się do oceny z zajęć i można go powtórzyć nieograniczoną liczbę razy.
Użyj tego quizu, aby sprawdzić swoje zrozumienie i zdecydować, czy (1) dalej studiować poprzednią sekcję, czy (2) przejść do następnej sekcji.
.