Outline the process of prokaryotic transcription and translation
The prokaryotes, which include bacteria and archaea, are mostly single-cell organisms that, by definition, lack membrane-bound nuclei and other organelles. Um cromossomo bacteriano é um círculo covalentemente fechado que, ao contrário dos cromossomos eucariotas, não está organizado em torno de proteínas histônicas. A região central da célula na qual reside o DNA procariótico é chamada nucleoide. Além disso, os procariotas têm frequentemente plasmídeos abundantes, que são moléculas circulares de DNA mais curtas que podem conter apenas um ou alguns genes. Os plasmídeos podem ser transferidos independentemente do cromossoma bacteriano durante a divisão celular e, muitas vezes, carregam traços como a resistência aos antibióticos. Devido a estas características únicas, a transcrição e regulação gênica é um pouco diferente entre as células procarióticas e as eucarióticas.
Objectivos de aprendizagem
- Entender os passos básicos na transcrição do ADN para RNA em células procarióticas
- Entender os princípios básicos da tradução procariótica e como difere da tradução eucariótica
Transcrição procariótica
Inscrição da Transcrição em procariotas
Prokariotas não têm membrana…núcleos fechados. Portanto, os processos de transcrição, tradução e degradação do mRNA podem ocorrer simultaneamente. O nível intracelular de uma proteína bacteriana pode ser rapidamente amplificado por múltiplos eventos de transcrição e tradução que ocorrem simultaneamente no mesmo modelo de DNA. A transcrição procariótica freqüentemente cobre mais de um gene e produz mRNAs policistrônicos que especificam mais de uma proteína.
Nossa discussão aqui exemplificará a transcrição descrevendo este processo na Escherichia coli, uma espécie bacteriana bem estudada. Embora existam algumas diferenças entre transcrição em E. coli e transcrição em arcaea, um entendimento da transcrição de E. coli pode ser aplicado a praticamente todas as espécies bacterianas.
Prokaryotic RNA Polimerase
Prokaryotes usam a mesma RNA polimerase para transcrever todos os seus genes. Na E. coli, a polimerase é composta por cinco subunidades de polipeptídeos, duas das quais são idênticas. Quatro dessas subunidades, denominadas α, α, β, e β′, compreendem a enzima núcleo da polimerase. Estas subunidades se reúnem cada vez que um gene é transcrito, e se desmontam uma vez que a transcrição está completa. Cada subunidade tem um papel único; as duas subunidades α são necessárias para montar a polimerase no DNA; a subunidade β liga-se ao trifosfato ribonucleósido que se tornará parte da nascente molécula mRNA “recém-nascida”; e a β′ liga-se à cadeia de modelos de DNA. A quinta subunidade, σ, está envolvida apenas na iniciação da transcrição. Ela confere uma especificidade transcripcional tal que a polimerase começa a sintetizar o mRNA a partir de um local de iniciação apropriado. Sem σ, a enzima central transcreveria a partir de locais aleatórios e produziria moléculas de mRNA que especificavam a algaraviada proteína. A polimerase composta de todas as cinco subunidades é chamada holoenzima (uma holoenzima é um composto bioquimicamente ativo composto de uma enzima e sua coenzima).
Prokaryotic Promoters
 Figure 1. A subunidade σ de RNA polimerase procariótica reconhece as sequências de consenso encontradas na região promotora a montante do ponto de início da transcrição. A subunidade σ dissocia-se da polimerase após a transcrição ter sido iniciada.
Figure 1. A subunidade σ de RNA polimerase procariótica reconhece as sequências de consenso encontradas na região promotora a montante do ponto de início da transcrição. A subunidade σ dissocia-se da polimerase após a transcrição ter sido iniciada. Um promotor é uma sequência de ADN na qual o mecanismo de transcrição se liga e inicia a transcrição. Na maioria dos casos, os promotores existem a montante dos genes que eles regulam. A seqüência específica de um promotor é muito importante porque determina se o gene correspondente é transcrito o tempo todo, em parte do tempo, ou raramente. Embora os promotores variem entre os genomas procarióticos, alguns poucos elementos são conservados. Nas regiões -10 e -35 a montante do local de iniciação, existem duas sequências de consenso de promotores, ou regiões que são semelhantes entre todos os promotores e entre várias espécies bacterianas (Figura 1).
A sequência de consenso -10, chamada de região -10, é TATAAT. A sequência -35, TTGACA, é reconhecida e vinculada por σ. Uma vez que esta interação é feita, as subunidades da enzima núcleo se ligam ao site. A região -10 rica em A-T facilita o desenrolamento do modelo de DNA, e são feitas várias ligações de fosfodiester. A fase de iniciação da transcrição termina com a produção de transcrições abortivas, que são polímeros de aproximadamente 10 nucleotídeos que são feitos e lançados.
Elongamento e Terminação em Prokaryotes
A fase de alongamento da transcrição começa com a liberação da subunidade σ da polimerase. A dissociação do σ permite que a enzima núcleo prossiga ao longo do modelo de DNA, sintetizando mRNA na direção 5′ a 3′ a uma taxa de aproximadamente 40 nucleotídeos por segundo. À medida que o alongamento prossegue, o DNA é continuamente desenrolado à frente da enzima do núcleo e rebobinado atrás dela (Figura 2). O emparelhamento básico entre DNA e RNA não é suficientemente estável para manter a estabilidade dos componentes de síntese do mRNA. Em vez disso, o RNA polimerase age como um elo estável entre o modelo de DNA e as cadeias nascentes de RNA para assegurar que o alongamento não seja interrompido prematuramente.

Figure 2. Clique para uma imagem maior. Durante o alongamento, a polimerase do RNA procariótica segue o modelo de DNA, sintetiza o mRNA na direção 5′ para 3′, e desenrola e rebobina o DNA conforme é lido.
Sinais de terminação procariótica
Após um gene ser transcrito, a polimerase procariótica precisa ser instruída para se dissociar do modelo de DNA e liberar o mRNA recém feito. Dependendo do gene a ser transcrito, existem dois tipos de sinais de terminação. Um é baseado em proteínas e o outro é baseado em RNA. A terminação dependente de rho é controlada pela proteína rho, que se localiza atrás da polimerase na cadeia crescente do mRNA. Perto do fim do gene, a polimerase encontra uma série de nucleotídeos G no modelo de ADN e estagia. Como resultado, a proteína rho colide com a polimerase. A interação com o rho libera o mRNA da bolha de transcrição.
Rho-independente é controlada por seqüências específicas na cadeia de modelos de DNA. À medida que a polimerase se aproxima do fim do gene transcrito, ela encontra uma região rica em nucleotídeos C-G. O mRNA se dobra sobre si mesmo, e os nucleotídeos C-G complementares se unem. O resultado é um grampo estável que faz com que a polimerase pare assim que ela começa a transcrever uma região rica em nucleotídeos A-T. A região complementar U-A da transcrição do mRNA forma apenas uma fraca interação com o DNA modelo. Isto, juntamente com a polimerase estagnada, induz instabilidade suficiente para que a enzima núcleo se quebre e liberte a nova transcrição do mRNA.
Terminação do mRNA, o processo de transcrição é completo. Quando a terminação ocorre, a transcrição procariótica já teria sido usada para iniciar a síntese de numerosas cópias da proteína codificada, pois estes processos podem ocorrer concomitantemente. A unificação da transcrição, tradução e até a degradação do mRNA é possível porque todos estes processos ocorrem na mesma direção 5′ a 3′, e porque não há compartimentação membranosa na célula procariótica (Figura 3). Em contraste, a presença de um núcleo em células eucarióticas impede a transcrição e tradução simultânea.

Figure 3. Múltiplas polimerases podem transcrever um único gene bacteriano enquanto numerosos ribossomos simultaneamente traduzem as transcrições do mRNA em polipéptidos. Desta forma, uma proteína específica pode rapidamente alcançar uma alta concentração na célula bacteriana.
Visita esta animação BioStudio para ver o processo de transcrição procariótica.
Practice Questions
Which subunit of the E. coli polimerase confere especificidade à transcrição?
- α
- β
- β′
- σ
As -10 e -35 regiões de promotores procarióticos são chamadas seqüências de consenso porque ________.
- eles são idênticos em todas as espécies bacterianas
- eles são semelhantes em todas as espécies bacterianas
- eles existem em todos os organismos
- eles têm a mesma função em todos os organismos
Tradução procariótica
Tradução é semelhante em procariotas e eucariotas. Aqui vamos explorar como a tradução ocorre em E. coli, um procariote representativo, e especificar quaisquer diferenças entre a tradução bacteriana e eucariótica.
Iniciação
O início da síntese de proteínas começa com a formação de um complexo iniciático. Em E. coli, este complexo envolve o pequeno ribossomo 30S, o modelo mRNA, três fatores iniciadores que ajudam a montagem correta do ribossomo, o trifosfato de guanosina (GTP) que atua como fonte de energia, e um iniciador especial tRNA portador de N-formil-metionina (fMet-tRNAfMet) (Figura 4). O tRNA iniciador interage com o códon inicial AUG do mRNA e carrega uma metionina formilada (fMet). Devido ao seu envolvimento na iniciação, fMet é inserido no início (terminal N) de cada cadeia de polipeptídeos sintetizada por E. coli. Em E. coli mRNA, uma sequência líder a montante do primeiro códon AUG, chamada sequência Shine-Dalgarno (também conhecida como o local de ligação do ribossomal AGGAGG), interage através de par de bases complementares com as moléculas de rRNA que compõem o ribossomo. Esta interação ancora a subunidade 30S do ribossomo no local correto no modelo de mRNA. Neste ponto, a subunidade do ribossomo 50S liga-se então ao complexo de iniciação, formando um ribossomo intacto.
Em eucariotas, a formação do complexo de iniciação é semelhante, com as seguintes diferenças:
- O tRNA iniciador é um tRNA especializado diferente carregando metionina, chamado Met-tRNAi
- Em vez de se ligar ao mRNA na sequência Shine-Dalgarno, o complexo de iniciação eucariótica reconhece a tampa 5′ do mRNA eucariótico, depois segue ao longo do mRNA na direcção 5′ para 3′ até que o códão de início AUG seja reconhecido. Neste ponto, a subunidade 60S liga-se ao complexo de Met-tRNAi, mRNA, e à subunidade 40S.

Figure 4. A tradução em bactérias começa com a formação do complexo iniciador, que inclui a pequena subunidade ribossômica, o mRNA, o tRNA iniciador portador de N-formil-metionina, e fatores iniciadores. Então a subunidade 50S liga-se, formando um ribossomo intacto.
Elongamento
Em procariotas e eucariotas, os princípios básicos de alongamento da tradução são os mesmos. Em E. coli, a ligação da subunidade 50S do ribossomo para produzir o ribossomo intacto forma três sítios funcionalmente importantes do ribossomo: O local A (aminoacyl) liga os aminoacyl tRNAs carregados de entrada. O local P (peptidil) liga tRNAs carregados que carregam aminoácidos que formaram ligações de peptídeos com a cadeia crescente de polipeptídeos, mas ainda não se dissociaram do tRNA correspondente deles. O local E (saída) libera tRNAs dissociados de modo que eles podem ser recarregados com aminoácidos livres. Há uma exceção notável para esta linha de montagem de tRNAs: Durante a formação do complexo de iniciação, a bactéria fMet-tRNAfMet ou eukaryotic Met-tRNAi entra diretamente no local P sem primeiro entrar no local A, fornecendo um local A livre pronto para aceitar o tRNA correspondente ao primeiro códon depois do AUG.
Elongamento prossegue com movimentos de um único códon do ribossomo cada um chamado evento de translocação. Durante cada evento de translocação, os tRNAs carregados entram no site A, depois mudam para o site P, e finalmente para o site E para remoção. Os movimentos do ribossoma, ou passos, são induzidos por alterações conformacionais que avançam o ribossoma por três bases na direcção 3′. Formam-se ligações peptídicas entre o grupo aminoácido do aminoácido ligado ao tRNA do local A e o grupo carboxil do aminoácido ligado ao tRNA do local P. A formação de cada ligação de peptídeo é catalisada pela peptidil transferase, uma ribozima à base de RNA que está integrada na subunidade ribossomal 50S. O aminoácido ligado ao tRNA do P-site também está ligado à cadeia crescente de polipeptídeos. À medida que o ribossoma atravessa o mRNA, o antigo tRNA do local P entra no local E, separa-se do aminoácido e é expelido. Várias das etapas durante o alongamento, incluindo a ligação de um aminoácil tRNA carregado ao local A e a translocação, requerem energia derivada da hidrólise de GTP, que é catalisada por fatores de alongamento específicos. Surpreendentemente, o aparelho de tradução de E. coli leva apenas 0,05 segundos para adicionar cada aminoácido, significando que uma proteína de 200 aminoácidos pode ser traduzida em apenas 10 segundos.
Terminação
O fim da tradução ocorre quando um códon sem sentido (UAA, UAG, ou UGA) é encontrado para o qual não há tRNA complementar. Ao se alinhar com o site A, esses códons sem sentido são reconhecidos por fatores de liberação em procariotas e eucariotas que resultam no desprendimento do aminoácido P do seu tRNA, liberando o polipéptido recém feito. As pequenas e grandes subunidades ribossômicas se dissociam do mRNA e umas das outras; elas são recrutadas quase que imediatamente em outro complexo de init iação de tradução.
Em resumo, há várias características-chave que distinguem a expressão gênica procariótica daquela vista nos eucariotas. Estes são ilustrados na Figura 5 e listados na Tabela 1.
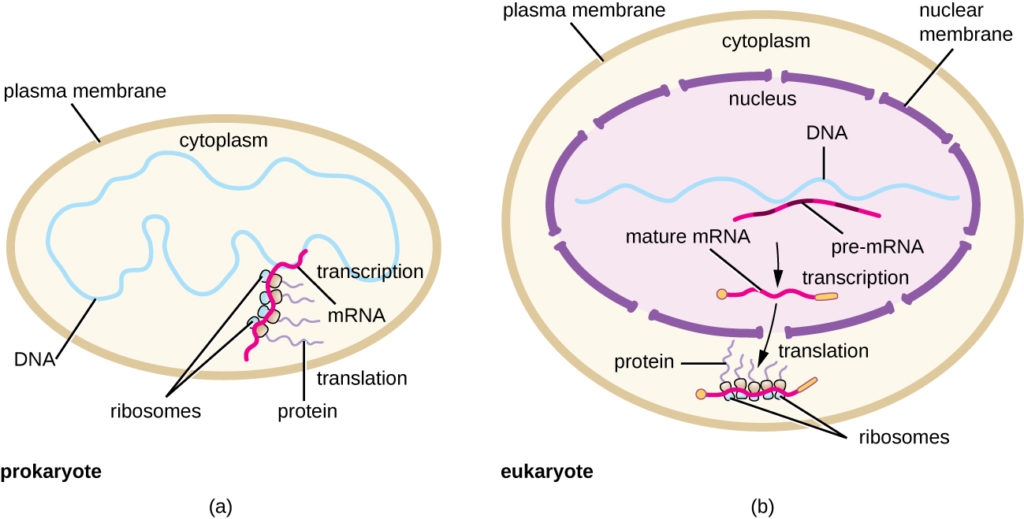
Figure 5. (a) Em procariotas, os processos de transcrição e translação ocorrem simultaneamente no citoplasma, permitindo uma resposta celular rápida a um taco ambiental. (b) Na eucariotas, a transcrição é localizada no núcleo e a translação é localizada no citoplasma, separando estes processos e necessitando do processamento do RNA para estabilidade.
| Tabela 1. Comparação da tradução em Bactérias versus eucariotas | ||
|---|---|---|
| Propriedade | Bactérias | Eukaryotes |
| Ribossomas | 70S
|
80S
|
| Amino ácido transportado pelo iniciador tRNA | fMet | Met |
| Shine-Sequência Dalgarno em mRNA | Atual | Absent |
| Transcrição e tradução simultânea | Sim | Não |
Check Your Understanding
Responda à(s) pergunta(s) abaixo para ver até que ponto entendeu bem os tópicos abordados na secção anterior. Este pequeno questionário não conta para a sua nota na classe, e você pode repeti-lo um número ilimitado de vezes.
Utilize este questionário para verificar a sua compreensão e decidir se (1) estudar mais a seção anterior ou (2) passar para a próxima seção.