Wprowadzenie
Gram-dodatnie bakterie z rodzajów Bacillus i Clostridium mogą różnicować się w celu utworzenia endospory (przetrwalnika), komórki metabolicznie wyciszonej, wytwarzanej w odpowiedzi na warunki, które nie pozwalają na wzrost komórek (McKenney i in., 2012). Po uwolnieniu do środowiska przetrwają w stanie uśpienia przez bardzo długi czas, odporne na szeroki zakres stresów, takich jak wysoka temperatura, odwodnienie, brak składników odżywczych i obecność toksycznych substancji chemicznych (McKenney i in., 2012). Jednakże, spokojne zarodniki są w stanie stale monitorować środowisko i reagować na obecność wody i składników odżywczych poprzez kiełkowanie i tworzenie komórek wegetatywnych, które są w stanie rosnąć i sporulować (McKenney i in., 2012). Odporność na warunki niefizjologiczne wynika po części ze struktur powierzchniowych przetrwalników. U Bacillus subtilis, systemu modelowego dla przetrwalników, powierzchnia zarodników jest zorganizowana w wielowarstwowy płaszcz oraz w skorupę, niedawno odkrytą warstwę otaczającą płaszcz zarodnika (McKenney i in., 2012). Spory B. subtilis są naładowane ujemnie (Huang i in., 2010; Pesce i in., 2014) i mają względną hydrofobowość, która częściowo wynika z glikozylacji niektórych białek powierzchni zarodników (Cangiano i in., 2014; Rusciano i in., 2014). U kilku gatunków Bacillus i Clostridium, w tym B. cereus, B. anthracis, B. megaterium i C. difficile, najbardziej zewnętrzną strukturą zarodnika jest egzosporium, morfologicznie odrębna warstwa złożona z białek i glikoprotein, która otacza płaszcz (Díaz-González i in., 2015; Manetsberger i in., 2015b; Stewart, 2015).
Spory bakteryjne zostały zaproponowane jako platforma do prezentacji heterologicznych białek, z potencjalnymi zastosowaniami, począwszy od rozwoju szczepionek śluzówkowych do biokatalizatorów wielokrotnego użytku, narzędzi diagnostycznych i urządzeń do bioremediacji (Knecht i in., 2011; Isticato i Ricca, 2014; Ricca i in., 2014). Niezwykła i dobrze udokumentowana odporność zarodników (McKenney i in., 2012), podatność kilku gatunków tworzących zarodniki na manipulacje genetyczne (Harwood i Cutting, 1990) oraz historia bezpieczeństwa kilku gatunków (Cutting, 2011; Baccigalupi i in., 2015) przemawiają za wykorzystaniem zarodników jako systemu prezentacji i dostarczania. Opracowano dwie strategie ekspozycji heterologicznych białek na powierzchni zarodników. Strategia rekombinacyjna, oparta na konstrukcji fuzji genów pomiędzy DNA kodującym wybrane białko powierzchni zarodnika i DNA kodującym białko, które ma być eksponowane, była przez lata wykorzystywana do ekspozycji różnych heterologicznych białek (Isticato i Ricca, 2014). Podejście nierekombinacyjne, oparte na spontanicznej adsorpcji pomiędzy oczyszczonymi zarodnikami i oczyszczonymi białkami, zostało również wykorzystane do wyświetlania różnych enzymów i antygenów (Ricca et al., 2014). Szczegóły molekularne kontrolujące adsorpcję zarodników nie zostały w pełni wyjaśnione. Wiadomo, że adsorpcja jest bardziej wydajna, gdy pH buforu wiążącego jest kwaśne (pH 4) (Huang i in., 2010; Sirec i in., 2012) i że kombinacja oddziaływań elektrostatycznych i hydrofobowych jest prawdopodobnie zaangażowana w interakcję (Huang i in., 2010; Sirec i in., 2012). Wiadomo również, że zmutowane zarodniki o silnie zmienionych powierzchniach zarodników oddziałują wydajniej niż izogeniczne zarodniki typu dzikiego z białkami modelowymi (Sirec i in., 2012; Donadio i in., 2016).
W tym miejscu użyliśmy białka fluorescencyjnego, monomerycznej formy białka czerwonej fluorescencji (mRFP) koralowca Discosoma sp. (Campbell i in., 2002), aby ocenić, czy zarodniki B. megaterium są w stanie oddziaływać i adsorbować modelowe heterologiczne białko. B. megaterium obejmuje szereg morfologicznie odrębnych szczepów, których cechą wspólną są nietypowo duże wymiary zarówno komórek (długość do 4 μm i średnica 1,5 μm), jak i zarodników (długość do 3 μm i średnica 1 μm) (Di Luccia i in., 2016). Zarodniki niektórych szczepów B. megaterium otoczone są egzosporium, a ponieważ do tej pory jako platformy adsorpcyjne rozważano jedynie zarodniki gatunków pozbawionych egzosporium, brak jest danych na temat wpływu egzosporium w interakcji z heterologicznymi białkami.
Szczep QM B1551 jest najlepiej scharakteryzowanym szczepem B. megaterium. Szczep ten przenosi około 11% swojego genomu na siedmiu rodzimych plazmidach (Rosso i Vary, 2005; Vary i in., 2007; Eppinger i in., 2011), z których dwa – pBM500 i pBM600 – zostały zidentyfikowane jako niosące geny, które są niezbędne do tworzenia charakterystycznego dla tego szczepu egzosporium w kształcie orzecha włoskiego (Manetsberger i in., 2015a). Skład białkowy egzosporium QM B1551 jest jak dotąd słabo scharakteryzowany, przy czym w wyniku analiz genomicznych zidentyfikowano jedynie kilka genów kodujących ortologi białek egzosporium rozpoznawanych w zarodnikach innych gatunków. Należą do nich geny kodujące białka BclA nap, które tworzą zlokalizowaną drzemkę w przetrwalnikach B. megaterium QM B1551, oraz ortolog białka BxpB, które tworzy podstawową warstwę egzosporium w przetrwalnikach B. cereus. To ostatnie wydaje się pełnić inną rolę w zarodnikach B. megaterium QM B1551, ponieważ szczep mutanta null zachował pozornie normalne egzosporium (Manetsberger i in., 2015a).
W niniejszej pracy przedstawiamy dane, które wykazują, że przetrwalniki B. megaterium QM B1551 mogą skutecznie adsorbować mRFP, i dostarczają dowodów, że cząsteczki białka są w stanie przekroczyć barierę przepuszczalności prezentowaną przez egzosporium, aby zlokalizować się w przestrzeni międzypowłokowej.
Materiały i metody
Szczepy bakteryjne, produkcja zarodników i RFP
Szczepy B. megaterium zastosowane w tym badaniu to QM B1551 i jego pozbawiona plazmidu pochodna PV361 (Rosso i Vary, 2005). Szczep B. subtilis użyty w niniejszych badaniach to PY79 (Youngman i in., 1984). Sporulacja wszystkich szczepów Bacillus była indukowana metodą wyczerpania (Cutting i Vander Horn, 1990). Po 30 h wzrostu w podłożu Difco Sporulation (DS) w temp. 37°C z energicznym wstrząsaniem zbierano zarodniki i oczyszczano je zgodnie z opisem Nicholsona i Setlowa (1990), stosując całonocną inkubację w H2O w temp. 4°C w celu zlizyfikowania resztkowych komórek sporangialnych. Liczbę uzyskanych oczyszczonych zarodników mierzono przez bezpośrednie liczenie za pomocą komory Bürkera pod mikroskopem optycznym (Olympus BH-2 z obiektywem 40×).
Do produkcji mRFP wykorzystano komórki szczepu Escherichia coli RH161 (Donadio i in., 2016), opatrzone wektorem ekspresyjnym pRSET-A niosącym fuzję in-frame 5′ końca regionu kodującego rfp z sześcioma kodonami histydyny pod kontrolą transkrypcyjną promotora T7, hodowano przez 18 h w 37°C w 100 ml pożywki autoindukcyjnej w celu ekspresji heterologicznego białka (Isticato i in., 2010). Białko RFP znakowane His6 było oczyszczane w warunkach natywnych przy użyciu kolumny His-Trap zgodnie z zaleceniami producenta (GE Healthcare Life Science). Oczyszczone białko odsalano przy użyciu kolumny PD10 (GE Healthcare Life Science) w celu usunięcia wysokich stężeń NaCl i imidazolu.
Reakcja adsorpcji
Jeśli nie określono inaczej 5 μg oczyszczonego rekombinowanego mRFP dodawano do zawiesiny zarodników (5 × 108) w 50 mM cytrynianie sodu pH 4,0 w 25°C w końcowej objętości 200 μl. Po 1 h inkubacji mieszaninę wiążącą odwirowywano (10 min przy 13 000 g) w celu frakcjonowania zarodników związanych z mRFP w osadzie od wolnego mRFP w supernatancie.
Analiza Western i Dot-Blot
Osad zarodników z reakcji adsorpcji ponownie zawieszano w 40 μl buforu do ekstrakcji płaszcza zarodników (Nicholson i Setlow, 1990; Giglio i in, 2011), inkubowano w 68°C przez 1 h w celu solubilizacji białek płaszcza zarodników i ładowano na żel 12% SDS-PAGE. Białka były następnie przenoszone elektrolitycznie na filtry nitrocelulozowe (Amersham Pharmacia Biotech) i wykorzystywane do analizy Western blot, jak opisano wcześniej (Isticato i in., 2008) z użyciem monoklonalnego przeciwciała anty-His rozpoznającego mRFP (Sigma). Ilościowe określenie ilości mRFP uzyskano poprzez eksperymenty dot blot porównujące seryjne rozcieńczenia oczyszczonego mRFP i supernatantu testu wiązania. Filtry były następnie wizualizowane metodą ECL-prime (Amersham Pharmacia Biotech) i poddawane analizie densytometrycznej przy użyciu oprogramowania Quantity One 1-D Analysis Software (Bio-Rad). Dot blot i względne analizy densytometryczne wykonywano trzykrotnie w celu weryfikacji istotności wyników.
Mikroskopia fluorescencyjna i immunofluorescencyjna
Zarodniki poadsorpcyjne zawieszano w 50 μl 1x PBS pH 4,0, a 5 μl zawiesiny umieszczano na szkiełkach mikroskopowych i przykrywano szkiełkiem nakrywkowym, uprzednio traktowanym przez 30 s poli-L-lizyną (Sigma). Immunofluorescencję przeprowadzono zgodnie z opisem Isticato i wsp. (2013), z następującymi modyfikacjami: 2,0 × 106 RFP-adsorbowane zarodniki szczepów QM B1551 i PV361 B. megaterium poddano wstępnej obróbce w 1% albuminie surowicy bydlęcej (BSA) – 1x PBS pH 4,0 przez 30 min przed 2 h inkubacją w 4°C z przeciwciałami anty-polihistydynowymi (mysimi; Sigma) rozcieńczonymi 1:20 w 1x PBS pH 4,0-1% BSA. Jako kontrola specyficzności tej techniki, nieadsorbowane zarodniki były bezpośrednio traktowane przeciwciałami anty-His. Po trzech przemyciach, próbki inkubowano z 64-krotnie rozcieńczonym przeciwciałem drugorzędowym anty-mysim sprzężonym z izotiocyjanianem fluoresceiny, FITC (Santa Cruz Biotechnology, Inc.) i przemywano czterokrotnie 1x PBS pH 4,0. Wypłukane próbki zawieszano ponownie w 20 μl 1x PBS pH 4,0, a 10 μl poddawano analizie. Wszystkie próbki obserwowano za pomocą mikroskopu fluorescencyjnego Olympus BX51 wyposażonego w obiektyw olejowy UPlan F1 100×; do wykrywania emisji czerwonej fluorescencji mRFP lub zielonej emisji przeciwciał sprzężonych z FITC stosowano odpowiednio filtry kostkowe U-MNG lub U-MWIBBP. Czasy ekspozycji mieściły się w zakresie od 200 do 3000 ms. Obrazy rejestrowano za pomocą aparatu cyfrowego Olympus DP70 i przetwarzano za pomocą oprogramowania Image Analysis Software (Olympus) w celu drobnych korekt jasności, kontrastu i balansu kolorów (McCloy i in., 2014). ImageJ (v1.48, NIH) został użyty do narysowania konturu wokół 80 zarodników dla każdego szczepu, a minimalne, maksymalne i średnie wartości fluorescencji na piksel zostały zarejestrowane dla każdego zarodnika. Wartości intensywności fluorescencji były następnie wyświetlane jako wykresy pudełkowe z 5-95% przedziałami ufności (McCloy i in., 2014).
Analiza statystyczna
Wyniki z analizy dot blot i mikroskopii fluorescencyjnej są średnimi z trzech niezależnych eksperymentów. Istotność statystyczną określono za pomocą testu t-Studenta, a poziom istotności ustalono na P < 0,05.
Wyniki
mRFP Discosoma sp. jest adsorbowany przez zarodniki B. megaterium Spores
Aby sprawdzić, czy zarodniki B. megaterium QM B1551 były w stanie adsorbować mRFP, 5 μg oczyszczonego białka (Materiały i Metody) inkubowano z 5,0 × 108 oczyszczonymi zarodnikami. Reakcję adsorpcji przeprowadzono w 50 mM cytrynianie sodu o pH 4,0, jak wcześniej opisano (Sirec i in., 2012). Po reakcji zarodniki przemywano dokładnie 1x PBS o pH 4,0, ekstrahowano białka powierzchniowe zarodników zgodnie z opisem w Materiałach i Metodach i analizowano metodą western blotting z przeciwciałem anty-poli-Histydyna-Peroksydaza (Sigma), które rozpoznaje znakowany histydyną N koniec mRFP. Jak pokazano na Figurze 1A, mRFP został wyekstrahowany z zarodników, wskazując, że został zaabsorbowany podczas reakcji, a następnie uwolniony przez obróbkę ekstrakcyjną. Aby ocenić wydajność adsorpcji, przeanalizowaliśmy ilość mRFP pozostawionego bez związku, tj. zarodniki po adsorpcji zostały zebrane przez odwirowanie, a supernatant seryjnie rozcieńczony i przeanalizowany przez dot blotting (Figura 1B). Analiza densytometryczna dot blot (Tabela uzupełniająca 1) wykazała, że gdy 5 μg mRFP zostało użyte w reakcji adsorpcji, mniej niż 1% pozostało niezwiązane, wskazując, że około 99% heterologicznego białka zostało zaadsorbowane do zarodników B. megaterium.

FIGURE 1. Zarodniki Bacillus megaterium QM B1551 adsorbują mRFP. 5 × 108 zarodników inkubowano z 5 μg oczyszczonego mRFP, a następnie próbki poddawano wirowaniu. (A) Białka powierzchniowe zarodników ekstrahowano z frakcji peletu przez traktowanie SDS-DTT, frakcjonowano na SDS-PAGE i analizowano metodą Western blot. Oczyszczone mRFP (20 μg) zostało użyte jako marker. (B) Supernatant, zawierający niezwiązany mRFP, był seryjnie rozcieńczany i analizowany metodą dot blot (QM B1551). Seryjne rozcieńczenia oczyszczonego mRFP (mRFP) były używane jako standard. (C) Zarodniki zaadsorbowane z mRFP przemywano dwukrotnie (W1 i W2) buforem PBS o pH 3,0 lub pH 7,0, albo 1M NaCl, 0,1% roztworem Triton X-100. Jako standardy stosowano seryjne rozcieńczenia oczyszczonego mRFP i niezwiązanego mRFP (UN-mRFP). Immunoreakcje we wszystkich panelach przeprowadzono z przeciwciałem anty-His rozpoznającym mRFP skoniugowanym z peroksydazą chrzanową.
Aby przeanalizować, czy zaadsorbowane cząsteczki mRFP były ściśle związane z powierzchnią zarodników, zarodniki po reakcji adsorpcji przemyto dwukrotnie buforem PBS o pH 3,0 lub pH 7,0, lub roztworem 1M NaCl, 0,1% Triton X-100, jak wcześniej opisano (Donadio i in., 2016). Jak pokazano na Figurze 1C i poparto densytometryczną analizą dot blot (Tabela uzupełniająca 2), płukanie w pH 3,0 nie spowodowało żadnego uwolnienia zaadsorbowanego mRFP, podczas gdy płukanie w pH 7,0 lub z 1M NaCl, 0,1% Triton X-100 spowodowało minimalne, poniżej 1%, uwolnienie cząsteczek mRFP. Wyniki przedstawione na Rysunku 1 sugerują zatem, że mRFP był wydajnie adsorbowany i ściśle związany z przetrwalnikami B. megaterium. Aby ocenić, czy zaadsorbowane na przetrwalnikach cząsteczki mRFP zachowały swoje właściwości fluorescencyjne, przeprowadziliśmy analizę mikroskopii fluorescencyjnej. Jak pokazano na Rysunku 2, zarodniki po reakcjiadsorpcji były związane z silnym sygnałem fluorescencji widocznym wokół całej powierzchni zarodników.
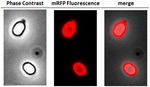
FIGURE 2. Analiza mikroskopii fluorescencyjnej zarodników B. megaterium QM B1551-mRFP. Zarodniki QM B1551 inkubowane z mRFP (5 μg), a następnie przemyte, analizowano za pomocą mikroskopii fluorescencyjnej. To samo pole mikroskopowe obserwowano za pomocą kontrastu fazowego i mikroskopii fluorescencyjnej. Skala 1 μm. Przedstawiono panel merge. Czas ekspozycji wynosił 200 ms.
The Exosporium has an Essential Role in mRFP Adsorption
Strain QM B1551 of B. megaterium contains seven indigenous plasmids (Rosso and Vary, 2005; Eppinger et al., 2011) and plasmid-encoded genes are essential for exosporium formation (Manetsberger et al., 2015a). PV361 jest szczepem utwardzanym QM B1551 pozbawionym wszystkich siedmiu plazmidów i w konsekwencji całkowicie pozbawionym egzosporium (Manetsberger i in., 2015a). Użyliśmy przetrwalników szczepu PV361 do analizy roli egzosporium w adsorpcji mRFP. Równolegle użyliśmy również zarodników B. subtilis PY79, które w poprzednim badaniu okazały się adsorbować mRFP (Donadio i in., 2016). Aby porównać efektywność adsorpcji zarodników szczepów B. subtilis PY79 oraz B. megaterium QM B1551 i PV361, adsorbowaliśmy 5 μg oczyszczonego mRFP z 5,0 × 108 zarodnikami każdego z trzech szczepów. Po reakcjach adsorpcji zarodniki zbierano przez odwirowanie, białka ekstrahowano przez traktowanie SDS-DTT i analizowano metodą western blotting z przeciwciałem anty-His rozpoznającym mRFP. Jak pokazano na Figurze 3A, mRFP był najwyraźniej ekstrahowany w większych ilościach z zarodników QM B1551 niż z zarodników pozostałych dwóch szczepów. W próbie ilościowego określenia tych pozornych różnic, niezwiązany mRFP z reakcji adsorpcyjnych był seryjnie rozcieńczany i analizowany metodą dot blottingu (Figura 3B). Analiza densytometryczna dot blot z Figury 3B (Tabela uzupełniająca 3) wskazała, że zarodniki PY79 i PV361 adsorbowały około 90% całkowitego mRFP, podczas gdy zarodniki QM B1551 adsorbowały prawie całe (ponad 99%) oczyszczone mRFP.
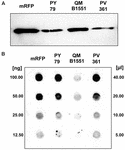
FIGURA 3. Adsorpcja monomerycznego białka czerwonej fluorescencji na przetrwalnikach B. megaterium QM B1551 i PV361 oraz B. subtilis PY79. 5 × 108 zarodników każdego szczepu inkubowano z 5 μg oczyszczonego mRFP, a następnie próbki poddawano wirowaniu. Zarodniki we frakcjach peletowych wykorzystywano do ekstrakcji białek powierzchniowych, które następnie analizowano metodą western blot (A), podczas gdy supernatanty były seryjnie rozcieńczane i analizowane metodą dot blot (B). Seryjne rozcieńczenia oczyszczonego mRFP były używane jako standardy. Reakcje immunologiczne w obu panelach przeprowadzono z przeciwciałem anty-His skoniugowanym z peroksydazą chrzanową.
Wydajność adsorpcji zarodników trzech szczepów analizowano również za pomocą mikroskopii fluorescencyjnej (Figura 4A i Supplementary Figure S1). Obrazy mikroskopowe analizowano za pomocą oprogramowania ImageJ (v1.48, NIH) w celu przeprowadzenia ilościowej analizy obrazu fluorescencyjnego, a fluorescencję zarodników obliczono zgodnie z opisem w Materiałach i Metodach. Analiza 80 zarodników każdego szczepu wykazała średnią wartość fluorescencji na piksel wynoszącą 52,43 (± 5,94), 20,81 (± 2,71) i 32,33 (± 2.97; jednostki arbitralne) dla QM B1551, PV361, i PY79, odpowiednio (Rysunek 4B), dając dalsze dowody, że zarodniki QM B1551 adsorbowały więcej mRFP niż zarodniki pozostałych dwóch szczepów.
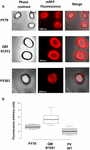
FIGURE 4. Efektywność adsorpcji mRFP na przetrwalnikach B. megaterium QM B1551 i PV361 oraz B. subtilis PY79. (A) Obrazy mikroskopii fluorescencyjnej zarodników PY79, QM B1551 i PV361 po adsorpcji mRFP i przemyciu. Podane są czasy ekspozycji. Pokazano nakładki kontrastu fazowego i czerwonej fluorescencji (panel łączący). Paski skali 1 μm. (B) Wykresy skrzynkowe przedstawiające intensywność fluorescencji osiemdziesięciu różnych zarodników każdego szczepu. Granice każdego pola reprezentują pierwszy i trzeci kwartyl (25 i 75%), a wartości poza polami reprezentują wartości maksymalne i minimalne. Linia dzieląca pole wskazuje wartość mediany dla każdego szczepu. Wartość P jest mniejsza niż 0,0001.
W oparciu o wyniki rysunków 3 i 4, stwierdzamy, że egzosporium, obecne w QM B1551 i nieobecne w PV361 ma istotną rolę w adsorpcji mRFP.
W dodatku, wyniki Figury 4 wskazały, że zarodniki B. subtilis PY79 są bardziej wydajne niż zarodniki B. megaterium PV361 w adsorpcji mRFP, podczas gdy dot blotting zgłoszony na Figurze 3B wskazał podobne wydajności adsorpcji dla tych dwóch szczepów. Uważamy, że ta rozbieżność jest spowodowana silnym zmniejszeniem fluorescencji, gdy mRFP jest związany z PV361, ale nie z zarodnikami PY79 lub QM B1551 (patrz poniżej).
Jakościowa ocena adsorpcji mRFP do zarodników QM B1551
Doświadczenia spot blot (Figura 3B) wskazały, że gdy 5 μg oczyszczonego mRFP zostało użyte w reakcjach adsorpcji z 5,0 × 108 zarodnikami szczepu QM B1551, prawie całe heterologiczne białko zostało związane do zarodnika. W celu określenia maksymalnej ilości mRFP, która może być adsorbowana na zarodnikach QM B1551, powtórzyliśmy reakcje ze wzrastającymi stężeniami oczyszczonego mRFP, tj. 5.0 × 108 zarodników QM B1551 reagowało z 5, 10, 20, 40, 80 i 160 μg oczyszczonego mRFP. Po reakcji zarodniki zbierano przez odwirowanie, a supernatanty zawierające niezwiązany mRFP były seryjnie rozcieńczane i analizowane metodą dot blottingu (Rysunek 5A). Rysunek 5B przedstawia wyniki densytometrycznej analizy dot blot (??), która wskazuje, że gdy 5-80 μg mRFP reagowało z 5 × 108 zarodnikami, procent białka związanego z zarodnikami wynosił ponad 90%. Spadek ilości związanego mRFP zaobserwowano, gdy do reakcji użyto 160 μg oczyszczonego białka. Jednakże, nawet gdy użyto 160 μg oczyszczonego mRFP ponad 60% białka zostało zaadsorbowane, co wskazuje, że 5,0 × 108 zarodników QM B1551 może zaadsorbować około 100 μg mRFP.
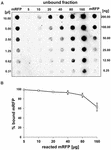
FIGURE 5. Ilościowa ocena adsorpcji mRFP na zarodnikach B. megaterium QM B1551. 5 × 108 zarodników inkubowano z 5, 10, 20, 40, 80 i 160 μg oczyszczonego mRFP. Mieszaniny reakcyjne były następnie poddawane wirowaniu, a supernatanty seryjnie rozcieńczane i analizowane metodą dot blot (A). Jako standardy zastosowano seryjne rozcieńczenia oczyszczonego mRFP. Reakcje immunologiczne w obu panelach przeprowadzono z przeciwciałem anty-His sprzężonym z peroksydazą chrzanową. (B) Procent mRFP zaadsorbowanego na zarodnikach po reakcji z określonymi ilościami endogennego mRFP. Słupki błędów pokazują standardowe błędy średniej z trzech eksperymentów, a wartość P nigdy nie przekracza 0,0025.
mRFP lokalizuje się w przestrzeni międzypłaszczowej w zarodnikach B. megaterium QM B1551
Podejście mikroskopii immunofluorescencyjnej zastosowano do oceny, czy zaadsorbowane cząsteczki mRFP były wyeksponowane na powierzchni zarodników B. megaterium QM B1551. Zarodniki QM B1551 zaadsorbowane różnymi ilościami mRFP reagowały z przeciwciałem monoklonalnym anty-His rozpoznającym rekombinowany mRFP, następnie z fluorescencyjnym przeciwciałem wtórnym anty-mysim (Santa Cruz Biotechnology, Inc.) i były obserwowane pod mikroskopem fluorescencyjnym (Figura 6). Przy najniższej ilości mRFP użytej w tym eksperymencie (2 μg) sygnał fluorescencji mRFP (czerwony) obserwowano dookoła zarodnika, podczas gdy sygnał immunofluorescencji (zielony) był słaby i skoncentrowany głównie na biegunach zarodnika, co sugeruje, że tylko w tych miejscach mRFP był eksponowany na powierzchni zarodnika. Wraz ze wzrostem ilości mRFP użytego w reakcji wzrastała liczba zielonych plamek (5 i 10 μg), a przy najwyższej ilości mRFP użytego w reakcji (20 μg) obserwowano prawie pełny zielony pierścień wokół zarodników. Na podstawie wyników przedstawionych na Rysunku 6 wysunęliśmy hipotezę, że cząsteczki mRFP przenikają przez egzosporium i lokalizują się w przestrzeni międzypłaszczowej pomiędzy płaszczem zewnętrznym a egzosporium, tzn. gdy użyta jest niska ilość mRFP prawie wszystkie cząsteczki białka znajdują się wewnątrz egzosporium i są dostępne dla przeciwciała tylko w kilku miejscach. Zwiększanie ilości mRFP w reakcjach adsorpcji powoduje, że przestrzeń międzypowłokowa „wypełnia się”, aż w końcu więcej cząsteczek mRFP jest dostępnych dla przeciwciała na powierzchni zarodnika. Ta hipoteza sugeruje, że jeśli egzosporium jest pozbawione, to cały mRFP powinien być dostępny dla przeciwciała. Aby to sprawdzić, porównaliśmy pod mikroskopem immunofluorescencyjnym równe liczby zarodników QM B1551 (z egzosporium) i PV361 (bez egzosporium) inkubowanych z taką samą ilością mRFP (5 μg). Gdy egzosporium było obecne (QM B1551) mRFP był tylko częściowo dostępny dla przeciwciała i obserwowano zielone plamy (Rysunek 7 i Supplementary Figure S2). Gdy egzosporium nie było obecne (PV361) zaadsorbowany mRFP był dostępny dla przeciwciała dookoła zarodnika i powstał kompletny zielony pierścień, wspierając hipotezę, że mRFP jest wewnątrz egzosporium w zarodnikach QM B1551.
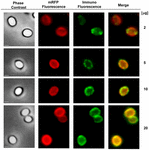
FIGURE 6. Analiza immunofluorescencyjna zaadsorbowanych mRFP zarodników B. megaterium QM B1551. Kwasy 5 × 108 zarodników QM B1551 inkubowano ze zmiennymi stężeniami mRFP, a następnie analizowano za pomocą mikroskopii kontrastu fazowego, fluorescencji i immunofluorescencji, jak opisano w Materiałach i Metodach. To samo pole mikroskopowe dla każdej reakcji jest przedstawione razem z panelem łączącym. Czas ekspozycji wynosił 200 ms dla wszystkich obrazów. Pasek skali, 1 μm.
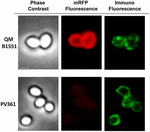
FIGURE 7. Immunofluorescencja mRFP zaadsorbowanego na zarodnikach B. megaterium QM B1551 i PV361. 5 × 108 zarodników QM B1551 i PV361 inkubowano z 5 μg mRFP, a następnie analizowano za pomocą mikroskopii immunofluorescencyjnej, jak opisano w Materiałach i Metodach. Dla każdego pola pokazano kontrast fazowy i mikroskopię immunofluorescencyjną. Czas ekspozycji wynosił 200 ms dla wszystkich obrazów. Pasek skali, 1 μm.
Podczas gdy zarodniki QM B1551 użyte w eksperymentach na Figurze 7 wykazały kompletny czerwony pierścień fluorescencyjny, jak na Figurze 2, zarodniki PV361 wykazały bardzo słaby czerwony sygnał fluorescencyjny. W przypadku przetrwalników PV361 czerwony sygnał był obserwowany tylko przy użyciu długich czasów ekspozycji w mikroskopie fluorescencyjnym (Rysunek 4). Ponieważ mRFP jest obecny wokół przetrwalników PV361 (Figury 3 i 7), wnioskujemy, że fluorescencja mRFP jest osłabiona, gdy białko jest zaadsorbowane do przetrwalników PV361. Dalsze eksperymenty będą potrzebne, aby w pełni zająć się tą kwestią.
Dyskusja
Główne wnioski z tego raportu są takie, że zarodniki B. megaterium są niezwykle wydajne w adsorpcji heterologicznego białka modelowego mRFP, że egzosporium odgrywa ważną rolę w tym procesie, i że cząsteczki mRFP przenikają przez egzosporium lokalizując się między płaszczem zewnętrznym a egzosporium. Wyniki te rozszerzają wcześniejsze prace wykonane na zarodnikach B. subtilis i pokazują, że zarodniki innych gatunków mogą być również wykorzystane do dostarczania heterologicznych białek metodą adsorpcji. Wysoka wydajność adsorpcji zaobserwowana w przypadku przetrwalników B. megaterium jest częściowo spowodowana dużym rozmiarem przetrwalnika w porównaniu z przetrwalnikiem B. subtilis. Rzeczywiście, powierzchnia zarodników B. megaterium jest około 2,5-krotnie większa niż powierzchnia zarodników B. subtilis, z powierzchnią 5,33 μm2 (h: 1,60 ± 0,16 w: 0,84 ± 0,07) vs. 1,98 μm2 (h: 1,07 ± 0,09 w: 0,48 ± 0,03). Duże wymiary pozwalają na adsorpcję do 100 μg mRFP, gdy 160 μg białka wchodzi w reakcję z zarodnikami.
Obserwacja, że mRFP przekracza egzosporium wskazuje, że jest ono przepuszczalne dla mRFP, białka o masie 27 kDa. Przepuszczalno¶ć egzosporium nie jest całkowicie zaskakuj±ca, ponieważ kiełki obecne w ¶rodowisku musz± przekroczyć zewnętrzne warstwy przetrwalnika, aby dotrzeć do swoich receptorów, aczkolwiek kiełki s± zazwyczaj małymi cz±steczkami o masach cz±steczkowych zwykle <200 Da. Dodatkowo, dane dotyczące mRFP są zasadniczo zgodne z wynikami poprzednich badań przeprowadzonych z użyciem znakowanych dekstranów i cząsteczek pokrewnych (Koshikawa i in., 1984; Nishihara i in., 1989). W tych badaniach sugerowano, że egzosporium B. megaterium QM B1551 stanowi barierę przepuszczalności dla cząsteczek o masach cząsteczkowych większych niż 100 KDa, wpływając jednocześnie na przechodzenie cząsteczek o masach pomiędzy 2 a 40 kDa (Koshikawa i in., 1984; Nishihara et al., 1989).
Interesującym wyzwaniem dla przyszłych prac będzie ustalenie mechanizmu lub drogi infiltracji, którą mRFP, a przez wnioskowanie inne heterologiczne białka zainteresowania, podejmuje, aby wejść do przestrzeni międzypowłokowej. Badanie metodą transmisyjnej mikroskopii elektronowej przekrojonych zarodników B. megaterium QM B1551 wskazuje, że egzosporium składa się z dwóch identycznych „powłok” (Manetsberger i in., 2015a), i być może interfejs pomiędzy każdą z tych struktur (we wczesnych pracach opisywany jako „otwory apikalne”) umożliwia wnikanie stosunkowo dużych cząsteczek. Rozszyfrowanie podstawy widocznej utraty fluorescencji mRFP po adsorpcji na zarodnikach PV361, oraz czy cząsteczki mRFP są w stanie przeniknąć do zewnętrznych warstw płaszcza, jak zaobserwowano dla zarodników B. subtilis (Donadio i in., 2016), będzie również interesujące.
W obecnym badaniu, stawiamy hipotezę, że cząsteczki mRFP preferencyjnie przekraczają egzosporium i gromadzą się w przestrzeni międzypłaszczowej pomiędzy płaszczem zewnętrznym a egzosporium. W tym modelu, cząsteczki mRFP są adsorbowane i wyświetlane na powierzchni zarodników tylko wtedy, gdy miejsca adsorpcji (lub pojemność objętościowa?) w przestrzeni międzypowłokowej są wystarczająco zajęte. Sugeruje to, że adsorpcyjne podejście do wyświetlania na powierzchni może być stosowane z zarodnikami B. megaterium QM B1551, chociaż system jest zależny od stosunku zarodników do białka stosowanego w reakcjach adsorpcji. Ponieważ różne szczepy B. megaterium od dawna są wykorzystywane przemysłowo do produkcji enzymów, takich jak amylazy i dehydrogenazy, witamin i cząsteczek przeciwdrobnoustrojowych (Vary i in., 2007), nasze dane sugerują nowe zastosowanie biotechnologiczne dla przetrwalników B. megaterium jako nośnika do wiązania i dostarczania heterologicznych białek.
Wkład autorów
ML i GD – wykonali większość eksperymentów; RA i AS – przyczynili się do interpretacji danych w pracy; GiC, LB i GrC – przyczynili się do sporządzenia pracy i krytycznej jej korekty; ER – przyczynili się do dyskusji i sugestii podczas pracy i przyczynili się do napisania manuskryptu; RI – kierował pracą i przyczynił się do napisania manuskryptu. All authors read and approved the final manuscript.
Funding
This work was supported by a grant (STAR Linea1- 2014) from the Federico II University of Naples and Compagnia di San Paolo, Italy to RI.
Oświadczenie o konflikcie interesów
Autorzy deklarują, że badania zostały przeprowadzone przy braku jakichkolwiek komercyjnych lub finansowych relacji, które mogłyby być interpretowane jako potencjalny konflikt interesów.
Materiały uzupełniające
Materiały uzupełniające do tego artykułu można znaleźć online pod adresem: https://www.frontiersin.org/article/10.3389/fmicb.2016.01752/full#supplementary-material
RYSUNEK S1 | Obrazy całego pola analizy mikroskopii fluorescencyjnej przedstawionej na Rysunku 4. To samo pole mikroskopowe było obserwowane za pomocą kontrastu fazowego i mikroskopii fluorescencyjnej. Skala paska 5 μm. Przedstawiono panel łączący. Czas ekspozycji jest podany.
RYSUNEK S2 | Obrazy całego pola analizy immunofluorescencji przedstawione na Rysunku 7. To samo pole mikroskopowe było obserwowane za pomocą kontrastu fazowego i mikroskopii fluorescencyjnej. Pasek skali 5 μm. Czas ekspozycji wynosił 200 ms dla wszystkich obrazów.
TABELA S1 | Densytometryczna analiza eksperymentów dot blot z supernatantami reakcji adsorpcji z zarodnikami QM B1551 (Figura 1B).
TABLE S2 | Densytometryczna analiza eksperymentów dot blot z supernatantami różnych przemywań po reakcji adsorpcji (Figura 1C).
TABLE S3 | Densytometryczna analiza eksperymentów dot blot z supernatantami reakcji adsorpcji przeprowadzonej z przetrwalnikami szczepów PY79, QM B1551, i PV361 (Figura 3B).
TABLE S4 | Analiza densytometryczna eksperymentów dot blot z supernatantami reakcji adsorpcji przeprowadzonej z różnymi ilościami mRFP.
Baccigalupi, L., Ricca, E., and Ghelardi, E. (2015). „Probiotyki non-LAB: formy przetrwalnikowe”, w Probiotics and Prebiotics: Current Research and Future Trends, eds K. Venema and A. P. do Carmo (Norfolk, VA: Caister Academic Press), 93-103. doi: 10.21775/9781910190098.06
CrossRef Full Text | Google Scholar
Campbell, R. E., Tour, O., Palmer, A. E., Steinbach, P. A., Baird, G. S., Zacharias, D. A., et al. (2002). A monomeric red fluorescent protein. Proc. Natl. Acad. Sci. U.S.A. 99, 7877-7882. doi: 10.1073/pnas.082243699
PubMed Abstract | CrossRef Full Text | Google Scholar
Cangiano, G., Sirec, T., Panarella, C., Isticato, R., Baccigalupi, L., De Felice, M., et al. (2014). The sps gene products affect germination, hydrophobicity and protein adsorption of Bacillus subtilis spores. Appl. Environ. Microbiol. 80, 7293-7302. doi: 10.1128/AEM.02893-14
PubMed Abstract | CrossRef Full Text | Google Scholar
Cutting, S. M. (2011). Probiotyki z rodzaju Bacillus. Food Microbiol. 28, 214-220. doi: 10.1016/j.fm.2010.03.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Cutting, S., and Vander Horn, P. B. (1990). „Genetic analysis,” in Molecular Biological Methods for Bacillus, eds C. Harwood and S. Cutting (Chichester: John Wiley and Sons), 27-74.
Google Scholar
Díaz-González, F., Milano, M., Olguin-Araneda, V., Pizarro-Cerda, J., Castro-Córdova, P., Tzeng, S. C., et al. (2015). Protein composition of the outermost exosporium-like layer of Clostridium difficile 630 spores. J. Proteom. 123, 1-13. doi: 10.1016/j.jprot.2015.03.035
PubMed Abstract | CrossRef Full Text | Google Scholar
Di Luccia, B., D’Apuzzo, E., Varriale, F., Baccigalupi, L., Ricca, E., and Pollice, A. (2016). Bacillus megaterium SF185 indukuje szlaki stresowe i wpływa na rozkład cyklu komórkowego ludzkich komórek nabłonka jelitowego. Benef. Microb. 7, 609-620. doi: 10.3920/BM2016.0020
PubMed Abstract | CrossRef Full Text | Google Scholar
Donadio, G., Lanzilli, M., Sirec, T., Ricca, E., and Isticato, R. (2016) Protein adsorption and localization on wild type and mutant spores of Bacillus subtilis. Microb. Cell Fact. 15:153. doi: 10.1186/s12934-016-0551-2
PubMed Abstract | CrossRef Full Text
Eppinger, M., Bunk, B., Johns, M. A., Edirisinghe, J. N., Kutumbaka, K. K., Koenig, S. S., et al. (2011). Genome sequences of the biotechnologically important Bacillus megaterium strains QM B1551 and DSM319. J. Bacteriol. 193, 4199-4213. doi: 10.1128/JB.00449-11
PubMed Abstract | CrossRef Full Text | Google Scholar
Giglio, R., Fani, R., Isticato, R., De Felice, M., Ricca, E., and Baccigalupi, L. (2011). Organization and evolution of the cotG and cotH genes of Bacillus subtilis. J. Bacteriol. 193, 6664-6673. doi: 10.1128/jb.06121-11
PubMed Abstract | CrossRef Full Text | Google Scholar
Harwood, C. R., and Cutting, S. M. (1990). Molecular Biological Methods for Bacillus. Chichester: John Wiley and Sons.
Google Scholar
Huang, J. M., Hong, H. A., Van Tong, H., Hoang, T. H., Brisson, A., and Cutting, S. M. (2010). Mucosal delivery of antigens using adsorption to bacterial spores. Vaccine 28, 1021-1030. doi: 10.1016/j.vaccine.2009.10.127
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., Pelosi, A., De Felice, M., and Ricca, E. (2010). CotE wiąże się z CotC i CotU i pośredniczy w ich interakcji podczas tworzenia płaszcza przetrwalnikowego u Bacillus subtilis. J. Bacteriol. 192, 949-954. doi: 10.1128/JB.01408-09
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., Pelosi, A., Zilhão, R., Baccigalupi, L., Henriques, A. O., De Felice, M., et al. (2008). CotC-CotU heterodimerization during assembly of the Bacillus subtilis spory coat. J. Bacteriol. 190, 1267-1275. doi: 10.1128/JB.01425-07
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., and Ricca, E. (2014). Spore surface display. Microbiol. Spectr. 2, 351-366. doi: 10.1128/microbiolspec.TBS-0011-2012
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., Sirec, T., Treppiccione, L., Maurano, F., De Felice, M., Rossi, M., et al. (2013). Nonrecombinant display of the B subunit of the heat labile toxin of Escherichia coli on wild type and mutant spores of Bacillus subtilis. Microb. Cell Fact. 12:98. doi: 10.1186/1475-2859-12-98
PubMed Abstract | CrossRef Full Text | Google Scholar
Knecht, L. D., Pasini, P., and Daunert, S. (2011). Bacterial spores as platforms for bioanalytical and biomedical applications. Anal. Bioanal. Chem. 400, 977-989. doi: 10.1007/s00216-011-4835-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Koshikawa, T., Beaman, T. C., Pankratz, H. S., Nakashio, S., Corner, T. R., and Gerhardt, P. (1984). Resistance, germination, and permeability correlates of Bacillus megaterium spores successively divested of integument layers. J. Bacteriol. 159, 624-632.
PubMed Abstract | Google Scholar
Manetsberger, J., Hall, E. A. H., and Christie, G. (2015a). Plasmid-encoded genes influence exosporium assembly and morphology in Bacillus megaterium QM B1551 spores. FEMS Microbiol. Lett. 362:fnv147. doi: 10.1093/femsle/fnv147
PubMed Abstract | CrossRef Full Text | Google Scholar
Manetsberger, J., Manton, J. D., Erdelyi, M. J., Lin, H., Rees, D., Christie, G., et al. (2015b). Ellipsoid localization microscopy infers the size and order of protein layers in Bacillus spory coats. Biophys. J. 109, 2058-2066. doi: 10.1016/j.bpj.2015.09.023
PubMed Abstract | CrossRef Full Text | Google Scholar
McCloy, R., Rogers, S., Caldon, E., Lorca, T., Castro, A., and Burgess, A. (2014). Partial inhibition of Cdk1 in G2 phase overrides the SAC and decouples mitotic events. Cell Cycle 13, 1400-1412. doi: 10.4161/cc.28401
PubMed Abstract | CrossRef Full Text | Google Scholar
McKenney, P. T., Driks, A., and Eichemberger, P. (2012). The Bacillus subtilis endospore: assembly and functions of the multilayered coat. Nat. Rev. Microbiol. 11, 33-44. doi: 10.1038/nrmicro2921
PubMed Abstract | CrossRef Full Text | Google Scholar
Nicholson, W. L., and Setlow, P. (1990). „Sporulacja, kiełkowanie i wyrastanie,” in Molecular biological methods for Bacillus, eds C. Harwood and S. Cutting (Chichester: John Wiley and Sons), 391-450.
Google Scholar
Nishihara, T., Takubo, Y., Kawamata, E., Koshikawa, T., Ogaki, J., and Kondo, M. (1989). Role of outer coat in resistance of Bacillus megaterium spory. J. Biochem. 106, 270-273.
PubMed Abstract | Google Scholar
Pesce, G., Rusciano, G., Sirec, T., Isticato, R., Sasso, A., and Ricca, E. (2014). Surface charge and hydrodynamic coefficient measurements of Bacillus subtilis spory by optical tweezers. Colloids Surf. B Biointerfaces 116C, 568-575. doi: 10.1016/j.colsurfb.2014.01.039
PubMed Abstract | CrossRef Full Text | Google Scholar
Ricca, E., Baccigalupi, L., Cangiano, G., De Felice, M., and Isticato, R. (2014). Mucosal vaccine delivery by non-recombinant spores of Bacillus subtilis. Microb. Cell Fact. 13, 115. doi: 10.1186/s12934-014-0115-2
PubMed Abstract | CrossRef Full Text | Google Scholar
Rosso, M. L., and Vary, P. S. (2005). Distribution of Bacillus megaterium QM B1551 plasmids among other B. megaterium strains and Bacillus species. Plasmid 53, 205-217. doi: 10.1016/j.plasmid.2004.10.005
PubMed Abstract | CrossRef Full Text | Google Scholar
Rusciano, G. Zito, G., Isticato, R., Sirec, T., Ricca, E., Bailo, E., and Sasso, A. (2014). Nanoscale chemical imaging of Bacillus subtilis spores by combining tip-enhanced Raman scattering and advanced statistical tools. ACS Nano 8, 12300-12309. doi: 10.1021/nn504595k
PubMed Abstract | CrossRef Full Text | Google Scholar
Sirec, T., Strazzulli, A., Isticato, R., De Felice, M., Moracci, M., and Ricca, E. (2012). Adsorption of beta-galactosidase of Alicyclobacillus acidocaldarius on wild type and mutants spores of Bacillus subtilis. Microb. Cell Fact. 11:100. doi: 10.1186/1475-2859-11-100
PubMed Abstract | CrossRef Full Text | Google Scholar
Stewart, G. C. (2015). Warstwa egzosporium zarodników bakterii: połączenie ze środowiskiem i zainfekowanym gospodarzem. Microbiol. Mol. Biol. Rev. 79, 437-457. doi: 10.1128/MMBR.00050-15
PubMed Abstract | CrossRef Full Text | Google Scholar
Vary, P. S., Biedendieck, R., Fuerch, T., Meinhardt, F., Rohde, M., Deckwer, W. -D., et al. (2007). Bacillus megaterium – od prostej bakterii glebowej do gospodarza przemysłowej produkcji białek. Appl. Microbiol. Biotechnol. 76, 957-967 doi: 10.1007/s00253-007-1089-3
PubMed Abstract | CrossRef Full Text | Google Scholar
Youngman, P., Perkins, J. B., and Losick, R. (1984). A novel method for the rapid cloning in Escherichia coli of Bacillus subtilis chromosomal DNA adjacent to Tn917 insertion. Mol. Gen. Genet. 195, 424-433. doi: 10.1007/BF00341443
CrossRef Full Text | Google Scholar
.