Introducción
Las bacterias grampositivas de los géneros Bacillus y Clostridium pueden diferenciarse para formar una endospora (espora), una célula metabólicamente quiescente producida en respuesta a condiciones que no permiten el crecimiento celular (McKenney et al., 2012). Una vez liberada en el medio ambiente, la espora sobrevive en su estado latente durante periodos extremadamente largos, resistiendo a una amplia gama de tensiones como las altas temperaturas, la deshidratación, la ausencia de nutrientes y la presencia de productos químicos tóxicos (McKenney et al., 2012). Sin embargo, la espora quiescente es capaz de vigilar continuamente el entorno y responder a la presencia de agua y nutrientes germinando y originando una célula vegetativa capaz de crecer y esporular (McKenney et al., 2012). La resistencia a condiciones no fisiológicas se debe, en parte, a las estructuras superficiales de las esporas. En Bacillus subtilis, el sistema modelo para los formadores de esporas, la superficie de la espora está organizada en una capa multicapa y en una costra, una capa recientemente descubierta que rodea la capa de la espora (McKenney et al., 2012). Las esporas de B. subtilis están cargadas negativamente (Huang et al., 2010; Pesce et al., 2014) y tienen una hidrofobicidad relativa que se debe en parte a la glicosilación de algunas proteínas de la superficie de la espora (Cangiano et al., 2014; Rusciano et al., 2014). En varias especies de Bacillus y Clostridium, como B. cereus, B. anthracis, B. megaterium y C. difficile, la estructura más externa de la espora es el exosporio, una capa morfológicamente distinta compuesta por proteínas y glicoproteínas que rodea la cubierta (Díaz-González et al., 2015; Manetsberger et al., 2015b; Stewart, 2015).
La espora bacteriana se ha propuesto como plataforma para desplegar proteínas heterólogas, con aplicaciones potenciales que van desde el desarrollo de vacunas para la mucosa hasta biocatalizadores reutilizables, herramientas de diagnóstico y dispositivos de biorremediación (Knecht et al., 2011; Isticato y Ricca, 2014; Ricca et al., 2014). La notable y bien documentada resistencia de la espora (McKenney et al., 2012), la amenidad de varias especies formadoras de esporas a la manipulación genética (Harwood y Cutting, 1990) y el historial de seguridad de varias especies (Cutting, 2011; Baccigalupi et al., 2015) apoyan el uso de la espora como sistema de visualización y entrega. Se han desarrollado dos estrategias para mostrar proteínas heterólogas en la superficie de la espora. Una estrategia recombinante, basada en la construcción de fusiones de genes entre el ADN que codifica una proteína seleccionada de la superficie de la espora y el ADN que codifica la proteína que se va a mostrar, se ha utilizado a lo largo de los años para mostrar una variedad de proteínas heterólogas (Isticato y Ricca, 2014). También se ha utilizado un enfoque no recombinante, basado en la adsorción espontánea entre esporas purificadas y proteínas purificadas, para mostrar diversas enzimas y antígenos (Ricca et al., 2014). Los detalles moleculares que controlan la adsorción de las esporas no se han dilucidado del todo. Se sabe que la adsorción es más eficiente cuando el pH del tampón de unión es ácido (pH 4) (Huang et al., 2010; Sirec et al., 2012) y que es probable que en la interacción intervenga una combinación de interacciones electrostáticas e hidrofóbicas (Huang et al., 2010; Sirec et al., 2012). También se sabe que las esporas mutantes con las superficies de las esporas severamente alteradas interactúan más eficientemente que las esporas isogénicas de tipo salvaje con las proteínas modelo (Sirec et al., 2012; Donadio et al., 2016).
Aquí, utilizamos una proteína fluorescente, la forma monomérica de la proteína roja fluorescente (mRFP) del coral Discosoma sp. (Campbell et al., 2002), para evaluar si las esporas de B. megaterium son capaces de interactuar y adsorber una proteína heteróloga modelo. B. megaterium comprende una serie de cepas morfológicamente distintas que comparten las inusuales grandes dimensiones tanto de las células (longitud de hasta 4 μm y un diámetro de 1,5 μm) como de las esporas (longitud de hasta 3 μm y diámetro de 1 μm) (Di Luccia et al., 2016). Las esporas de algunas cepas de B. megaterium están rodeadas por un exosporio, y dado que hasta ahora solo se han considerado como plataformas de adsorción las esporas de especies que carecen de exosporio, no se dispone de datos sobre el impacto del exosporio en la interacción con proteínas heterólogas.
La cepa QM B1551 es la cepa mejor caracterizada de B. megaterium. Esta cepa lleva alrededor del 11% de su genoma en siete plásmidos autóctonos (Rosso y Vary, 2005; Vary et al., 2007; Eppinger et al., 2011), dos de los cuales -pBM500 y pBM600- han sido identificados como portadores de genes que son esenciales para la formación del distintivo exosporio «en forma de nuez» de esta cepa (Manetsberger et al., 2015a). La composición proteica del exosporio de QM B1551 está aún poco caracterizada, y solo se han identificado mediante análisis genómicos unos pocos genes que codifican ortólogos de proteínas reconocidas del exosporio en esporas de otras especies. Entre ellos se encuentran los genes que codifican las proteínas de la siesta BclA, que forman una siesta localizada en las esporas de B. megaterium QM B1551, además de un ortólogo de la proteína BxpB que forma la capa basal del exosporio en las esporas de la familia B. cereus. Esta última parece cumplir una función diferente en las esporas de B. megaterium QM B1551, ya que una cepa mutante nula conservó un exosporio aparentemente normal (Manetsberger et al., 2015a).
En este trabajo, presentamos datos que demuestran que las esporas de B. megaterium QM B1551 pueden adsorber eficientemente la mRFP, y aportan pruebas de que las moléculas proteicas son capaces de atravesar la barrera de permeabilidad que presenta el exosporio para localizarse en el espacio intercapa.
Materiales y métodos
Estreñas bacterianas, esporas y producción de RFP
Las cepas de B. megaterium empleadas en este estudio son QM B1551 y su derivado sin plásmido PV361 (Rosso y Vary, 2005). La cepa de B. subtilis utilizada en este estudio fue PY79 (Youngman et al., 1984). La esporulación de todas las cepas de Bacillus se indujo mediante el método de agotamiento (Cutting y Vander Horn, 1990). Después de 30 horas de crecimiento en el medio Difco Sporulation (DS) a 37°C con agitación vigorosa, se recogieron las esporas y se purificaron según lo descrito por Nicholson y Setlow (1990), utilizando la incubación de una noche en H2O a 4°C para lisar las células esporangiales residuales. El número de esporas purificadas obtenidas se midió por recuento directo con una cámara Bürker bajo un microscopio óptico (Olympus BH-2 con objetivo de 40×).
Para la producción de mRFP, se utilizaron células de la cepa RH161 de Escherichia coli (Donadio et al, 2016), portadoras del vector de expresión pRSET-A que lleva una fusión in-frame del extremo 5′ de la región codificante de rfp a seis codones de histidina bajo el control transcripcional de un promotor T7, se cultivaron durante 18 h a 37°C en 100 ml de medio de autoinducción para expresar la proteína heteróloga (Isticato et al., 2010). La proteína RFP marcada con His6 se purificó en condiciones nativas utilizando una columna His-Trap según las recomendaciones del fabricante (GE Healthcare Life Science). La proteína purificada se desaló utilizando una columna PD10 (GE Healthcare Life Science) para eliminar las altas concentraciones de NaCl e imidazol.
Reacción de adsorción
A menos que se especifique lo contrario, se añadieron 5 μg de mRFP recombinante purificada a una suspensión de esporas (5 × 108) en Citrato de Sodio 50 mM pH 4.0 a 25°C en un volumen final de 200 μl. Después de 1 h de incubación, la mezcla de unión se centrifugó (10 min a 13.000 g) para fraccionar las esporas unidas a la mRFP en el pellet de la mRFP libre en el sobrenadante.
Análisis Western y Dot-Blot
Los pellets de esporas de las reacciones de adsorción se resuspendieron en 40 μl de tampón de extracción de la capa de esporas (Nicholson y Setlow, 1990; Giglio et al, 2011), se incubaron a 68°C durante 1 h para solubilizar las proteínas de la cubierta de las esporas y se cargaron en un gel de SDS-PAGE al 12%. A continuación, las proteínas se electrotransfirieron a filtros de nitrocelulosa (Amersham Pharmacia Biotech) y se utilizaron para el análisis de Western blot como se informó anteriormente (Isticato et al., 2008) utilizando un anticuerpo monoclonal anti-His que reconoce la mRFP (Sigma). Se obtuvo una determinación cuantitativa de la cantidad de mRFP mediante experimentos de dot blot comparando diluciones seriadas de mRFP purificada y sobrenadante del ensayo de unión. A continuación, los filtros se visualizaron mediante el método ECL-prime (Amersham Pharmacia Biotech) y se sometieron a un análisis densitométrico con el software de análisis 1-D Quantity One (Bio-Rad). Los análisis de Dot blot y de densitometría relativa se realizaron tres veces para verificar la significación de los resultados.
Microscopía de fluorescencia e inmunofluorescencia
Las esporas post-adsorción se resuspendieron en 50 μl de 1x PBS pH 4,0 y 5 μl de la suspensión se colocaron en portaobjetos de microscopio y se cubrieron con un cubreobjetos previamente tratado durante 30 s con poli-L-lisina (Sigma). La inmunofluorescencia se realizó según lo descrito por Isticato et al. (2013), con las siguientes modificaciones: Se pretrataron 2,0 × 106 esporas adsorbidas por RFP de las cepas QM B1551 y PV361 de B. megaterium con albúmina de suero bovino (BSA) al 1% – 1x PBS pH 4,0 durante 30 min antes de la incubación de 2 h a 4°C con los anticuerpos anti-polihistidina (ratón; Sigma) diluidos 1:20 en 1x PBS pH 4,0-1% BSA. Como control de la especificidad de esta técnica, las esporas no adsorbidas se trataron directamente con anticuerpos anti-His. Después de tres lavados, las muestras se incubaron con un anticuerpo secundario anti-ratón diluido 64 veces y conjugado con isotiocianato de fluoresceína, FITC (Santa Cruz Biotechnology, Inc.) y se lavaron cuatro veces con 1x PBS pH 4,0. Las muestras lavadas se resuspendieron en 20 μl de 1x PBS pH 4,0 y se analizaron 10 μl. Todas las muestras se observaron con un microscopio de fluorescencia Olympus BX51 equipado con un objetivo de aceite UPlan F1 de 100×; se utilizaron filtros de cubo U-MNG o U-MWIBBP para detectar la emisión de fluorescencia roja de la mRFP o la emisión verde de los anticuerpos conjugados con FITC, respectivamente. Los tiempos de exposición oscilaron entre 200 y 3000 ms. Las imágenes se capturaron con una cámara digital Olympus DP70 y se procesaron con el software de análisis de imágenes (Olympus) para realizar pequeños ajustes de brillo, contraste y balance de color (McCloy et al., 2014). Se utilizó ImageJ (v1.48, NIH) para dibujar un contorno alrededor de 80 esporas para cada cepa y se registraron los valores de fluorescencia mínimos, máximos y medios por píxel para cada espora. Los valores de la intensidad de la fluorescencia se mostraron posteriormente como diagramas de caja con intervalos de confianza del 5-95% (McCloy et al., 2014).
Análisis estadístico
Los resultados de los análisis de dot blot y microscopía de fluorescencia son los promedios de tres experimentos independientes. La significación estadística se determinó mediante la prueba t de Student, y el nivel de significación se fijó en P < 0,05.
Resultados
La mRFP de Discosoma sp. es adsorbida por esporas de B. megaterium
Para verificar si las esporas de B. megaterium QM B1551 eran capaces de adsorber la mRFP, se incubaron 5 μg de la proteína purificada (Materiales y Métodos) con 5,0 × 108 esporas purificadas. La reacción de adsorción se realizó en citrato de sodio 50 mM a pH 4,0, como se describió previamente (Sirec et al., 2012). Después de la reacción, las esporas se lavaron extensamente con 1x PBS pH 4,0, se extrajeron las proteínas de la superficie de las esporas como se describe en Materiales y Métodos y se analizaron mediante western blot con el anticuerpo anti-polyHistidine-Peroxidase (Sigma), que reconoce el extremo N marcado con histidina de la mRFP. Como se muestra en la Figura 1A, la mRFP fue extraída de las esporas, lo que indica que fue absorbida durante la reacción y luego liberada por el tratamiento de extracción. Para evaluar la eficiencia de la adsorción, analizamos la cantidad de mRFP que quedaba sin unir, es decir, las esporas post-adsorbidas se recogieron por centrifugación y el sobrenadante se diluyó en serie y se analizó por dot blotting (Figura 1B). Un análisis densitométrico del dot blot (Tabla Suplementaria 1) mostró que cuando se utilizaron 5 μg de mRFP en la reacción de adsorción quedó menos del 1% sin unir, lo que indica que aproximadamente el 99% de la proteína heteróloga se adsorbió a las esporas de B. megaterium.

FIGURA 1. Las esporas de Bacillus megaterium QM B1551 adsorben mRFP. Se incubaron 5 × 108 esporas con 5 μg de mRFP purificada y luego se sometieron las muestras a centrifugación. (A) Las proteínas de la superficie de las esporas se extrajeron de la fracción de pellets mediante el tratamiento SDS-DTT, se fraccionaron en SDS-PAGE y se analizaron mediante Western blot. Se utilizó mRFP purificada (20 μg) como marcador. (B) El sobrenadante, que contiene la mRFP no unida, se diluyó en serie y se analizó mediante dot blot (QM B1551). Se utilizaron diluciones seriadas de mRFP purificada (mRFP) como estándar. (C) Las esporas adsorbidas con mRFP se lavaron dos veces (W1 y W2) con tampón PBS a pH 3,0 o pH 7,0, o con una solución de NaCl 1M, Tritón X-100 al 0,1%. Se utilizaron diluciones seriadas de mRFP purificada y de mRFP no unida (UN-mRFP) como estándares. Las inmunorreacciones en todos los paneles se realizaron con anticuerpos anti-His que reconocen la mRFP conjugados con peroxidasa de rábano picante.
Para analizar si las moléculas de mRFP adsorbidas estaban fuertemente unidas a la superficie de la espora, las esporas de la reacción de post-adsorción se lavaron dos veces con tampón PBS a pH 3,0 o pH 7,0, o con una solución de 1M NaCl, 0,1% Triton X-100 como se describió anteriormente (Donadio et al., 2016). Como se muestra en la Figura 1C, y apoyado por el análisis densitométrico del dot blot (Tabla Suplementaria 2), los lavados a pH 3,0 no causaron ninguna liberación de la mRFP adsorbida, mientras que los lavados a pH 7,0 o con 1M NaCl, 0,1% Triton X-100 causaron una liberación mínima, menos del 1%, de moléculas de mRFP. Por lo tanto, los resultados presentados en la Figura 1 sugieren que la mRFP se adsorbió eficientemente y se unió fuertemente a las esporas de B. megaterium. Para evaluar si las moléculas de mRFP adsorbidas a las esporas conservaban sus propiedades de fluorescencia, realizamos un análisis de microscopía de fluorescencia. Como se muestra en la Figura 2, las esporas de reacción post-adsorción se asociaron con una fuerte señal de fluorescencia visible alrededor de toda la superficie de la espora.
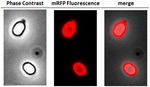
FIGURA 2. Análisis por microscopía de fluorescencia de esporas de B. megaterium QM B1551-mRFP. Las esporas QM B1551 incubadas con mRFP (5 μg), y posteriormente lavadas, se analizaron mediante microscopía de fluorescencia. El mismo campo de microscopía se observó mediante contraste de fase y microscopía de fluorescencia. Barra de escala 1 μm. Se informa del panel de fusión. El tiempo de exposición fue de 200 ms.
El exosporio tiene un papel esencial en la adsorción de mRFP
La cepa QM B1551 de B. megaterium contiene siete plásmidos autóctonos (Rosso y Vary, 2005; Eppinger et al., 2011) y los genes codificados por plásmidos son esenciales para la formación del exosporio (Manetsberger et al., 2015a). PV361 es una cepa curada por QM B1551 que carece de los siete plásmidos y, en consecuencia, carece totalmente del exosporio (Manetsberger et al., 2015a). Utilizamos esporas de la cepa PV361 para analizar el papel del exosporio en la adsorción de mRFP. Paralelamente, también utilizamos esporas de B. subtilis PY79 que en un estudio anterior han demostrado adsorber mRFP (Donadio et al., 2016). Para comparar la eficiencia de adsorción de las esporas de las cepas B. subtilis PY79 y B. megaterium QM B1551 y PV361, adsorbimos 5 μg de mRFP purificada con 5,0 × 108 esporas de cada una de las tres cepas. Después de las reacciones de adsorción, se recogieron las esporas por centrifugación, se extrajeron las proteínas por tratamiento SDS-DTT y se analizaron por western blot con anticuerpos anti-His que reconocen la mRFP. Como se muestra en la Figura 3A, la mRFP se extrajo aparentemente en mayores cantidades de las esporas de QM B1551 que de las esporas de las otras dos cepas. En un intento de cuantificar estas aparentes diferencias, la mRFP no ligada de las reacciones de adsorción se diluyó en serie y se analizó mediante dot blotting (Figura 3B). Un análisis densitométrico del dot blot de la Figura 3B (Tabla Suplementaria 3) indicó que las esporas PY79 y PV361 adsorbieron alrededor del 90% de la mRFP total mientras que las esporas QM B1551 adsorbieron casi toda (más del 99%) la mRFP purificada.
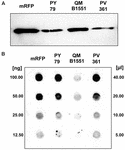
FIGURA 3. Adsorción de la proteína roja fluorescente monomérica a esporas de B. megaterium QM B1551 y PV361 y B. subtilis PY79. Se incubaron 5 × 108 esporas de cada cepa con 5 μg de mRFP purificada y luego se sometieron las muestras a centrifugación. Las esporas de las fracciones del pellet se utilizaron para extraer proteínas de superficie que posteriormente se analizaron por western blot (A), mientras que los sobrenadantes se diluyeron en serie y se analizaron por dot blot (B). Se utilizaron diluciones seriadas de mRFP purificada como estándares. Las inmunorreacciones en ambos paneles se realizaron con un anticuerpo anti-His conjugado con peroxidasa de rábano picante.
La eficiencia de adsorción de las esporas de las tres cepas también se analizó mediante microscopía de fluorescencia (Figura 4A y Figura Suplementaria S1). Las imágenes de microscopía se analizaron con el software ImageJ (v1.48, NIH) para realizar un análisis cuantitativo de las imágenes de fluorescencia y se calculó la fluorescencia de las esporas como se describe en Materiales y Métodos. El análisis de 80 esporas de cada cepa indicó un valor medio de fluorescencia por píxel de 52,43 (± 5,94), 20,81 (± 2,71) y 32,33 (± 2.97; unidades arbitrarias) para QM B1551, PV361, y PY79, respectivamente (Figura 4B), lo que confiere una prueba más de que las esporas de QM B1551 adsorbieron más mRFP que las esporas de las otras dos cepas.
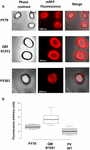
FIGURA 4. Eficiencia de adsorción de la mRFP a las esporas de B. megaterium QM B1551 y PV361 y B. subtilis PY79. (A) Imágenes de microscopía de fluorescencia de esporas de PY79, QM B1551 y PV361 tras la adsorción de mRFP y el lavado. Se indican los tiempos de exposición. Se muestran superposiciones de contraste de fase y fluorescencia roja (panel de fusión). Barras de escala de 1 μm. (B) Gráficos de caja que muestran la intensidad de la fluorescencia de ochenta esporas diferentes de cada cepa. Los límites de cada caja representan el primer y el tercer cuartil (25 y 75%) y los valores fuera de las cajas representan los valores máximos y mínimos. La línea que divide la caja indica el valor medio de cada cepa. El valor P es inferior a 0,0001.
En base a los resultados de las figuras 3 y 4, concluimos que el exosporio, presente en QM B1551 y ausente en PV361 tiene un papel relevante en la adsorción de mRFP.
Además, los resultados de la Figura 4 indicaron que las esporas de B. subtilis PY79 son más eficientes que las esporas de B. megaterium PV361 en la adsorción de mRFP, mientras que el dot blot reportado en la Figura 3B indicó eficiencias de adsorción similares para las dos cepas. Creemos que esta discrepancia se debe a una fuerte reducción de la fluorescencia cuando la mRFP se une a PV361 pero no a PY79 o a las esporas de QM B1551 (véase más adelante).
Evaluación cuantitativa de la adsorción de la mRFP a las esporas de QM B1551
Los experimentos de dot blot (Figura 3B) indicaron que cuando se utilizaron 5 μg de mRFP purificada en reacciones de adsorción con 5,0 × 108 esporas de la cepa QM B1551 casi toda la proteína heteróloga se unió a la espora. Para definir la cantidad máxima de mRFP que puede adsorberse a las esporas de la cepa QM B1551, repetimos las reacciones con concentraciones crecientes de mRFP purificada, es decir, 5,0 × 108 esporas de la cepa QM B1551 se hicieron reaccionar con 5, 10, 20, 40, 80 y 160 μg de mRFP purificada. Tras las reacciones, las esporas se recogieron por centrifugación y los sobrenadantes que contenían la mRFP no unida se diluyeron en serie y se analizaron mediante dot blotting (Figura 5A). La Figura 5B muestra los resultados de un análisis densitométrico del dot blot (??), que indica que cuando 5-80 μg de mRFP se hicieron reaccionar con 5 × 108 esporas, el porcentaje de proteína unida a las esporas fue superior al 90%. Se observó una disminución de la mRFP unida cuando se utilizaron 160 μg de proteína purificada en la reacción. Sin embargo, incluso cuando se utilizaron 160 μg de mRFP purificada se absorbió más del 60% de la proteína, lo que indica que 5,0 × 108 esporas de QM B1551 pueden adsorber unos 100 μg de mRFP.
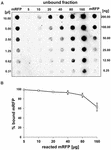
FIGURA 5. Evaluación cuantitativa de la adsorción de mRFP a esporas de B. megaterium QM B1551. Se incubaron 5 × 108 esporas con 5, 10, 20, 40, 80 y 160 μg de mRFP purificada. Las mezclas de reacción se sometieron posteriormente a centrifugación y los sobrenadantes se diluyeron en serie y se analizaron mediante dot blot (A). Se utilizaron diluciones seriadas de mRFP purificada como estándares. Las inmunorreacciones en ambos paneles se realizaron con un anticuerpo anti-His conjugado con peroxidasa de rábano picante. (B) Porcentaje de mRFP adsorbida a las esporas tras la reacción con cantidades definidas de mRFP endógena. Las barras de error muestran los errores estándar de la media de tres experimentos y el valor P nunca es superior a 0,0025.
La mRFP se localiza en el espacio intercapa de las esporas de B. megaterium QM B1551
Se empleó un enfoque de microscopía de inmunofluorescencia para evaluar si las moléculas de mRFP adsorbidas estaban expuestas en la superficie de las esporas de B. megaterium QM B1551. Las esporas QM B1551 adsorbidas con diversas cantidades de mRFP se hicieron reaccionar con un anticuerpo monoclonal anti-His que reconocía la mRFP recombinante, y luego con un anticuerpo secundario fluorescente anti-ratón (Santa Cruz Biotechnology, Inc.) y se observaron bajo el microscopio de fluorescencia (Figura 6). Con la menor cantidad de mRFP utilizada en este experimento (2 μg) la señal de fluorescencia de la mRFP (roja) se observó en todo el contorno de la espora, mientras que la señal de inmunofluorescencia (verde) fue débil y se concentró principalmente en los polos de la espora, lo que sugiere que sólo en esos puntos la mRFP estaba expuesta en la superficie de la espora. Al aumentar la cantidad de mRFP utilizada en la reacción, el número de puntos verdes aumentó (5 y 10 μg) y con la mayor cantidad de mRFP utilizada en la reacción (20 μg) se observó un anillo verde casi completo alrededor de las esporas. Sobre la base de los resultados presentados en la figura 6, formulamos la hipótesis de que las moléculas de mRFP se infiltran a través del exosporio y se localizan en el espacio entre la capa externa y el exosporio, es decir, cuando se utiliza una cantidad baja de mRFP casi todas las moléculas de proteína son internas al exosporio y están disponibles para el anticuerpo sólo en unos pocos lugares. El aumento de las cantidades de mRFP en las reacciones de adsorción hace que el espacio entre las capas se «llene», hasta que finalmente hay más moléculas de mRFP disponibles para el anticuerpo en la superficie de la espora. Esta hipótesis implica que si falta el exosporio, toda la mRFP debería estar disponible para el anticuerpo. Para comprobarlo, comparamos mediante microscopía de inmunofluorescencia números iguales de esporas de QM B1551 (con exosporio) y de PV361 (sin exosporio) incubadas con la misma cantidad de mRFP (5 μg). Cuando el exosporio estaba presente (QM B1551) la mRFP sólo estaba parcialmente disponible para el anticuerpo y se observaron manchas verdes (Figura 7 y Figura Suplementaria S2). Cuando el exosporio no estaba presente (PV361) la mRFP adsorbida estaba disponible para el anticuerpo alrededor de toda la espora y se formaba un anillo verde completo, apoyando la hipótesis de que la mRFP es interna al exosporio en las esporas de QM B1551.
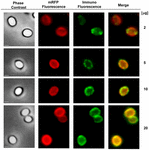
FIGURA 6. Análisis de inmunofluorescencia de esporas de B. megaterium QM B1551 adsorbidas por mRFP. Se incubaron alícuotas de 5 × 108 esporas de QM B1551 con concentraciones variables de mRFP y posteriormente se analizaron mediante microscopía de contraste de fase, fluorescencia e inmunofluorescencia, como se describe en los Materiales y Métodos. El mismo campo de microscopía para cada reacción se reporta junto con el panel de fusión. El tiempo de exposición fue de 200 ms para todas las imágenes. Barra de escala, 1 μm.
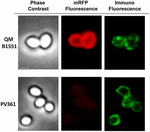
FIGURA 7. Inmunofluorescencia de mRFP adsorbida a esporas de B. megaterium QM B1551 y PV361. Se incubaron 5 × 108 esporas de QM B1551 y PV361 con 5 μg de mRFP y luego se analizaron mediante microscopía de inmunofluorescencia, como se describe en los Materiales y Métodos. Para cada campo se muestra el contraste de fase y la microscopía de inmunofluorescencia. El tiempo de exposición fue de 200 ms para todas las imágenes. Barra de escala, 1 μm.
Mientras que las esporas QM B1551 utilizadas en los experimentos de la Figura 7 mostraron un anillo fluorescente rojo completo como en la Figura 2, las esporas PV361 mostraron una señal fluorescente roja muy débil. Con las esporas PV361 sólo se observó una señal roja utilizando largos tiempos de exposición en el microscopio de fluorescencia (Figura 4). Dado que la mRFP está presente alrededor de las esporas PV361 (Figuras 3 y 7), concluimos que la fluorescencia de la mRFP se debilita cuando la proteína se adsorbe a las esporas PV361. Serán necesarios más experimentos para abordar plenamente este punto.
Discusión
Los principales hallazgos de este informe son que las esporas de B. megaterium son extremadamente eficientes en la adsorción de la proteína modelo heteróloga mRFP, que el exosporio tiene un papel importante en este proceso, y que las moléculas de mRFP se infiltran a través del exosporio localizándose entre la capa externa y el exosporio. Estos resultados amplían los trabajos anteriores realizados con esporas de B. subtilis y demuestran que las esporas de una especie diferente también pueden utilizarse para suministrar proteínas heterólogas mediante el método de adsorción. La alta eficiencia de adsorción observada con las esporas de B. megaterium se debe en parte al gran tamaño de su espora en comparación con la de B. subtilis. En efecto, la superficie de la espora de B. megaterium es aproximadamente 2,5 veces mayor que la de B. subtilis, con una superficie de 5,33 μm2 (h: 1,60 ± 0,16 w: 0,84 ± 0,07) frente a 1,98 μm2 (h: 1,07 ± 0,09 w: 0,48 ± 0,03). Las grandes dimensiones permiten la adsorción de hasta 100 μg de mRFP cuando 160 μg de proteína reaccionan con las esporas.
La observación de que la mRFP atraviesa el exosporio indica que es permeable a la mRFP, una proteína de 27 kDa. La permeabilidad del exosporio no es del todo sorprendente ya que los germinantes presentes en el medio ambiente tienen que atravesar las capas externas de la espora para alcanzar sus receptores, aunque los germinantes son típicamente moléculas pequeñas con masas moleculares típicamente <200 Da. Además, los datos de la mRFP coinciden ampliamente con los resultados de estudios anteriores realizados con dextranos marcados y moléculas relacionadas (Koshikawa et al., 1984; Nishihara et al., 1989). En esos estudios, se sugirió que el exosporio de B. megaterium QM B1551 representaba una barrera de permeabilidad para las moléculas con pesos moleculares superiores a 100 KDa, mientras que influía en el paso de moléculas con masas entre 2 y 40 kDa (Koshikawa et al, 1984; Nishihara et al., 1989).
Un reto interesante para futuros trabajos será establecer el mecanismo o ruta de infiltración que la mRFP, y por inferencia otras proteínas heterólogas de interés, toma para entrar en el espacio intercapa. El examen por microscopía electrónica de transmisión de esporas seccionadas de B. megaterium QM B1551 indica que el exosporio comprende dos «caparazones» idénticos (Manetsberger et al., 2015a), y puede ser que la interfaz entre cada una de estas estructuras (descritas como «aberturas apicales» en los primeros artículos) permita la entrada de moléculas relativamente grandes. También será interesante discernir la base de la aparente pérdida de fluorescencia de la mRFP tras la adsorción a las esporas de PV361, y si las moléculas de mRFP son capaces de infiltrarse en las capas de la capa externa, como se observó en las esporas de B. subtilis (Donadio et al., 2016).
En el estudio actual, planteamos la hipótesis de que las moléculas de mRFP atraviesan preferentemente el exosporio y se acumulan en el espacio inter-capa entre la capa externa y el exosporio. En este modelo, las moléculas de mRFP sólo se adsorben y se muestran en la superficie de la espora una vez que los sitios de adsorción (¿o la capacidad volumétrica?) en el espacio inter-capa están suficientemente ocupados. Esto implica que el enfoque de adsorción para la visualización en la superficie puede utilizarse con las esporas de B. megaterium QM B1551, aunque el sistema depende de la proporción espora-proteína utilizada en las reacciones de adsorción. Dado que varias cepas de B. megaterium se han utilizado durante mucho tiempo a nivel industrial para la producción de enzimas como amilasas y deshidrogenasas, vitaminas y moléculas antimicrobianas (Vary et al., 2007), nuestros datos sugieren una nueva aplicación biotecnológica para la espora de B. megaterium como vehículo para unir y entregar proteínas heterólogas.
Contribuciones de los autores
ML y GD – realizaron la mayor parte de los experimentos; RA y AS – contribuyeron a la interpretación de los datos del trabajo; GiC, LB, y GrC – contribuyeron a la redacción del trabajo y a su revisión crítica; ER – contribuyó a las discusiones y sugerencias durante el trabajo y contribuyó a la redacción del manuscrito; RI – dirigió el trabajo y contribuyó a la redacción del manuscrito. Todos los autores leyeron y aprobaron el manuscrito final.
Financiación
Este trabajo fue apoyado por una beca (STAR Linea1- 2014) de la Universidad Federico II de Nápoles y la Compagnia di San Paolo, Italia a RI.
Declaración de conflicto de intereses
Los autores declaran que la investigación se llevó a cabo en ausencia de cualquier relación comercial o financiera que pudiera interpretarse como un potencial conflicto de intereses.
Material suplementario
El material suplementario para este artículo se puede encontrar en línea en: https://www.frontiersin.org/article/10.3389/fmicb.2016.01752/full#supplementary-material
FIGURA S1 | Imágenes de campo completo del análisis de microscopía de fluorescencia informado en la Figura 4. El mismo campo de microscopía se observó por contraste de fase y microscopía de fluorescencia. Barra de escala de 5 μm. Se informa del panel de fusión. Se indica el tiempo de exposición.
FIGURA S2 | Imágenes de campo completo del análisis de inmunofluorescencia reportado en la Figura 7. El mismo campo de microscopía se observó por contraste de fase y microscopía de fluorescencia. Barra de escala de 5 μm. El tiempo de exposición fue de 200 ms para todas las imágenes.
TABLA S1 | Análisis densitométrico de los experimentos dot blot con los sobrenadantes de la reacción de adsorción con esporas QM B1551 (Figura 1B).
TABLA S2 | Análisis densitométrico de los experimentos de dot blot con los sobrenadantes de varios lavados después de la reacción de adsorción (Figura 1C).
TABLA S3 | Análisis densitométrico de los experimentos de dot blot con los sobrenadantes de la reacción de adsorción realizada con esporas de las cepas PY79, QM B1551 y PV361 (Figura 3B).
Cuadro S4 | Análisis densitométrico de los experimentos de dot blot con los sobrenadantes de la reacción de adsorción realizada con diferentes cantidades de mRFP.
Baccigalupi, L., Ricca, E., y Ghelardi, E. (2015). «Probióticos no LAB: formadores de esporas», en Probióticos y prebióticos: Current Research and Future Trends, eds K. Venema y A. P. do Carmo (Norfolk, VA: Caister Academic Press), 93-103. doi: 10.21775/9781910190098.06
CrossRef Full Text | Google Scholar
Campbell, R. E., Tour, O., Palmer, A. E., Steinbach, P. A., Baird, G. S., Zacharias, D. A., et al. (2002). A monomeric red fluorescent protein. Proc. Natl. Acad. Sci. U.S.A. 99, 7877-7882. doi: 10.1073/pnas.082243699
PubMed Abstract | CrossRef Full Text | Google Scholar
Cangiano, G., Sirec, T., Panarella, C., Isticato, R., Baccigalupi, L., De Felice, M., et al. (2014). Los productos del gen sps afectan a la germinación, la hidrofobicidad y la adsorción de proteínas de las esporas de Bacillus subtilis. Appl. Environ. Microbiol. 80, 7293-7302. doi: 10.1128/AEM.02893-14
PubMed Abstract | CrossRef Full Text | Google Scholar
Cutting, S. M. (2011). Bacillus probióticos. Food Microbiol. 28, 214-220. doi: 10.1016/j.fm.2010.03.007
PubMed Abstract | CrossRef Full Text | Google Scholar
Cutting, S., y Vander Horn, P. B. (1990). «Genetic analysis», en Molecular Biological Methods for Bacillus, eds C. Harwood y S. Cutting (Chichester: John Wiley and Sons), 27-74.
Google Scholar
Díaz-González, F., Milano, M., Olguin-Araneda, V., Pizarro-Cerda, J., Castro-Córdova, P., Tzeng, S. C., et al. (2015). Composición proteica de la capa más externa tipo exosporio de las esporas de Clostridium difficile 630. J. Proteom. 123, 1-13. doi: 10.1016/j.jprot.2015.03.035
PubMed Abstract | CrossRef Full Text | Google Scholar
Di Luccia, B., D’Apuzzo, E., Varriale, F., Baccigalupi, L., Ricca, E., y Pollice, A. (2016). Bacillus megaterium SF185 induce vías de estrés y afecta a la distribución del ciclo celular de las células epiteliales intestinales humanas. Benef. Microb. 7, 609-620. doi: 10.3920/BM2016.0020
PubMed Abstract | CrossRef Full Text | Google Scholar
Donadio, G., Lanzilli, M., Sirec, T., Ricca, E., and Isticato, R. (2016) Protein adsorption and localization on wild type and mutant spores of Bacillus subtilis. Microb. Cell Fact. 15:153. doi: 10.1186/s12934-016-0551-2
PubMed Abstract | CrossRef Full Text
Eppinger, M., Bunk, B., Johns, M. A., Edirisinghe, J. N., Kutumbaka, K. K., Koenig, S. S., et al. (2011). Genome sequences of the biotechnologically important Bacillus megaterium strains QM B1551 and DSM319. J. Bacteriol. 193, 4199-4213. doi: 10.1128/JB.00449-11
PubMed Abstract | CrossRef Full Text | Google Scholar
Giglio, R., Fani, R., Isticato, R., De Felice, M., Ricca, E., and Baccigalupi, L. (2011). Organización y evolución de los genes cotG y cotH de Bacillus subtilis. J. Bacteriol. 193, 6664-6673. doi: 10.1128/jb.06121-11
PubMed Abstract | CrossRef Full Text | Google Scholar
Harwood, C. R., y Cutting, S. M. (1990). Molecular Biological Methods for Bacillus. Chichester: John Wiley and Sons.
Google Scholar
Huang, J. M., Hong, H. A., Van Tong, H., Hoang, T. H., Brisson, A., y Cutting, S. M. (2010). Mucosal delivery of antigens using adsorption to bacterial spores. Vaccine 28, 1021-1030. doi: 10.1016/j.vaccine.2009.10.127
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., Pelosi, A., De Felice, M., and Ricca, E. (2010). CotE binds to CotC and CotU and mediates their interaction during spore coat formation in Bacillus subtilis. J. Bacteriol. 192, 949-954. doi: 10.1128/JB.01408-09
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., Pelosi, A., Zilhão, R., Baccigalupi, L., Henriques, A. O., De Felice, M., et al. (2008). CotC-CotU heterodimerization during assembly of the Bacillus subtilis spore coat. J. Bacteriol. 190, 1267-1275. doi: 10.1128/JB.01425-07
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., and Ricca, E. (2014). Visualización de la superficie de las esporas. Microbiol. Spectr. 2, 351-366. doi: 10.1128/microbiolspec.TBS-0011-2012
PubMed Abstract | CrossRef Full Text | Google Scholar
Isticato, R., Sirec, T., Treppiccione, L., Maurano, F., De Felice, M., Rossi, M., et al. (2013). Nonrecombinant display of the B subunit of the heat labile toxin of Escherichia coli on wild type and mutant spores of Bacillus subtilis. Microb. Cell Fact. 12:98. doi: 10.1186/1475-2859-12-98
PubMed Abstract | CrossRef Full Text | Google Scholar
Knecht, L. D., Pasini, P., y Daunert, S. (2011). Esporas bacterianas como plataformas para aplicaciones bioanalíticas y biomédicas. Anal. Bioanal. Chem. 400, 977-989. doi: 10.1007/s00216-011-4835-4
PubMed Abstract | CrossRef Full Text | Google Scholar
Koshikawa, T., Beaman, T. C., Pankratz, H. S., Nakashio, S., Corner, T. R., and Gerhardt, P. (1984). Resistencia, germinación y correlación de permeabilidad de las esporas de Bacillus megaterium despojadas sucesivamente de las capas de tegumento. J. Bacteriol. 159, 624-632.
PubMed Abstract | Google Scholar
Manetsberger, J., Hall, E. A. H., y Christie, G. (2015a). Los genes codificados por plásmidos influyen en el ensamblaje y la morfología del exosporio en las esporas de Bacillus megaterium QM B1551. FEMS Microbiol. Lett. 362:fnv147. doi: 10.1093/femsle/fnv147
PubMed Abstract | CrossRef Full Text | Google Scholar
Manetsberger, J., Manton, J. D., Erdelyi, M. J., Lin, H., Rees, D., Christie, G., et al. (2015b). La microscopía de localización elipsoidal infiere el tamaño y el orden de las capas de proteínas en las cubiertas de las esporas de Bacillus. Biophys. J. 109, 2058-2066. doi: 10.1016/j.bpj.2015.09.023
PubMed Abstract | CrossRef Full Text | Google Scholar
McCloy, R., Rogers, S., Caldon, E., Lorca, T., Castro, A., y Burgess, A. (2014). La inhibición parcial de Cdk1 en la fase G2 anula el SAC y desacopla los eventos mitóticos. Cell Cycle 13, 1400-1412. doi: 10.4161/cc.28401
PubMed Abstract | CrossRef Full Text | Google Scholar
McKenney, P. T., Driks, A., and Eichemberger, P. (2012). La endospora de Bacillus subtilis: ensamblaje y funciones de la capa multicapa. Nat. Rev. Microbiol. 11, 33-44. doi: 10.1038/nrmicro2921
PubMed Abstract | CrossRef Full Text | Google Scholar
Nicholson, W. L., y Setlow, P. (1990). «Sporulation, germination and out-growth,» en Molecular biological methods for Bacillus, eds C. Harwood and S. Cutting (Chichester: John Wiley and Sons), 391-450.
Google Scholar
Nishihara, T., Takubo, Y., Kawamata, E., Koshikawa, T., Ogaki, J., y Kondo, M. (1989). Role of outer coat in resistance of Bacillus megaterium spore. J. Biochem. 106, 270-273.
PubMed Abstract | Google Scholar
Pesce, G., Rusciano, G., Sirec, T., Isticato, R., Sasso, A., y Ricca, E. (2014). Mediciones de carga superficial y coeficiente hidrodinámico de la espora de Bacillus subtilis mediante pinzas ópticas. Colloids Surf. B Biointerfaces 116C, 568-575. doi: 10.1016/j.colsurfb.2014.01.039
PubMed Abstract | CrossRef Full Text | Google Scholar
Ricca, E., Baccigalupi, L., Cangiano, G., De Felice, M., and Isticato, R. (2014). Entrega de vacunas en la mucosa mediante esporas no recombinantes de Bacillus subtilis. Microb. Cell Fact. 13, 115. doi: 10.1186/s12934-014-0115-2
PubMed Abstract | CrossRef Full Text | Google Scholar
Rosso, M. L., y Vary, P. S. (2005). Distribución de los plásmidos de Bacillus megaterium QM B1551 entre otras cepas de B. megaterium y especies de Bacillus. Plasmid 53, 205-217. doi: 10.1016/j.plasmid.2004.10.005
PubMed Abstract | CrossRef Full Text | Google Scholar
Rusciano, G. Zito, G., Isticato, R., Sirec, T., Ricca, E., Bailo, E., y Sasso, A. (2014). Nanoscale chemical imaging of Bacillus subtilis spores by combining tip-enhanced Raman scattering and advanced statistical tools. ACS Nano 8, 12300-12309. doi: 10.1021/nn504595k
PubMed Abstract | CrossRef Full Text | Google Scholar
Sirec, T., Strazzulli, A., Isticato, R., De Felice, M., Moracci, M., and Ricca, E. (2012). Adsorción de beta-galactosidasa de Alicyclobacillus acidocaldarius en esporas de tipo salvaje y mutantes de Bacillus subtilis. Microb. Cell Fact. 11:100. doi: 10.1186/1475-2859-11-100
PubMed Abstract | CrossRef Full Text | Google Scholar
Stewart, G. C. (2015). La capa Exosporium de las esporas bacterianas: una conexión con el medio ambiente y el huésped infectado. Microbiol. Mol. Biol. Rev. 79, 437-457. doi: 10.1128/MMBR.00050-15
PubMed Abstract | CrossRef Full Text | Google Scholar
Vary, P. S., Biedendieck, R., Fuerch, T., Meinhardt, F., Rohde, M., Deckwer, W. -D., et al. (2007). Bacillus megaterium: de simple bacteria del suelo a huésped de producción industrial de proteínas. Appl. Microbiol. Biotechnol. 76, 957-967 doi: 10.1007/s00253-007-1089-3
PubMed Abstract | CrossRef Full Text | Google Scholar
Youngman, P., Perkins, J. B., y Losick, R. (1984). Un método novedoso para la clonación rápida en Escherichia coli del ADN cromosómico de Bacillus subtilis adyacente a la inserción Tn917. Mol. Gen. Genet. 195, 424-433. doi: 10.1007/BF00341443
CrossRef Full Text | Google Scholar